Content from RとRStudio入門
最終更新日:2024-11-22 | ページの編集
概要
質問
- RStudio内でどのように操作するのか?
- Rとの対話方法は?
- 環境をどのように管理するのか?
- パッケージをどのようにインストールするのか?
目的
- RStudioの各ペインの目的と使用方法を説明する
- RStudio内のボタンやオプションの場所を見つける
- 変数を定義する
- データを変数に代入する
- インタラクティブなRセッションでワークスペースを管理する
- 数学および比較演算子を使用する
- 関数を呼び出す
- パッケージを管理する
ワークショップを始める前に
ワークショップで使用する一部のパッケージが正しくインストールされない(または全くインストールされない)場合があるため、お使いのマシンにRとRStudioの最新バージョンがインストールされていることを確認してください。
なぜRとRStudioを使うのか?
Software CarpentryワークショップのRセクションへようこそ!
科学は多段階のプロセスです。 実験を設計してデータを収集した後、本当の楽しみは分析から始まります!このレッスンでは、R言語の基本を教えるとともに、科学プロジェクトのためのコードを整理するベストプラクティスを学び、作業をより簡単にする方法を紹介します。
データを分析するためにMicrosoft ExcelやGoogleスプレッドシートを使用することもできますが、これらのツールは柔軟性やアクセス性に限界があります。さらに、元データの変更や探索の手順を共有することが難しいため、これは「再現可能な」研究にとって重要なポイントです(再現可能な研究についてはこちら)。
したがって、このレッスンでは、RとRStudioを使用してデータの探索を始める方法を学びます。RプログラムはWindows、Mac、Linuxオペレーティングシステムで利用可能で、上記のリンクから無料でダウンロードできます。Rを実行するために必要なのはRプログラムだけです。
しかし、Rをより使いやすくするために、同じくダウンロードしたRStudioというプログラムを使用します。RStudioは無料でオープンソースの統合開発環境(IDE)で、組み込みエディタを提供し、すべてのプラットフォームで動作します(サーバー上でも利用可能)。バージョン管理やプロジェクト管理との統合など、多くの利点があります。
概要
生データから始め、探索的な分析を行い、結果をグラフでプロットする方法を学びます。この例では、gapminder.orgのデータセットを使用し、時間を通じた各国の人口情報を扱います。データをRに読み込むことができますか?セネガルの人口をプロットできますか?アジア大陸の国々の平均所得を計算できますか?このレッスンの終わりまでに、これらの国々の人口を1分以内にプロットできるようになります!
基本レイアウト
RStudioを初めて開くと、次の3つのパネルが表示されます:
- インタラクティブなRコンソール/ターミナル(左全体)
- 環境/履歴/接続(右上のタブ)
- ファイル/プロット/パッケージ/ヘルプ/ビューア(右下のタブ)
ファイル(例えばRスクリプト)を開くと、上部左にエディタパネルも表示されます。
Rスクリプト
Rコンソールに書いたコマンドはファイルに保存し、再実行することができます。このようなRコードを含むファイルをRスクリプトと呼びます。Rスクリプトは名前の末尾が.Rとなっており、それがRスクリプトであることを示します。
RStudio内でのワークフロー
RStudio内で作業する主な方法は2つあります:
- インタラクティブなRコンソール内でテストや試行を行い、そのコードをコピーして.Rファイルに貼り付け、後で実行する。
- 小規模なテストや初期の段階では効果的です。
- しかし、すぐに手間がかかるようになります。
- 最初から.Rファイルに記述し、RStudioのショートカットキーを使用して「Run」コマンドを実行し、現在の行、選択した行、または変更した行をインタラクティブなRコンソールに送る。
- これは作業を始める良い方法です。すべてのコードが後で使用するために保存されます。
- RStudio内またはRの
source()関数を使用して作成したファイルを実行できます。
ヒント:コードのセグメントを実行する
RStudioでは、エディタウィンドウからコードを実行する柔軟性があります。ボタン、メニューオプション、およびキーボードショートカットがあります。現在の行を実行するには、次の方法があります:
- エディタパネルの上部にある「Run」ボタンをクリックする
- 「Code」メニューから「Run Lines」を選択する
- WindowsまたはLinuxではCtrl+Return、OS Xでは⌘+Returnを押す (このショートカットはボタンの上にマウスをホバーさせると確認できます)。コードブロックを実行するには、選択して「Run」をクリックします。 最近実行したコードブロックを修正した場合、セクションを再選択して「Run」を押す必要はありません。次のボタン「Re-run the previous region」を使用すると、修正を含む前のコードブロックを実行できます。
R入門
Rでの作業の多くは、インタラクティブなRコンソール内で行います。ここでは、すべてのコードを実行し、Rスクリプトファイルに追加する前にアイデアを試すのに便利な環境です。RStudioのコンソールは、コマンドライン環境でRと入力した場合と同じです。
Rのインタラクティブセッションを開くと、最初に情報が表示され、その後に「>」と点滅するカーソルが現れます。これは、シェルレッスンで学んだシェル環境と多くの点で似ています。「Read, evaluate, print loop」(読み取り、評価、印刷ループ)の考え方に基づいて動作します:コマンドを入力すると、Rがそれを実行し、結果を返します。
Rを計算機として使う
Rで最も簡単なことは、算術を行うことです:
R
1 + 100
出力
[1] 101Rは答えを表示し、その前に”[1]“を付けます。[1]はコンソールに表示される行の最初の要素のインデックスを示します。ベクトルのインデックスについての詳細は、エピソード6:データのサブセット化を参照してください。
不完全なコマンドを入力すると、R
は完了を待機します。Unix Shellのbashに慣れている場合、この動作をbashで見たことがあるかもしれません。
出力
+「>」ではなく「+」が表示された場合、Rはコマンドの完了を待機しています。コマンドをキャンセルしたい場合はEscを押すと、RStudioは「>」プロンプトに戻ります。
ヒント:コマンドのキャンセル
コマンドラインからRを使用している場合は、RStudioの代わりにEscではなくCtrl+Cを使用してコマンドをキャンセルする必要があります。これはMacユーザーにも適用されます!
コマンドのキャンセルは、不完全なコマンドを終了させるだけでなく、予想以上に時間がかかる場合にコードの実行を停止したり、現在書いているコードを削除したりするためにも役立ちます。
Rを計算機として使用する場合、演算の順序は学校で学んだものと同じです。
優先順位が高いものから低いものへ:
- 括弧:
(,) - 累乗:
^または** - 乗算:
* - 除算:
/ - 加算:
+ - 減算:
-
R
3 + 5 * 2
出力
[1] 13評価の順序を変更したい場合や意図を明確にしたい場合は、括弧を使用してグループ化します。
R
(3 + 5) * 2
出力
[1] 16必要ない場合は煩雑になりますが、意図を明確にできます。他の人が後でコードを読むかもしれないことを忘れないでください。
R
(3 + (5 * (2 ^ 2))) # 読みにくい
3 + 5 * 2 ^ 2 # 規則を覚えていれば明快
3 + 5 * (2 ^ 2) # 規則を忘れた場合はこれが助けになる
各コード行の後にあるテキストは「コメント」と呼ばれます。ハッシュ記号#の後に続く内容は、コードを実行する際にRによって無視されます。
非常に小さいまたは大きい数値は、科学表記法で表示されます:
R
2/10000
出力
[1] 2e-04これは「10^XXで掛ける」という短縮形です。したがって、2e-4は2 * 10^(-4)の短縮形です。
科学表記法で数値を書くこともできます:
R
5e3 # マイナスがない点に注意
出力
[1] 5000数学関数
Rには多くの組み込み数学関数があります。関数を呼び出すには、関数名を入力し、その後に開き括弧と閉じ括弧を続けます。関数は引数を入力として受け取ります。関数の括弧内に入力したものはすべて引数と見なされます。関数によって引数の数は異なり、引数を必要としないものから複数の引数を必要とするものまであります。例:
R
getwd() # 絶対パスを返す
この例では引数は不要ですが、以下の数学関数では結果を計算するために値を渡す必要があります。
R
sin(1) # 三角関数
出力
[1] 0.841471R
log(1) # 自然対数
出力
[1] 0R
log10(10) # 常用対数(底10)
出力
[1] 1R
exp(0.5) # e^(1/2)
出力
[1] 1.648721Rのすべての関数を覚えようとする必要はありません。Googleで検索するか、関数名の最初の数文字を覚えていれば、RStudioのタブ補完機能を使うことができます。
RStudioの大きな利点の一つは、オートコンプリート機能があることです。これにより、関数、引数、および受け取る値を簡単に調べることができます。
コマンド名の前に?を付けると、そのコマンドのヘルプページが開きます。RStudioを使用している場合、’Help’ペインに表示されます。ターミナルでRを使用している場合は、ブラウザでヘルプページが開きます。ヘルプページにはコマンドの詳細な説明と動作の仕組みが含まれています。ページの下部までスクロールすると、通常、コマンドの使用例が掲載されています。後ほど例を見ていきます。
比較演算
Rでは比較を行うこともできます:
R
1 == 1 # 等しい(等号が2つ、"等しい"と読む)
出力
[1] TRUER
1 != 2 # 等しくない("等しくない"と読む)
出力
[1] TRUER
1 < 2 # より小さい
出力
[1] TRUER
1 <= 1 # 以下
出力
[1] TRUER
1 > 0 # より大きい
出力
[1] TRUER
1 >= -9 # 以上
出力
[1] TRUEヒント:数値の比較
数値を比較する際の注意点として、整数(小数を含まない数値型)以外を比較する場合は、==を使用しないでください。
コンピュータは小数を特定の精度でしか表現できないため、Rが表示する際に同じに見える2つの数値が、内部表現では異なる場合があります。このわずかな差異は「数値計算誤差(Machine numeric tolerance)」と呼ばれます。
代わりにall.equal関数を使用してください。
さらに詳しく知りたい方はこちら:http://floating-point-gui.de/
変数と代入
代入演算子<-を使用して、値を変数に格納できます:
R
x <- 1/40
代入は値を表示しません。代わりに、それを後で使用するために変数というものに格納します。この場合、xには値0.025が格納されています:
R
x
出力
[1] 0.025正確には、この格納された値は浮動小数点数と呼ばれる分数の10進数近似値です。
RStudioの右上ペインにあるEnvironmentタブを確認すると、xとその値が表示されていることがわかります。変数xは、数値を期待する計算の中で数値の代わりに使用できます:
R
log(x)
出力
[1] -3.688879また、変数には再代入も可能です:
R
x <- 100
以前はxに0.025が格納されていましたが、現在は100が格納されています。
代入値には、代入先の変数を含めることもできます:
R
x <- x + 1 # RStudioの右上タブでxの説明が更新されることに注目
y <- x * 2
代入の右辺には有効なR式を使用できます。右辺は代入が行われる前に完全に評価されます。
変数名には、文字、数字、アンダースコア、ピリオドを含めることができますが、スペースは含められません。また、変数名は文字またはピリオドで始める必要があります(数字やアンダースコアでは始めることはできません)。ピリオドで始まる変数は隠し変数と見なされます。
長い変数名については、異なる人が異なる規約を使用します。その例として:
- periods.between.words
- underscores_between_words
- camelCaseToSeparateWords
どれを使用するかは自由ですが、一貫性を保つことが重要です。
代入には=演算子を使用することも可能です:
R
x = 1/40
しかし、これはRユーザーの間ではあまり一般的ではありません。最も重要なのは、使用する演算子に一貫性を持つことです。<-を使用したほうが混乱が少ない場合もあり、コミュニティでは最も一般的に使われています。そのため、<-を使用することを推奨します。
ベクトル化
Rの特徴の1つに、Rがベクトル化されているという点があります。つまり、変数や関数にベクトルを値として持たせることができます。物理学や数学におけるベクトルとは異なり、Rにおけるベクトルは同じデータ型の値が順序付けられた集合を指します。例:
R
1:5
出力
[1] 1 2 3 4 5R
2^(1:5)
出力
[1] 2 4 8 16 32R
x <- 1:5
2^x
出力
[1] 2 4 8 16 32この機能は非常に強力で、今後のレッスンでさらに詳しく説明します。
環境の管理
Rセッションとやり取りするための便利なコマンドがいくつかあります。
lsを使用すると、グローバル環境(現在のRセッション)に保存されているすべての変数と関数を一覧表示できます:
R
ls()
出力
[1] "x" "y"ヒント:隠しオブジェクト
シェルと同様に、lsではデフォルトで”.”で始まる変数や関数は表示されません。すべてのオブジェクトを一覧表示するには、ls(all.names=TRUE)と入力してください。
ここでは、lsに引数を渡していませんが、関数を呼び出すために括弧は必要です。
lsだけを入力すると、オブジェクト一覧ではなくコードが表示されます:
R
ls
出力
function (name, pos = -1L, envir = as.environment(pos), all.names = FALSE,
pattern, sorted = TRUE)
{
if (!missing(name)) {
pos <- tryCatch(name, error = function(e) e)
if (inherits(pos, "error")) {
name <- substitute(name)
if (!is.character(name))
name <- deparse(name)
warning(gettextf("%s converted to character string",
sQuote(name)), domain = NA)
pos <- name
}
}
all.names <- .Internal(ls(envir, all.names, sorted))
if (!missing(pattern)) {
if ((ll <- length(grep("[", pattern, fixed = TRUE))) &&
ll != length(grep("]", pattern, fixed = TRUE))) {
if (pattern == "[") {
pattern <- "\\["
warning("replaced regular expression pattern '[' by '\\\\['")
}
else if (length(grep("[^\\\\]\\[<-", pattern))) {
pattern <- sub("\\[<-", "\\\\\\[<-", pattern)
warning("replaced '[<-' by '\\\\[<-' in regular expression pattern")
}
}
grep(pattern, all.names, value = TRUE)
}
else all.names
}
<bytecode: 0x55c229e87d60>
<environment: namespace:base>これはどういうことでしょうか?
Rではすべてがオブジェクトであり、オブジェクト名だけを入力すると、その内容が表示されます。先ほど作成したオブジェクトxには1,
2, 3, 4, 5が格納されています:
R
x
出力
[1] 1 2 3 4 5オブジェクトlsには、ls関数を動作させるRコードが格納されています!関数の仕組みや作成方法については後のレッスンで説明します。
不要になったオブジェクトを削除するには、rmを使用します:
R
rm(x)
多くのオブジェクトが環境にあり、それらをすべて削除したい場合は、lsの結果をrm関数に渡します:
R
rm(list = ls())
この場合、2つの関数を組み合わせています。演算の順序と同様に、最も内側の括弧内の内容が最初に評価されます。
この場合、lsの結果がrmのlist引数として使用されるよう指定しています。引数に値を名前で割り当てる場合、必ず=演算子を使用する必要があります!
代わりに<-を使用すると、予期しない副作用が発生するか、エラーメッセージが表示される可能性があります:
R
rm(list <- ls())
エラー
Error in rm(list <- ls()): ... must contain names or character stringsヒント:警告とエラー
Rが予期しない動作をした場合は注意してください!エラーはRが計算を続行できない場合に発生します。一方、警告は通常、関数が実行されたものの、期待通りに動作しなかったことを意味します。
どちらの場合も、Rが表示するメッセージには問題を解決するための手がかりが含まれていることが多いです。
Rパッケージ
Rにはパッケージを作成することで関数を追加することができます。また、他の人が作成したパッケージを利用することも可能です。この執筆時点で、CRAN(Comprehensive R Archive Network)には10,000を超えるパッケージが利用可能です。RとRStudioにはパッケージを管理するための機能があります:
- インストールされているパッケージを確認するには、
installed.packages()を入力します。 - パッケージをインストールするには、
install.packages("packagename")と入力します。ここでpackagenameはパッケージ名で、引用符で囲みます。 - インストール済みのパッケージを更新するには、
update.packages()を入力します。 - パッケージを削除するには、
remove.packages("packagename")を使用します。 - パッケージを利用可能にするには、
library(packagename)を入力します。
RStudioの右下ペインの「Packages」タブからもパッケージを表示、ロード、デタッチすることができます。このタブをクリックすると、インストール済みのパッケージがチェックボックス付きで表示されます。パッケージ名の横にあるチェックボックスがオンの場合、そのパッケージはロードされており、オフの場合はロードされていません。空のボックスをクリックするとそのパッケージがロードされ、チェックボックスをクリックするとパッケージがデタッチされます。
また、「Packages」タブの上部にある「Install」ボタンと「Update」ボタンを使用して、パッケージをインストールおよび更新できます。
チャレンジ 2
次のプログラムの各文の後で、各変数の値はどうなるでしょうか?
R
mass <- 47.5
age <- 122
mass <- mass * 2.3
age <- age - 20
R
mass <- 47.5
この時点で変数massの値は47.5になります。
R
age <- 122
この時点で変数ageの値は122になります。
R
mass <- mass * 2.3
既存の値47.5に2.3を掛け、新しい値109.25をmassに格納します。
R
age <- age - 20
既存の値122から20を引き、新しい値102をageに格納します。
チャレンジ 3
前のチャレンジのコードを実行し、massとageを比較するコマンドを書きなさい。massはageより大きいですか?
この質問に答える方法の1つとして、次のように>を使用できます:
R
mass > age
出力
[1] TRUEこのコードは、109.25が102より大きいため、論理値TRUEを返すはずです。
チャレンジ 4
作業環境を整理し、massとageの変数を削除しなさい。
このタスクを達成するには、rmコマンドを使用します:
R
rm(age, mass)
チャレンジ 5
以下のパッケージをインストールしなさい:ggplot2,
plyr, gapminder
必要なパッケージをインストールするには、install.packages()コマンドを使用します。
R
install.packages("ggplot2")
install.packages("plyr")
install.packages("gapminder")
1つのinstall.packages()コマンドで複数のパッケージ
をインストールする別の方法は次のとおりです:
R
install.packages(c("ggplot2", "plyr", "gapminder"))
まとめ
- RStudioを使用してRプログラムを作成および実行します。
- Rには通常の算術演算子と数学関数があります。
-
<-を使用して変数に値を代入します。 -
ls()を使用してプログラム内の変数を一覧表示します。 -
rm()を使用してプログラム内のオブジェクトを削除します。 -
install.packages()を使用してパッケージ(ライブラリ)をインストールします。
Content from RStudio を使ったプロジェクト管理
最終更新日:2024-11-22 | ページの編集
概要
質問
- R でプロジェクトをどのように管理できますか?
目的
- RStudio で自己完結型のプロジェクトを作成する
はじめに
科学的なプロセスは本質的に段階的なものであり、多くのプロジェクトはランダムなメモ、一部のコード、次に原稿と進行し、最終的にはすべてが混ざり合ってしまうことがよくあります。
プロジェクトを再現可能な方法で管理することは、科学を再現可能にするだけでなく、生活をより簡単にします。
— Vince Buffalo (@vsbuffalo) 2013年4月15日
ほとんどの人はプロジェクトを次のように整理しがちです:
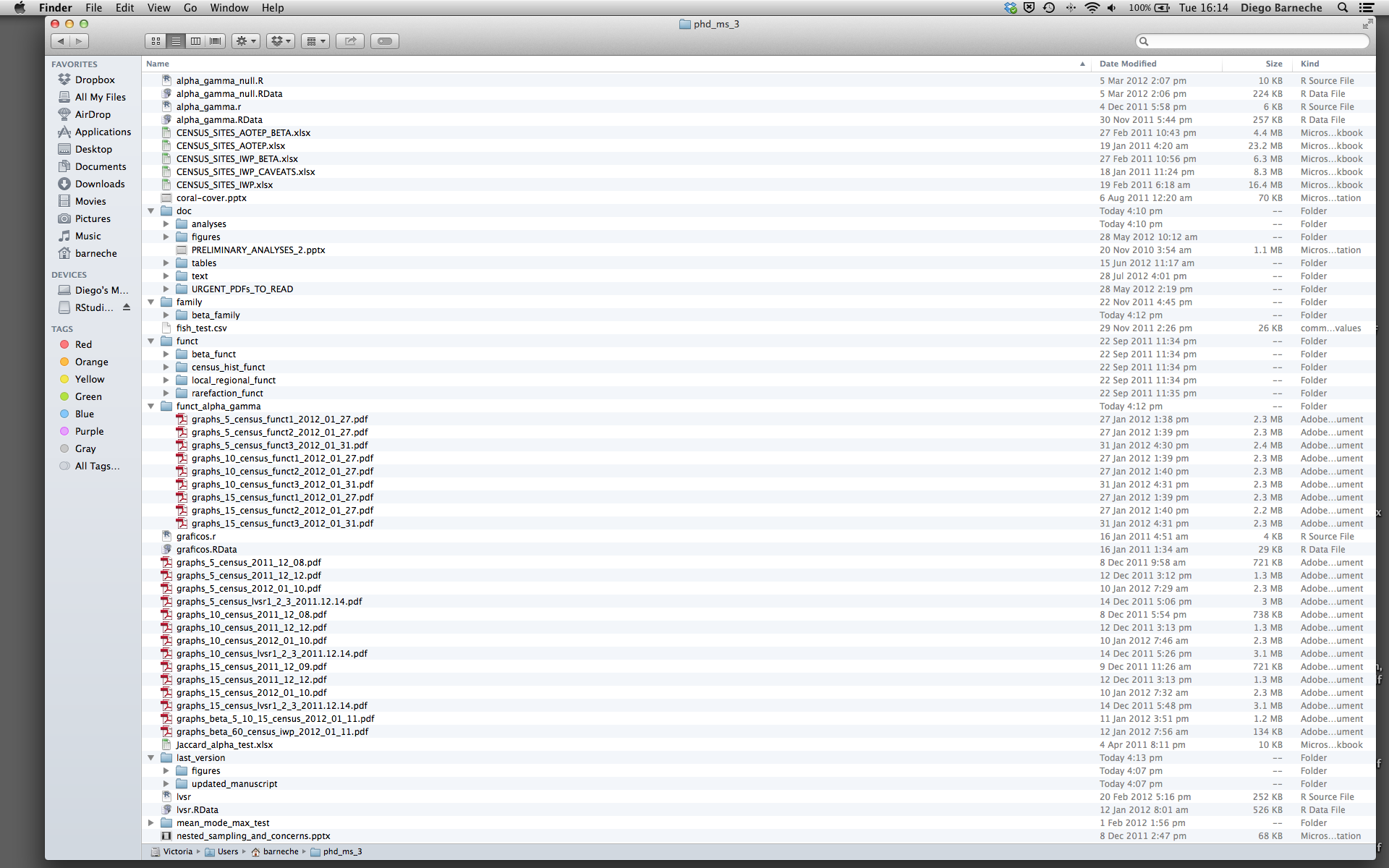
このような方法を絶対に避けるべき理由は数多くあります:
- データのどのバージョンがオリジナルで、どれが修正済みなのかを区別するのが非常に難しい。
- 様々な拡張子のファイルが混在して、非常に散らかる。
- 必要なものを見つけたり、正確なコードで生成した正しい図表を関連付けたりするのに非常に時間がかかる。
良いプロジェクト構成は、最終的に生活をより簡単にします:
- データの整合性を確保しやすくなる。
- 他の人(研究室の同僚、共同研究者、指導教員)とコードを共有するのが簡単になる。
- 原稿の投稿時にコードを簡単にアップロードできる。
- しばらく休んだ後にプロジェクトを再開しやすくなる。
考えられる解決策
幸いなことに、作業を効果的に管理するためのツールやパッケージが存在します。
RStudio の最も強力で便利な機能の一つがプロジェクト管理機能です。本日はこれを使って自己完結型の再現可能なプロジェクトを作成します。
チャレンジ 1: 自己完結型プロジェクトの作成
RStudio で新しいプロジェクトを作成します:
- 「File」メニューをクリックし、「New Project」を選択します。
- 「New Directory」をクリックします。
- 「New Project」をクリックします。
- プロジェクトを保存するディレクトリの名前(例:
my_project)を入力します。 - 「Create a git repository」のチェックボックスが表示される場合は選択します。
- 「Create Project」ボタンをクリックします。
作成した RStudio
プロジェクトを開く最も簡単な方法は、ファイルシステムをたどって保存したディレクトリに移動し、.Rproj
ファイルをダブルクリックすることです。これにより RStudio が開き、R
セッションが .Rproj
ファイルと同じディレクトリで開始します。データ、プロット、スクリプトはすべてプロジェクトディレクトリに関連付けられます。さらに、RStudio
プロジェクトは複数のプロジェクトを同時に開くことが可能で、それぞれのプロジェクトディレクトリに分離されます。これにより、複数のプロジェクトを開いても相互に干渉しません。
チャレンジ 2: ファイルシステムを使った RStudio プロジェクトの開き方
- RStudio を終了します。
- チャレンジ 1 で作成したプロジェクトのディレクトリに移動します。
- そのディレクトリ内の
.Rprojファイルをダブルクリックします。
プロジェクト管理のベストプラクティス
プロジェクトを整理するための「ベスト」な方法はありませんが、管理を容易にするために従うべきいくつかの一般原則があります:
データを読み取り専用として扱う
プロジェクトを設定する際の最も重要な目標はこれです。データの収集は通常、時間と費用がかかります。データをインタラクティブに操作する(例:Excel で)と、データの出所や収集後にどのように変更されたかを把握できなくなります。そのため、データを「読み取り専用」として扱うのが良い考えです。
データのクリーニング
多くの場合、データは「汚れて」おり、R(または他のプログラミング言語)で有用な形式にするために大幅な前処理が必要です。このタスクは「データマンジング」と呼ばれることもあります。これらのスクリプトを別のフォルダに保存し、クリーンなデータセットを保持する「読み取り専用」データフォルダを作成することで、両者の混同を防ぐことができます。
生成された出力を使い捨てとみなす
スクリプトによって生成されたものはすべて使い捨てとみなすべきです:スクリプトからすべてを再生成できる必要があります。
出力を管理する方法はたくさんあります。各分析ごとに異なるサブディレクトリを持つ出力フォルダを用意すると、後で便利です。多くの分析は探索的で最終プロジェクトに使用されないことが多く、一部の分析はプロジェクト間で共有されることもあります。
ヒント: 科学的コンピューティングのための「十分に良い」実践
科学的コンピューティングのための「十分に良い」実践 では、プロジェクトの構成について以下の推奨事項を挙げています:
- 各プロジェクトを専用のディレクトリに配置し、そのディレクトリにプロジェクト名を付ける。
- プロジェクトに関連するテキスト文書を
docディレクトリに配置する。 - 生データとメタデータを
dataディレクトリに、クリーンアップや分析中に生成されたファイルをresultsディレクトリに配置する。 - プロジェクトのスクリプトやプログラムのソースを
srcディレクトリに配置し、他から持ち込んだプログラムやローカルでコンパイルしたプログラムをbinディレクトリに配置する。 - すべてのファイルに内容や機能を反映した名前を付ける。
関数の定義と適用を分離する
R を効率的に使用する最も効果的な方法の一つは、最初に .R スクリプトに実行したいコードを書き、RStudio のキーボードショートカットを使用するか「Run」ボタンをクリックして、選択した行をインタラクティブな R コンソールで実行することです。
プロジェクトの初期段階では、最初の .R スクリプトファイルに多くの直接実行されるコード行が含まれることがよくあります。プロジェクトが進むにつれて、再利用可能なコードチャンクが独自の関数に分離されます。これらの関数を保存するためのフォルダと分析スクリプトを保存するためのフォルダを分けるのが良いアイデアです。
データを data ディレクトリに保存する
良好なディレクト
リ構造ができたら、データファイルを data/
ディレクトリに保存します。
チャレンジ 3
このリンクから CSV ファイルをダウンロードしてください。
- ファイルをダウンロードします(上記リンクを右クリック -> 「リンク先を名前を付けて保存」/「名前を付けて保存」、またはリンクをクリックしページが読み込まれた後に Ctrl+S を押すか、メニューの「ファイル」 -> 「ページを名前を付けて保存」を選択)。
-
gapminder_data.csvという名前で保存されていることを確認します。 - ファイルをプロジェクト内の
data/フォルダに保存します。
後ほどこのデータを読み込み、確認します。
チャレンジ 4
R に読み込む前に、コマンドラインからデータセットについての一般的な情報を得ることは有益です。これにより、R に読み込む際の判断に役立ちます。コマンドラインシェルを使用して以下の質問に答えてください:
- ファイルのサイズはどれくらいですか?
- このファイルには何行のデータがありますか?
- このファイルにはどのような値が含まれていますか?
次のコマンドをシェルで実行します:
出力
-rw-r--r-- 1 runner docker 80K Nov 22 08:04 data/gapminder_data.csvファイルサイズは 80K です。
出力
1705 data/gapminder_data.csv行数は 1705 行です。データの内容は次のようになります:
出力
country,year,pop,continent,lifeExp,gdpPercap
Afghanistan,1952,8425333,Asia,28.801,779.4453145
Afghanistan,1957,9240934,Asia,30.332,820.8530296
Afghanistan,1962,10267083,Asia,31.997,853.10071
Afghanistan,1967,11537966,Asia,34.02,836.1971382
Afghanistan,1972,13079460,Asia,36.088,739.9811058
Afghanistan,1977,14880372,Asia,38.438,786.11336
Afghanistan,1982,12881816,Asia,39.854,978.0114388
Afghanistan,1987,13867957,Asia,40.822,852.3959448
Afghanistan,1992,16317921,Asia,41.674,649.3413952ヒント: RStudio のコマンドライン
RStudio のコンソールペインにある「Terminal」タブを使用すると、RStudio 内で直接コマンドラインを操作できます。
作業ディレクトリ
R の現在の作業ディレクトリを知ることは重要です。なぜなら、他のファイルにアクセスする必要があるとき(例:データファイルをインポートする場合)、R は現在の作業ディレクトリを基準にそれらのファイルを探すからです。
新しい RStudio
プロジェクトを作成するたびに、そのプロジェクトの新しいディレクトリが作成されます。既存の
.Rproj ファイルを開くと、そのプロジェクトが開き、R
の作業ディレクトリがそのファイルがあるフォルダに設定されます。
チャレンジ 5
getwd() コマンドを使用するか、RStudio
のメニューを使って現在の作業ディレクトリを確認します。
- コンソールで
getwd()(“wd” は “working directory” の略)と入力し、Enter を押します。 - ファイルペインで、
dataフォルダをダブルクリックして開く(または他の任意のフォルダに移動)。作業ディレクトリに戻るには、ファイルペインの「More」をクリックし、「Go To Working Directory」を選択します。
setwd() コマンドを使用するか、RStudio
のメニューを使って作業ディレクトリを変更します。
- コンソールで
setwd("data")と入力し、Enter を押します。その後、getwd()と入力して Enter を押し、新しい作業ディレクトリを確認します。 - RStudio ウィンドウ上部のメニューで「Session」をクリックし、「Set
Working Directory」を選択して「Choose
Directory」をクリックします。その後、開いたウィンドウでプロジェクトディレクトリに戻り、「Open」をクリックします。コンソールに
setwdコマンドが自動的に表示されます。
ヒント: ファイルが存在しないエラー
R コードでファイルを参照しようとして「ファイルが存在しない」というエラーが出た場合は、作業ディレクトリを確認するのが良いです。 ファイルへの絶対パスを指定するか、作業ディレクトリ内(またはそのサブフォルダ)にファイルを保存し、相対パスを指定する必要があります。
バージョン管理
プロジェクトではバージョン管理を使用することが重要です。RStudio で Git を使用する方法についての良いレッスンはこちらを参照してください。
まとめ
- RStudio を使用して一貫したレイアウトでプロジェクトを作成および管理する。
- 生データを読み取り専用として扱う。
- 生成された出力を使い捨てとみなす。
- 関数の定義と適用を分離する。
Content from ヘルプの利用
最終更新日:2024-11-22 | ページの編集
概要
質問
- R でどのようにしてヘルプを得ることができますか?
目的
- 関数や特殊な演算子に関する R のヘルプファイルを読むことができる。
- 問題を解決するためのパッケージを特定するために CRAN タスクビューを利用できる。
- 仲間に助けを求める方法を理解する。
ヘルプファイルを読む
R および各パッケージには関数用のヘルプファイルが用意されています。特定のパッケージに含まれる関数についてヘルプを検索する際の一般的な構文は次の通りです:
R
?function_name
help(function_name)
たとえば、write.table()
のヘルプファイルを見てみましょう。この関数に似た機能を持つ関数を今後のエピソードで使用します。
R
?write.table()
これにより、RStudio ではヘルプページが表示され、R 本体ではプレーンテキストとして表示されます。
各ヘルプページは次のようなセクションに分かれています:
- Description(説明): 関数が何をするかの詳細な説明。
- Usage(使用法): 関数の引数とそのデフォルト値(変更可能)。
- Arguments(引数): 各引数が期待するデータの説明。
- Details(詳細): 注意すべき重要な点。
- Value(戻り値): 関数が返すデータ。
- See Also(関連項目): 有用な関連関数。
- Examples(例): 関数の使用例。
関数によってはセクションが異なる場合がありますが、これらが主なポイントです。
関連する関数が同じヘルプファイルを参照する場合があることに注意してください:
R
?write.table()
?write.csv()
これらの関数は非常に似た用途を持ち、引数も共通しているため、パッケージ作者が同じヘルプファイルで文書化していることがよくあります。
ヒント: 実例を実行する
ヘルプページ内の Examples セクションからコードをハイライトして Ctrl+Return を押すと、RStudio コンソールで実行されます。 関数の動作を素早く理解する方法です。
ヒント: ヘルプファイルを読む
R の大きな課題の一つは、利用可能な関数の数が膨大であることです。 すべての関数の正しい使用法を記憶するのは現実的ではありません。 しかし、ヘルプファイルを利用すれば、記憶する必要はありません!
特殊な演算子
特殊な演算子に関するヘルプを検索するには、引用符またはバッククォートを使用します:
R
?"<-"
?`<-`
パッケージに関するヘルプ
多くのパッケージには「ビネット」と呼ばれるチュートリアルや拡張的な例のドキュメントが含まれています。
引数なしで vignette()
を実行すると、インストール済みのすべてのパッケージのビネットが表示されます。
特定のパッケージについては vignette(package="パッケージ名")
を使用します。 特定のビネットを開くには
vignette("ビネット名") を実行します。
パッケージにビネットがない場合は、通常、次のコマンドでヘルプを探せます:
R
help("パッケージ名")
また、RStudio には多くのパッケージ向けに優れた チートシート があります。
関数名の一部を覚えている場合
関数がどのパッケージに属しているか、または正確なスペルがわからない場合、ファジー検索が可能です:
R
??function_name
ファジー検索では文字列の近似一致を検索します。たとえば、作業ディレクトリを設定する関数に「set」が含まれていることを覚えている場合、次のように検索できます:
R
??set
どこから始めるべきかわからない場合
どの関数やパッケージを使用すべきかわからない場合、 CRAN Task Views を利用するとよいでしょう。 これは、パッケージを分野別にグループ化した特別なリストで、出発点として適しています。
コードが動作しない場合: 仲間に助けを求める
関数の使用に問題がある場合、その答えのほとんどはすでに Stack Overflow
で回答されています。 [r]
タグを使って検索してください。質問の仕方については、Stack Overflow の 良い質問の仕方のページを参照してください。
答えが見つからない場合、以下の便利な関数を使って仲間に助けを求めるとよいでしょう:
R
?dput
この関数は、使用しているデータを他の人が自分の R セッションでコピー&ペーストできる形式に出力します。
R
sessionInfo()
出力
R version 4.4.2 (2024-10-31)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.5 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.10.0
LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.10.0
locale:
[1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
[4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
[7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
[10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
time zone: UTC
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
loaded via a namespace (and not attached):
[1] compiler_4.4.2 tools_4.4.2 yaml_2.3.10 knitr_1.48 xfun_0.49
[6] renv_1.0.11 evaluate_1.0.1この関数は、R の現在のバージョンやロードしているパッケージを表示します。 これは他の人が問題を再現し、デバッグするのに役立ちます。
チャレンジ 1
c
関数のヘルプページを見てください。次のコードを評価した場合、どのようなベクトルが作成されると思いますか?
R
c(1, 2, 3)
c('d', 'e', 'f')
c(1, 2, 'f')
c()
関数はすべての要素が同じ型のベクトルを作成します。最初の場合、要素は数値型、
2 番目の場合は文字型、そして 3
番目の場合も文字型です。数値型の値は文字型に「強制変換」されます。
チャレンジ 2
paste
関数のヘルプを見てください。この関数を後ほど使用します。
sep 引数と collapse 引数の違いは何ですか?
paste() 関数のヘルプを見るには以下を実行します:
R
help("paste")
?paste
sep と collapse
の違いは少し複雑です。paste
関数は任意の数の引数を受け取り、それぞれが任意の長さのベクトルであることができます。
sep
引数は連結される各項目の間に使用される文字列を指定します(デフォルトはスペース)。
結果は、paste
に渡された最も長い引数と同じ長さのベクトルです。
一方、collapse
引数は、連結後の要素を指定された区切り文字を使用して「まとめて結合」することを示します。
その結果、単一の文字列になります。
引数を明示的に指定することが重要です。たとえば sep = ","
と入力すると、関数は区切り文字として “,”
を使用し、結合する項目としてではないと認識します。
例:
R
paste(c("a","b"), "c")
出力
[1] "a c" "b c"R
paste(c("a","b"), "c", ",")
出力
[1] "a c ," "b c ,"R
paste(c("a","b"), "c", sep = ",")
出力
[1] "a,c" "b,c"R
paste(c("a","b"), "c", collapse = "|")
出力
[1] "a c|b c"R
paste(c("a","b"), "c", sep = ",", collapse = "|")
出力
[1] "a,c|b,c"(詳細については、?paste
ヘルプページの末尾の例を参照するか、example('paste')
を試してください。)
チャレンジ 3
ヘルプを使用して、タブ区切り(\t)の列と小数点が
“.”(ピリオド)で表される表形式のファイルを読み込むために使用できる関数(および関連するパラメータ)を見つけてください。
特に国際的な同僚と協力している場合、小数点の表記(例:コンマ対ピリオド)は異なる場合があるため、この確認が重要です。
ヒント:??"read table"
を使用して表形式データの読み込みに関連する関数を調べてください。
タブ区切りファイルを小数点がピリオドで表される形式で読み込む標準的な
R 関数は read.delim() です。
また、read.table(file, sep="\t")
を使用することもできます(read.table()
のデフォルトの小数点はピリオドです)。
ただし、データファイルにハッシュ(#)文字が含まれている場合は、comment.char
引数を変更する必要があるかもしれません。
その他のリソース
まとめ
- R のオンラインヘルプを取得するには
help()を使用します。
Content from データ構造
最終更新日:2024-11-22 | ページの編集
概要
質問
- R でデータをどのように読み取ることができますか?
- R の基本的なデータ型は何ですか?
- R でカテゴリ情報をどのように表現しますか?
目的
- 5 つの主なデータ型を特定できるようになる。
- データフレームを探索し始め、ベクトルやリストとの関連を理解する。
- R からオブジェクトの型、クラス、構造に関する質問ができるようになる。
- “names”、“class”、“dim” 属性の情報を理解する。
R の最も強力な機能の 1 つは、スプレッドシートや CSV
ファイルにすでに保存されているような表形式データを処理する能力です。まずは、data/
ディレクトリに feline-data.csv
という名前の小さなデータセットを作成しましょう:
R
cats <- data.frame(coat = c("calico", "black", "tabby"),
weight = c(2.1, 5.0, 3.2),
likes_catnip = c(1, 0, 1))
次に、cats を CSV
ファイルとして保存します。引数名を明示的に指定することは良い習慣であり、関数が変更されたデフォルト値を認識できます。この場合は
row.names = FALSE
を設定しています。引数名やそのデフォルト値を確認するには、?write.csv
を使用してヘルプファイルを表示してください。
R
write.csv(x = cats, file = "data/feline-data.csv", row.names = FALSE)
新しいファイル feline-data.csv
の内容は次の通りです:
ヒント: R でテキストファイルを編集する
または、テキストエディタ(Nano)や RStudio の File -> New
File -> Text File メニュー項目を使用して
data/feline-data.csv を作成することもできます。
このデータを R に読み込むには、以下のコマンドを使用します:
R
cats <- read.csv(file = "data/feline-data.csv")
cats
出力
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1read.table 関数は、CSV ファイル(csv = comma-separated
values)などのテキストファイルに保存された表形式データを読み取るために使用されます。タブやカンマは、CSV
ファイルでデータポイントを区切るために最も一般的に使用される記号です。R
では read.table の便利なバージョンとして
read.csv(データがカンマで区切られている場合)と
read.delim(データがタブで区切られている場合)が用意されています。この
3 つの中で、read.csv
が最も一般的に使用されます。必要に応じて、デフォルトの区切り記号を変更することもできます。
データが因子かどうかを確認する
最近、R がテキストデータを処理する方法が変更されました。以前は、R はテキストデータを自動的に “因子” という形式に変換していましたが、現在は “文字列” という形式で処理されるようになりました。因子の使用用途については後ほど学びますが、ほとんどの場合は必要なく、使用することで複雑になるだけです。そのため、新しい R バージョンではテキストデータが “文字列” として読み取られます。因子が自動的に作成されているかを確認し、必要に応じて文字列形式に変換してください:
- 入力データの型を確認するには、
str(cats)を入力します。 - 出力で、コロンの後にある 3
文字のコードを確認します:
numとchrのみが表示される場合は、レッスンを続けることができます。このボックスはスキップしてください。fctが見つかった場合は、次の手順に進んでください。 - R
が因子データを自動的に作成しないようにするには、以下のコードを実行します:
options(stringsAsFactors = FALSE)。その後、catsテーブルを再読み込みして変更を反映させます。 - R を再起動するたびに、このオプションを設定する必要があります。忘れないように、データを読み込む前にスクリプトの最初の行のいずれかに含めてください。
- R バージョン 4.0.0 以降では、テキストデータは因子に変換されなくなりました。問題を回避するためにこのバージョン以降をインストールすることを検討してください。研究所や会社のコンピュータを使用している場合は、管理者に依頼してください。
データセットをすぐに探索し始めることができます。たとえば、$
演算子を使用して列を指定します:
R
cats$weight
出力
[1] 2.1 5.0 3.2R
cats$coat
出力
[1] "calico" "black" "tabby" 列に対して操作を実行することもできます:
R
## たとえば、スケールが 2kg 軽いことが判明した場合:
cats$weight + 2
出力
[1] 4.1 7.0 5.2R
paste("My cat is", cats$coat)
出力
[1] "My cat is calico" "My cat is black" "My cat is tabby" しかし、次のコードではどうでしょうか?
R
cats$weight + cats$coat
エラー
Error in cats$weight + cats$coat: non-numeric argument to binary operatorここで何が起こったのかを理解することが、R でデータを成功裏に分析する鍵です。
データ型
最後のコマンドがエラーを返す理由が 2.1 と
"black"
を加算するのは無意味だからだと推測したなら、あなたは正しいです!これはプログラミングにおける重要な概念である
データ型
に関する直感をすでに持っているということです。データの型を調べるには、次のように入力します:
R
typeof(cats$weight)
出力
[1] "double"主なデータ型は次の 5
種類です:double、integer、complex、logical、character。
歴史的な理由で、double は numeric
とも呼ばれます。
R
typeof(3.14)
出力
[1] "double"R
typeof(1L) # L サフィックスを付けると数値を整数に強制します(R はデフォルトで浮動小数点数を使用)
出力
[1] "integer"R
typeof(1+1i)
出力
[1] "complex"R
typeof(TRUE)
出力
[1] "logical"R
typeof('banana')
出力
[1] "character"分析がどれだけ複雑であっても、R ではすべてのデータがこれらの基本的なデータ型のいずれかとして解釈されます。この厳格さには非常に重要な意味があります。
別の猫の詳細を追加した情報が、ファイル
data/feline-data_v2.csv に保存されています。
R
file.show("data/feline-data_v2.csv")
この新しい猫データを以前と同じ方法で読み込み、weight
列にどのようなデータ型が含まれているか確認します:
R
cats <- read.csv(file="data/feline-data_v2.csv")
typeof(cats
$weight)
出力
[1] "character"あらら、weight 列の型が double
ではなくなっています!以前と同じ計算を試みると、問題が発生します:
R
cats$weight + 2
エラー
Error in cats$weight + 2: non-numeric argument to binary operator何が起こったのでしょうか? 私たちが扱っている cats
データは データフレーム と呼ばれるものです。データフレームは、R
で最も一般的で多用途な データ構造 の 1 つです。
データフレームの特定の列には異なるデータ型を混在させることはできません。
この場合、R はデータフレーム列 weight のすべてを
double
として読み取らなかったため、列全体のデータ型がその列内のすべてに適した型に変わります。
R が CSV ファイルを読み取ると、それは データフレーム
として読み込まれます。そのため、cats CSV
ファイルを読み込むと、データフレームとして保存されます。データフレームは
str() 関数によって表示される最初の行で認識できます:
R
str(cats)
出力
'data.frame': 4 obs. of 3 variables:
$ coat : chr "calico" "black" "tabby" "tabby"
$ weight : chr "2.1" "5" "3.2" "2.3 or 2.4"
$ likes_string: int 1 0 1 1データフレーム は行と列で構成され、各列は同じ数の行を持ちます。データフレームの異なる列は異なるデータ型で構成できます(これがデータフレームを非常に柔軟にする理由です)が、特定の列内ではすべてが同じ型である必要があります(例:ベクトル、因子、リストなど)。
この振る舞いをさらに調査する間、猫のデータから余分な行を削除し、それを再読み込みしましょう:
feline-data.csv:
coat,weight,likes_catnip
calico,2.1,1
black,5.0,0
tabby,3.2,1そして RStudio 内で:
R
cats <- read.csv(file="data/feline-data.csv")
ベクトルと型の強制変換
この挙動をよりよく理解するために、別のデータ構造である ベクトル を紹介します。
R
my_vector <- vector(length = 3)
my_vector
出力
[1] FALSE FALSE FALSER
におけるベクトルは、基本的に順序付けられた要素のリストです。ただし、特別な条件として、ベクトル内のすべての要素は同じ基本データ型である必要があります。データ型を指定しない場合、デフォルトで
logical
型になります。また、任意の型の空のベクトルを宣言することも可能です。
R
another_vector <- vector(mode='character', length=3)
another_vector
出力
[1] "" "" ""あるオブジェクトがベクトルかどうかを確認することもできます:
R
str(another_vector)
出力
chr [1:3] "" "" ""このコマンドのやや難解な出力は、このベクトルに含まれる基本データ型(この場合は
chr、文字型)を示し、ベクトル内の要素数(この場合は
[1:3])、および実際に含まれる要素(この場合は空の文字列)を示します。同様に次のコマンドを実行すると、
R
str(cats$weight)
出力
num [1:3] 2.1 5 3.2cats$weight もベクトルであることがわかります。R
のデータフレームに読み込まれる列はすべてベクトルです。これが、R
が列内のすべての要素を同じ基本データ型に強制する理由の根本です。
討論 1
なぜ R は列に含まれるデータに対してこれほど厳格なのでしょうか? この厳格さは私たちにどのように役立つのでしょうか?
列内のすべてのデータが同じであることで、データに対して単純な仮定を行うことができます。たとえば、列の 1 つの要素を数値として解釈できるなら、列内のすべての要素を数値として解釈できます。そのため、毎回確認する必要がなくなります。この一貫性こそが、人々が「クリーンデータ」と呼ぶものです。長い目で見ると、この厳格な一貫性は R におけるデータ操作を非常に簡単にしてくれます。
ベクトルを結合する際の型の強制変換
明示的な内容を持つベクトルを c()
関数で作成できます:
R
combine_vector <- c(2,6,3)
combine_vector
出力
[1] 2 6 3これまで学んだ内容を考えると、次のコードは何を生成すると思いますか?
R
quiz_vector <- c(2,6,'3')
これは 型の強制変換
と呼ばれるもので、予想外の結果をもたらすことがあり、基本データ型と R
がそれをどのように解釈するかを理解する必要があります。R
は、異なる型(ここでは double と
character)が単一のベクトルに結合される場合、それらをすべて同じ型に強制します。例を見てみましょう:
R
coercion_vector <- c('a', TRUE)
coercion_vector
出力
[1] "a" "TRUE"R
another_coercion_vector <- c(0, TRUE)
another_coercion_vector
出力
[1] 0 1型の階層
型の強制変換ルールは次の通りです:logical -> integer ->
double (“numeric”) -> complex
-> character
この矢印は「変換される」と読めます。たとえば、logical
と character を結合すると、結果は character
に変換されます:
R
c('a', TRUE)
出力
[1] "a" "TRUE"character
ベクトルは、印刷時にクォートで囲まれていることで簡単に認識できます。
逆方向の強制変換を試みる場合は、as.
関数を使用します:
R
character_vector_example <- c('0','2','4')
character_vector_example
出力
[1] "0" "2" "4"R
character_coerced_to_double <- as.double(character_vector_example)
character_coerced_to_double
出力
[1] 0 2 4R
double_coerced_to_logical <- as.logical(character_coerced_to_double)
double_coerced_to_logical
出力
[1] FALSE TRUE TRUER が基本データ型を他の型に強制する際に驚くべきことが起こる場合があります!型の強制変換の細かい点はさておき、重要なのは:データが予想していた形式と異なる場合、それは型の強制変換が原因である可能性が高いです。ベクトルやデータフレームの列内のすべてのデータが同じ型であることを確認してください。さもなければ、予想外の問題が発生する可能性があります!
しかし、強制変換は非常に便利な場合もあります!たとえば、cats
データの likes_catnip 列は数値型ですが、実際には 1 と 0
がそれぞれ TRUE と FALSE
を表しています。このデータには logical
型を使用すべきです。この型は TRUE または FALSE
の 2 状態を持ち、データの意味に完全に一致します。この列を
logical に「強制変換」するには、as.logical
関数を使用します:
R
cats$likes_catnip
出力
[1] 1 0 1R
cats$likes_catnip <- as.logical(cats$likes_catnip)
cats$likes_catnip
出力
[1] TRUE FALSE TRUEチャレンジ 1
データ分析の重要な部分は、入力データのクリーンアップです。入力データがすべて同じ形式(例:数値)であることを知っていると、分析がはるかに簡単になります!型の強制変換に関する章で扱った猫のデータセットをクリーンアップしましょう。
コードテンプレートをコピー
RStudio で新しいスクリプトを作成し、以下のコードをコピー&ペーストしてください。その後、以下のタスクを参考にギャップ(______)を埋めてください。
# データを読み込み
cats <- read.csv("data/feline-data_v2.csv")
# 1. データを表示
_____
# 2. 表の概要をデータ型と共に表示
_____(cats)
# 3. "weight" 列の現在のデータ型 __________。
# 正しいデータ型は: ____________。
# 4. 4 番目の "weight" データポイントを指定された 2 つの値の平均に修正
cats$weight[4] <- 2.35
# 効果を確認するためにデータを再表示
cats
# 5. "weight" を正しいデータ型に変換
cats$weight <- ______________(cats$weight)
# 自分でテストするために平均を計算
mean(cats$weight)
# 正しい平均値(NA ではない)が表示されたら、演習は完了です!2. データ型の概要を表示する
データ型はデータ自体と同じくらい重要です。以前見た関数を使用して、cats
テーブルのすべての列のデータ型を表示します。
3. 必要
なデータ型はどれですか?
表示されるデータ型は、このデータ(猫の体重)には適していません。必要なデータ型はどれですか?
- なぜ
read.csv()関数は正しいデータ型を選ばなかったのでしょうか? - コメントのギャップに猫の体重に適したデータ型を埋めてください!
型の階層 のセクションに戻り、利用可能なデータ型を確認してください。
4. 問題のある値を修正する
問題のある 4 行目に新しい体重値を割り当てるコードが提供されています。実行する前に考えてみてください。この例のように数値を割り当てた後のデータ型はどうなりますか? 実行後にデータ型を確認して、自分の予測が正しいか確認してください。
5. 列 “weight” を正しいデータ型に変換する
猫の体重は数値です。しかし、列にはまだ適切なデータ型が設定されていません。この列を浮動小数点数に強制変換してください。
データ型を変換する関数は as.
で始まります。このスクリプトの上部で関数を確認するか、RStudio
のオートコンプリート機能を使用してください。 “as.”
と入力し、TAB キーを押します。
チャレンジ 1.5 の解答
歴史的な理由で、2 つの同義の関数があります:
R
cats$weight <- as.double(cats$weight)
cats$weight <- as.numeric(cats$weight)
基本的なベクトル関数
c()
関数を使用すると、既存のベクトルに新しい要素を追加することができます:
R
ab_vector <- c('a', 'b')
ab_vector
出力
[1] "a" "b"R
combine_example <- c(ab_vector, 'SWC')
combine_example
出力
[1] "a" "b" "SWC"また、数列を生成することも可能です:
R
mySeries <- 1:10
mySeries
出力
[1] 1 2 3 4 5 6 7 8 9 10R
seq(10)
出力
[1] 1 2 3 4 5 6 7 8 9 10R
seq(1, 10, by=0.1)
出力
[1] 1.0 1.1 1.2 1.3 1.4 1.5 1.6 1.7 1.8 1.9 2.0 2.1 2.2 2.3 2.4
[16] 2.5 2.6 2.7 2.8 2.9 3.0 3.1 3.2 3.3 3.4 3.5 3.6 3.7 3.8 3.9
[31] 4.0 4.1 4.2 4.3 4.4 4.5 4.6 4.7 4.8 4.9 5.0 5.1 5.2 5.3 5.4
[46] 5.5 5.6 5.7 5.8 5.9 6.0 6.1 6.2 6.3 6.4 6.5 6.6 6.7 6.8 6.9
[61] 7.0 7.1 7.2 7.3 7.4 7.5 7.6 7.7 7.8 7.9 8.0 8.1 8.2 8.3 8.4
[76] 8.5 8.6 8.7 8.8 8.9 9.0 9.1 9.2 9.3 9.4 9.5 9.6 9.7 9.8 9.9
[91] 10.0ベクトルについていくつかの質問をすることもできます:
R
sequence_example <- 20:25
head(sequence_example, n=2)
出力
[1] 20 21R
tail(sequence_example, n=4)
出力
[1] 22 23 24 25R
length(sequence_example)
出力
[1] 6R
typeof(sequence_example)
出力
[1] "integer"ベクトルの特定の要素を取得するには、角括弧記法を使用します:
R
first_element <- sequence_example[1]
first_element
出力
[1] 20特定の要素を変更するには、角括弧を矢印の右側に使用します:
R
sequence_example[1] <- 30
sequence_example
出力
[1] 30 21 22 23 24 25チャレンジ 2
1 から 26 までの数を含むベクトルを作成します。その後、このベクトルを 2 倍にします。
R
x <- 1:26
x <- x * 2
リスト
次に紹介するデータ構造は list
です。リストは他のデータ型よりもシンプルで、何でも入れることができるのが特徴です。ベクトルでは要素の基本データ型を統一する必要がありましたが、リストは異なるデータ型を持つことができます:
R
list_example <- list(1, "a", TRUE, 1+4i)
list_example
出力
[[1]]
[1] 1
[[2]]
[1] "a"
[[3]]
[1] TRUE
[[4]]
[1] 1+4istr()
を使用してオブジェクトの構造を表示すると、すべての要素のデータ型を確認できます:
R
str(list_example)
出力
List of 4
$ : num 1
$ : chr "a"
$ : logi TRUE
$ : cplx 1+4iリストの用途は何でしょうか?例えば、異なるデータ型を持つ関連データを整理できます。これは、Excel のスプレッドシートのように複数の表をまとめるのと似ています。他にも多くの用途があります。
次の章で、驚くかもしれない別の例を紹介します。
リストの特定の要素を取得するには 二重角括弧 を使用します:
R
list_example[[2]]
出力
[1] "a"リストの要素には 名前 を付けることもできます。名前を値の前に等号で指定します:
R
another_list <- list(title = "Numbers", numbers = 1:10, data = TRUE )
another_list
出力
$title
[1] "Numbers"
$numbers
[1] 1 2 3 4 5 6 7 8 9 10
$data
[1] TRUEこれにより 名前付きリスト が生成されます。これで新しいアクセス方法が追加されます!
R
another_list$title
出力
[1] "Numbers"名前
名前を使用すると、要素に意味を持たせることができます。これにより、データだけでなく説明情報も持つことができます。これはオブジェクトに貼り付けられるラベルのような メタデータ です。R ではこれは 属性 と呼ばれます。属性により、オブジェクトをさらに操作することが可能になります。ここでは、定義された名前で要素にアクセスすることができます。
名前を使用してベクトルやリストにアクセスする
名前付きリストの生成方法はすでに学びました。名前付きベクトルを生成する方法も非常に似ています。以前このような関数を見たことがあるはずです:
R
pizza_price <- c( pizzasubito = 5.64, pizzafresh = 6.60, callapizza = 4.50 )
しかし、要素の取得方法はリストとは異なります:
R
pizza_price["pizzasubito"]
出力
pizzasubito
5.64 リストのアプローチは機能しません:
R
pizza_price$pizzafresh
エラー
Error in pizza_price$pizzafresh: $ operator is invalid for atomic vectorsこのエラーメッセージを覚えておくと役立ちます。同じようなエラーに遭遇することが多いですが、これはリストと勘違いしてベクトルの要素にアクセスしようとした場合に発生します。
名前の取得と変更
名前だけに興味がある場合は、names()
関数を使用します:
R
names(pizza_price)
出力
[1] "pizzasubito" "pizzafresh" "callapizza" ベクトルの要素にアクセスしたり変更したりする方法を学びました。同じことが名前についても可能です:
R
names(pizza_price)[3]
出力
[1] "callapizza"R
names(pizza_price)[3] <- "call-a-pizza"
pizza_price
出力
pizzasubito pizzafresh call-a-pizza
5.64 6.60 4.50 チャレンジ 3
-
pizza_priceの名前のデータ型は何ですか?str()またはtypeof()関数を使用して調べてください。
オブジェクトの名前を取得するには、その名前を names(...)
で囲みます。同様に、名前のデータ型を取得するには、全体をさらに
typeof(...) で囲みます:
typeof(names(pizza))または、コードをわかりやすくするために新しい変数を使用します:
n <- names(pizza)
typeof(n)チャレンジ 4
既存のベクトルやリストの一部の名前を変更する代わりに、オブジェクトのすべての名前を設定することも可能です。次のコード形式を使用します(すべての大文字部分を置き換えてください):
names( OBJECT ) <- CHARACTER_VECTORアルファベットの各文字に番号を割り当てるベクトルを作成しましょう!
- 1 から 26 の数列を持つ
letter_noというベクトルを作成します。 - R には
LETTERSという組み込みオブジェクトがあります。これは A から Z までの 26 文字を含むベクトルです。この 26 文字をletter_noの名前として設定します。 -
letter_no["B"]を呼び出して、値が 2 であることを確認してください!
letter_no <- 1:26 # or seq(1,26)
names(letter_no) <- LETTERS
letter_no["B"]データフレーム
このレッスンの冒頭でデータフレームについて簡単に触れましたが、それはデータの表形式を表しています。例として示した猫のデータフレームについては詳細に掘り下げていませんでした:
R
cats
出力
coat weight likes_catnip
1 calico 2.1 TRUE
2 black 5.0 FALSE
3 tabby 3.2 TRUEここで少し驚くべきことに気づくかもしれません。次のコマンドを実行してみましょう:
R
typeof(cats)
出力
[1] "list"データフレームが「内部的にはリストのように見える」ことがわかります。以前、リストについて次のように説明しました:
リストは異なる型のデータを整理するためのもの
データフレームの列は、それぞれが異なる型のベクトルであり、同じ表に属することで整理されています。
データフレームは実際にはベクトルのリストです。データフレームが特別なのは、すべてのベクトルが同じ長さでなければならない点です。
この「特別さ」はどのようにオブジェクトに組み込まれているのでしょうか?R がそれを単なるリストではなく、表として扱うのはなぜでしょう?
R
class(cats)
出力
[1] "data.frame"クラス は名前と同様に、オブジェクトに付加される属性です。この属性は、そのオブジェクトが人間にとって何を意味するのかを示します。
ここで疑問に思うかもしれません:なぜオブジェクトの型を判断するための関数がもう一つ必要なのでしょうか?すでに
typeof() がありますよね?typeof()
はオブジェクトがコンピュータ内でどのように構築されているかを教えてくれます。一方、class()
はオブジェクトの人間にとっての意味を示します。したがって、typeof()
の出力は R で固定されています(主に 5
種類のデータ型)が、class() の出力は R
パッケージによって多様で拡張可能です。
cats
の例では、整数型、倍精度数値型、論理型の変数が含まれています。すでに見たように、データフレームの各列はベクトルです:
R
cats$coat
出力
[1] "calico" "black" "tabby" R
cats[,1]
出力
[1] "calico" "black" "tabby" R
typeof(cats[,1])
出力
[1] "character"R
str(cats[,1])
出力
chr [1:3] "calico" "black" "tabby"一方、各行は異なる変数の観測値であり、それ自体がデータフレームであり、異なる型の要素で構成されることができます:
R
cats[1,]
出力
coat weight likes_catnip
1 calico 2.1 TRUER
typeof(cats[1,])
出力
[1] "list"R
str(cats[1,])
出力
'data.frame': 1 obs. of 3 variables:
$ coat : chr "calico"
$ weight : num 2.1
$ likes_catnip: logi TRUEチャレンジ 5
データフレームから変数、観測値、要素を取得する方法はいくつかあります:
cats[1]cats[[1]]cats$coatcats["coat"]cats[1, 1]cats[, 1]cats[1, ]
これらの例を試して、それぞれが何を返すのかを説明してください。
ヒント: 返されるものを調べるには、typeof()
関数を使用してください。
R
cats[1]
出力
coat
1 calico
2 black
3 tabbyデータフレームはベクトルのリストと考えられます。単一ブラケット
[1]
はリストの最初のスライスを別のリストとして返します。この場合、それはデータフレームの最初の列です。
R
cats[[1]]
出力
[1] "calico" "black" "tabby" 二重ブラケット [[1]]
はリスト項目の内容を返します。この場合、最初の列の内容である
character 型のベクトルです。
R
cats$coat
出力
[1] "calico" "black" "tabby" $
を使用して名前で項目にアクセスします。coat
はデータフレームの最初の列であり、character
型のベクトルです。
R
cats["coat"]
出力
coat
1 calico
2 black
3 tabby単一ブラケット ["coat"]
を使用し、インデックス番号の代わりに列名を指定します。例 1
と同様に、返されるオブジェクトは list です。
R
cats[1, 1]
出力
[1] "calico"単一ブラケットを使用し、行と列の座標を指定します。この場合、1 行目 1 列目の値が返されます。オブジェクトは character 型のベクトルです。
R
cats[, 1]
出力
[1] "calico" "black" "tabby" 前の例と同様に単一ブラケットを使用し、行と列の座標を指定しますが、行座標が指定されていません。この場合、R は欠損値をその列のすべての要素として解釈し、ベクトル として返します。
R
cats[1, ]
出力
coat weight likes_catnip
1 calico 2.1 TRUE再び単一ブラケットを使用し、行と列の座標を指定しますが、今回は列座標が指定されていません。返される値は 1 行目のすべての値を含む list です。
ヒント: データフレーム列の名前変更
データフレームには列名があり、names()
関数でアクセスできます:
R
names(cats)
出力
[1] "coat" "weight" "likes_catnip"cats の 2
番目の列の名前を変更したい場合は、names(cats) の 2
番目の要素に新しい名前を割り当てます:
R
names(cats)[2] <- "weight_kg"
cats
出力
coat weight_kg likes_catnip
1 calico 2.1 TRUE
2 black 5.0 FALSE
3 tabby 3.2 TRUE行列(Matrix)
最後に紹介するのは行列です。ゼロで満たされた行列を宣言してみましょう:
R
matrix_example <- matrix(0, ncol=6, nrow=3)
matrix_example
出力
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 0 0 0 0 0 0
[2,] 0 0 0 0 0 0
[3,] 0 0 0 0 0 0行列を特別なものにしているのは dim() 属性です:
R
dim(matrix_example)
出力
[1] 3 6他のデータ構造と同様に、行列について質問することも可能です:
R
typeof(matrix_example)
出力
[1] "double"R
class(matrix_example)
出力
[1] "matrix" "array" R
str(matrix_example)
出力
num [1:3, 1:6] 0 0 0 0 0 0 0 0 0 0 ...R
nrow(matrix_example)
出力
[1] 3R
ncol(matrix_example)
出力
[1] 6チャレンジ 6
次のコードの結果はどうなるでしょうか?
R
length(matrix_example)
出力
[1] 18実行して確認してください。予想は当たりましたか?なぜそのような結果になるのでしょうか?
行列は次元属性を持つベクトルであるため、length
は行列内の要素の総数を返します:
R
matrix_example <- matrix(0, ncol=6, nrow=3)
length(matrix_example)
出力
[1] 18チャレンジ 7
1 から 50 の数値を含む、列数 5、行数 10 の行列を作成します。
デフォルトの動作として、この行列は列ごとに値が埋められますか、それとも行ごとですか?
その動作を変更する方法を調べてください。(ヒント:matrix
のドキュメントを参照)
R
x <- matrix(1:50, ncol=5, nrow=10)
x <- matrix(1:50, ncol=5, nrow=10, byrow = TRUE) # 行ごとに埋める
チャレンジ 8
このワークショップの次のセクションに対応する 2 つの要素を持つリストを作成します:
- データ型
- データ構造
各データ型およびデータ構造の名前を文字型ベクトルに格納してください。
R
dataTypes <- c('double', 'complex', 'integer', 'character', 'logical')
dataStructures <- c('data.frame', 'vector', 'list', 'matrix')
answer <- list(dataTypes, dataStructures)
チャレンジ 9
以下の行列の R 出力を考えてみてください:
出力
[,1] [,2]
[1,] 4 1
[2,] 9 5
[3,] 10 7この行列を作成するために使用された正しいコマンドはどれでしょうか?各コマンドを確認し、入力する前に正しいものを考えてください。
他のコマンドでどのような行列が作成されるかを考えてみてください。
matrix(c(4, 1, 9, 5, 10, 7), nrow = 3)matrix(c(4, 9, 10, 1, 5, 7), ncol = 2, byrow = TRUE)matrix(c(4, 9, 10, 1, 5, 7), nrow = 2)matrix(c(4, 1, 9, 5, 10, 7), ncol = 2, byrow = TRUE)
R
matrix(c(4, 1, 9, 5, 10, 7), ncol = 2, byrow = TRUE)
まとめ
-
read.csvを使用して R で表形式データを読み取ります。 - R の基本データ型は、double、integer、complex、logical、character です。
- データフレームや行列のようなデータ構造は、リストやベクトルを基にし、いくつかの属性が追加されています。
Content from データフレームの操作
最終更新日:2024-11-22 | ページの編集
概要
質問
- データフレームをどのように操作できますか?
目的
- 行や列を追加または削除する。
- 2 つのデータフレームを結合する。
- データフレームのサイズ、列のクラス、名前、最初の数行などの基本的なプロパティを表示する。
これまでに、R の基本的なデータ型とデータ構造について学びました。以降の作業は、それらのツールを操作することに集約されます。最も頻繁に登場するのは、CSV ファイルから情報を読み込んで作成するデータフレームです。このレッスンでは、データフレームの操作についてさらに学びます。
データフレームに列や行を追加する
データフレームの列はベクトルであるため、列全体でデータ型が一貫しています。そのため、新しい列を追加したい場合は、まず新しいベクトルを作成します:
R
age <- c(2, 3, 5)
cats
出力
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1これを列として追加するには、次のようにします:
R
cbind(cats, age)
出力
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5ただし、データフレームの行数と異なる要素数を持つベクトルを追加しようとすると失敗します:
R
age <- c(2, 3, 5, 12)
cbind(cats, age)
エラー
Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 4R
age <- c(2, 3)
cbind(cats, age)
エラー
Error in data.frame(..., check.names = FALSE): arguments imply differing number of rows: 3, 2なぜ失敗するのでしょうか?R は、新しい列の各行に 1 つの要素が必要だと考えています:
R
nrow(cats)
出力
[1] 3R
length(age)
出力
[1] 2したがって、nrow(cats) と length(age)
が等しい必要があります。新しいデータフレームを作成して、cats
に上書きしてみましょう。
R
age <- c(2, 3, 5)
cats <- cbind(cats, age)
次に、行を追加してみましょう。データフレームの行はリストであることを既に学びました:
R
newRow <- list("tortoiseshell", 3.3, TRUE, 9)
cats <- rbind(cats, newRow)
新しい行が正しく追加されたことを確認します。
R
cats
出力
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5
4 tortoiseshell 3.3 1 9行を削除する
データフレームに行や列を追加する方法を学びました。次に、行を削除する方法を見てみましょう。
R
cats
出力
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5
4 tortoiseshell 3.3 1 9最後の行を削除したデータフレームを取得するには:
R
cats[-4, ]
出力
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5コンマの後に何も指定しないことで、4 行目全体を削除することを示します。
複数の行を削除することもできます。たとえば、次のようにベクトル内に行番号を指定します:cats[c(-3,-4), ]
列を削除する
データフレームの列を削除することもできます。「age」列を削除する場合、変数番号またはインデックスを使用する方法があります。
R
cats[,-4]
出力
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1
4 tortoiseshell 3.3 1コンマの前に何も指定しないことで、すべての行を保持することを示します。
または、インデックス名と %in%
演算子を使用して列を削除することもできます。%in%
演算子は、左側の引数(ここでは cats
の名前)の各要素について「この要素は右側の引数に含まれますか?」と尋ねます。
R
drop <- names(cats) %in% c("age")
cats[,!drop]
出力
coat weight likes_catnip
1 calico 2.1 1
2 black 5.0 0
3 tabby 3.2 1
4 tortoiseshell 3.3 1論理演算子(%in%
など)による部分集合化については、次のエピソードで詳しく説明します。詳細は
論理演算を使用した部分集合化
を参照してください。
データフレームの結合
データフレームにデータを追加する際に覚えておくべき重要な点は、列はベクトル、行はリストであることです。2
つのデータフレームを rbind
を使用して結合することもできます:
R
cats <- rbind(cats, cats)
cats
出力
coat weight likes_catnip age
1 calico 2.1 1 2
2 black 5.0 0 3
3 tabby 3.2 1 5
4 tortoiseshell 3.3 1 9
5 calico 2.1 1 2
6 black 5.0 0 3
7 tabby 3.2 1 5
8 tortoiseshell 3.3 1 9チャレンジ 1
次の構文を使用して、新しいデータフレームを R 内で作成できます:
R
df <- data.frame(id = c("a", "b", "c"),
x = 1:3,
y = c(TRUE, TRUE, FALSE))
以下の情報を持つデータフレームを作成してください:
- 名
- 姓
- ラッキーナンバー
次に、rbind
を使用して隣の人のエントリを追加します。最後に、cbind
を使用して「コーヒーブレイクの時間ですか?」という質問への各人の回答を含む列を追加してください。
R
df <- data.frame(first = c("Grace"),
last = c("Hopper"),
lucky_number = c(0))
df <- rbind(df, list("Marie", "Curie", 238) )
df <- cbind(df, coffeetime = c(TRUE, TRUE))
実用的な例
これまで、猫データを使ってデータフレーム操作の基本を学びました。次に、これらのスキルを使用して、より現実的なデータセットを扱います。以前ダウンロードした
gapminder データセットを読み込んでみましょう:
R
gapminder <- read.csv("data/gapminder_data.csv")
その他のヒント
タブ区切り値ファイル(.tsv)を扱う場合は、区切り文字として
"\\t"を指定するか、read.delim()を使用します。ファイルをインターネットから直接ダウンロードしてコンピュータの指定したローカルフォルダに保存するには、
download.file関数を使用できます。保存されたファイルをread.csv関数で読み込む例:
R
download.file("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/main/episodes/data/gapminder_data.csv", destfile = "data/gapminder_data.csv")
gapminder <- read.csv("data/gapminder_data.csv")
- また、ファイルパスの代わりに Web アドレスを
read.csvに指定して、ファイルを直接 R に読み込むこともできます。この場合、ローカルにファイルを保存する必要はありません。例:
R
gapminder <- read.csv("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/main/episodes/data/gapminder_data.csv")
readxl パッケージ を使用すると、Excel スプレッドシートをプレーンテキストに変換せずに直接読み込むことができます。
"stringsAsFactors"引数を使用すると、文字列を因子として読み込むか文字列として読み込むかを指定できます。R バージョン 4.0 以降では、デフォルトで文字列は文字型として読み込まれますが、古いバージョンでは因子として読み込まれるのがデフォルトでした。詳細は前のエピソードのコールアウトを参照してください。
gapminder
データセットを調べてみましょう。最初に行うべきことは、str
を使用してデータの構造を確認することです:
R
str(gapminder)
出力
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...gapminder
の構造を調べる別の方法として、summary
関数を使用します。この関数は R
のさまざまなオブジェクトで使用できます。データフレームの場合、summary
は各列の数値的、表形式、または記述的な概要を提供します。数値または整数型の列は記述統計(四分位数や平均値)で、文字列型の列はその長さ、クラス、モードで説明されます。
R
summary(gapminder)
出力
country year pop continent
Length:1704 Min. :1952 Min. :6.001e+04 Length:1704
Class :character 1st Qu.:1966 1st Qu.:2.794e+06 Class :character
Mode :character Median :1980 Median :7.024e+06 Mode :character
Mean :1980 Mean :2.960e+07
3rd Qu.:1993 3rd Qu.:1.959e+07
Max. :2007 Max. :1.319e+09
lifeExp gdpPercap
Min. :23.60 Min. : 241.2
1st Qu.:48.20 1st Qu.: 1202.1
Median :60.71 Median : 3531.8
Mean :59.47 Mean : 7215.3
3rd Qu.:70.85 3rd Qu.: 9325.5
Max. :82.60 Max. :113523.1 str や summary
関数と合わせて、typeof
関数を使ってデータフレームの個々の列を調べることもできます:
R
typeof(gapminder$year)
出力
[1] "integer"R
typeof(gapminder$country)
出力
[1] "character"R
str(gapminder$country)
出力
chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...データフレームの次元に関する情報も調べることができます。str(gapminder) の出力によると、gapminder には
6 つの変数の 1704
個の観測値があります。このことを覚えた上で、次のコードが何を返すか考えてみてください:
R
length(gapminder)
出力
[1] 6データフレームの長さが行数(1704)であると考えるのが妥当ですが、実際にはそうではありません。データフレームはベクトルや因子のリストで構成されていることを思い出してください:
R
typeof(gapminder)
出力
[1] "list"length が 6 を返した理由は、gapminder が 6
列のリストで構築されているためです。データセットの行数と列数を取得するには次のようにします:
R
nrow(gapminder)
出力
[1] 1704R
ncol(gapminder)
出力
[1] 6あるいは、両方を一度に取得するには:
R
dim(gapminder)
出力
[1] 1704 6すべての列のタイトルを調べることもできます。後でアクセスする際に便利です:
R
colnames(gapminder)
出力
[1] "country" "year" "pop" "continent" "lifeExp" "gdpPercap"ここで、R が報告する構造が自分の直感や予想と一致しているかどうかを確認することが重要です。各列のデータ型が妥当かどうかを確認してください。そうでない場合は、これまで学んだ R のデータ解釈の仕組みや、一貫性の重要性に基づいて問題を解決する必要があります。
データ型や構造が合理的であることを確認したら、データの探索を開始しましょう。最初の数行を確認します:
R
head(gapminder)
出力
country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
4 Afghanistan 1967 11537966 Asia 34.020 836.1971
5 Afghanistan 1972 13079460 Asia 36.088 739.9811
6 Afghanistan 1977 14880372 Asia 38.438 786.1134チャレンジ 2
データの最後の数行や中間のいくつかの行も確認するのが良い習慣です。これをどのように行いますか?
特に中間の行を探すのは難しくありませんが、ランダムな行をいくつか取得することもできます。これをどのようにコード化しますか?
最後の数行を確認するには、R に既にある関数を使用すれば簡単です:
R
tail(gapminder)
tail(gapminder, n = 15)
では、途中の任意の行を確認するにはどうすればよいでしょうか?
再現性のある分析を確保するために、コードをスクリプトファイルに保存し、後で再利用できるようにしましょう。
チャレンジ 3
File -> New File -> R Script に移動し、gapminder
データセットを読み込むための R スクリプトを作成します。このスクリプトを
scripts/
ディレクトリに保存し、バージョン管理に追加してください。
その後、source
関数を使用してスクリプトを実行します。ファイルパスを引数として指定するか、RStudio
の「Source」ボタンを押します。
source
関数はスクリプト内で別のスクリプトを使用するために使用できます。同じ種類のファイルを何度も読み込む必要がある場合、一度スクリプトとして保存すれば、以降はそれを繰り返し利用できます。
R
download.file("https://raw.githubusercontent.com/swcarpentry/r-novice-gapminder/main/episodes/data/gapminder_data.csv", destfile = "data/gapminder_data.csv")
gapminder <- read.csv(file = "data/gapminder_data.csv")
データを gapminder
変数に読み込むには次のようにします:
R
source(file = "scripts/load-gapminder.R")
チャレンジ 4
str(gapminder)
の出力をもう一度読み、リストやベクトルについて学んだこと、および
colnames や dim
の出力を活用して、str
が表示する内容を説明してください。理解できない部分があれば、隣の人と相談してみてください。
オブジェクト gapminder
はデータフレームで、列は次のようになっています:
-
countryとcontinentは文字列(character)。 -
yearは整数型のベクトル。 -
pop、lifeExp、gdpPercapは数値型のベクトル。
まとめ
- 新しい列をデータフレームに追加するには
cbind()を使用します。 - 新しい行をデータフレームに追加するには
rbind()を使用します。 - データフレームから行を削除します。
- データフレームの構造を理解するために、
str()、summary()、nrow()、ncol()、dim()、colnames()、head()、typeof()を使用します。 -
read.csv()を使用して CSV ファイルを読み込みます。 - データフレームの
length()が何を表しているのか理解します。
Content from データの部分集合化
最終更新日:2024-11-22 | ページの編集
概要
質問
- R でデータの部分集合をどのように扱うことができますか?
目的
- ベクトル、因子、行列、リスト、データフレームの部分集合化を学ぶ
- インデックス、名前、比較演算を使って個々の要素や複数の要素を抽出する方法を理解する
- さまざまなデータ構造から要素をスキップまたは削除する方法を習得する
R には強力な部分集合化のための演算子が数多く用意されています。それらを習得することで、あらゆる種類のデータセットで複雑な操作を簡単に行えるようになります。
あらゆる種類のオブジェクトを部分集合化するために 6 つの異なる方法があり、データ構造ごとに 3 種類の部分集合化演算子があります。
では、R の基本となる数値ベクトルを使って始めましょう。
R
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
x
出力
a b c d e
5.4 6.2 7.1 4.8 7.5 アトミックベクトル
R では、文字列、数値、または論理値を含む単純なベクトルを アトミックベクトル と呼びます。これは、さらに単純化できないためです。
このようにダミーベクトルを作成しました。この中身をどのように取得するのでしょうか?
インデックスを使用した要素のアクセス
ベクトルの要素を抽出するには、対応するインデックスを指定します(1 から始まります):
R
x[1]
出力
a
5.4 R
x[4]
出力
d
4.8 角括弧 [] 演算子は関数であり、ベクトルや行列に対して「n
番目の要素を取得する」という意味を持ちます。
複数の要素を一度に取得することもできます:
R
x[c(1, 3)]
出力
a c
5.4 7.1 またはベクトルの一部(スライス)を取得することも可能です:
R
x[1:4]
出力
a b c d
5.4 6.2 7.1 4.8 :
演算子は左の値から右の値までの数値のシーケンスを生成します。
R
1:4
出力
[1] 1 2 3 4R
c(1, 2, 3, 4)
出力
[1] 1 2 3 4同じ要素を複数回取得することもできます:
R
x[c(1,1,3)]
出力
a a c
5.4 5.4 7.1 ベクトルの長さを超えるインデックスを指定すると、R は欠損値を返します:
R
x[6]
出力
<NA>
NA これは NA を含む長さ 1 のベクトルであり、名前も
NA です。
0 番目の要素を要求すると、空のベクトルが返されます:
R
x[0]
出力
named numeric(0)R のベクトルの番号付けは 1 から始まる
多くのプログラミング言語(C や Python など)では、ベクトルの最初の要素のインデックスは 0 です。一方、R では最初の要素は 1 です。
要素のスキップと削除
ベクトルのインデックスに負の数を使用すると、指定した要素を除くすべての要素が返されます:
R
x[-2]
出力
a c d e
5.4 7.1 4.8 7.5 複数の要素をスキップすることも可能です:
R
x[c(-1, -5)] # または x[-c(1,5)]
出力
b c d
6.2 7.1 4.8 ヒント: 演算の順序
ベクトルの一部をスキップしようとすると、新しい人はよく間違えます。例えば:
R
x[-1:3]
このコードは次のようなエラーを返します:
エラー
Error in x[-1:3]: only 0's may be mixed with negative subscriptsこれは演算の順序が関係しています。:
は実際には関数であり、最初の引数を -1、2 番目の引数を
3
としてシーケンスを生成します:c(-1, 0, 1, 2, 3)。
正しい解決策は、この関数呼び出しを括弧で囲み、-
演算子を結果に適用することです:
R
x[-(1:3)]
出力
d e
4.8 7.5 ベクトルから要素を削除するには、結果を変数に再割り当てする必要があります:
R
x <- x[-4]
x
出力
a b c e
5.4 6.2 7.1 7.5 チャレンジ 1
次のコードが与えられた場合:
R
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
print(x)
出力
a b c d e
5.4 6.2 7.1 4.8 7.5 次の出力を生成する少なくとも 2 つの異なるコマンドを考え出してください:
出力
b c d
6.2 7.1 4.8 2 つの異なるコマンドを見つけたら、隣の人と比較してみてください。異なる戦略を持っていましたか?
R
x[2:4]
出力
b c d
6.2 7.1 4.8 R
x[-c(1,5)]
出力
b c d
6.2 7.1 4.8 R
x[c(2,3,4)]
出力
b c d
6.2 7.1 4.8 名前によるサブセット
インデックスではなく名前を使って要素を抽出することができます:
R
x <- c(a=5.4, b=6.2, c=7.1, d=4.8, e=7.5) # ベクトルにその場で名前を付ける
x[c("a", "c")]
出力
a c
5.4 7.1 これは、オブジェクトをサブセットする際に、通常より信頼性の高い方法です。
サブセット操作を連続して行う場合、要素の位置が変わることがありますが、名前は常に変わりません!
他の論理演算を用いたサブセット
論理ベクトルを使ってサブセットを取ることもできます:
R
x[c(FALSE, FALSE, TRUE, FALSE, TRUE)]
出力
c e
7.1 7.5 比較演算子(例:>、<、==)は論理ベクトルを生成するため、それを使ってベクトルを簡潔にサブセットできます。以下のステートメントは、前の例と同じ結果を返します。
R
x[x > 7]
出力
c e
7.1 7.5 このステートメントを分解すると、まず x>7
が評価されて論理ベクトル c(FALSE, FALSE, TRUE, FALSE, TRUE)
が生成され、それに基づいて x の要素が選択されます。
名前によるインデックス操作を模倣するには、==
を使うことができます(比較には = ではなく ==
を使用する必要がある点に注意):
R
x[names(x) == "a"]
出力
a
5.4 ヒント: 複数の論理条件の結合
複数の論理条件を結合したい場合があります。たとえば、特定の範囲内の寿命を持つアジアまたはヨーロッパに位置するすべての国を見つけたいとします。このような場合、R では論理ベクトルを結合するための以下の演算子を使用できます:
-
&(論理AND): 左右がともにTRUEの場合にTRUEを返します。 -
|(論理OR): 左右のいずれか、または両方がTRUEの場合にTRUEを返します。
& や | の代わりに
&& や ||
が使われることもありますが、これらはベクトルの最初の要素だけを見て残りを無視します。データ解析では、通常1文字の演算子(&
や
|)を使用し、2文字の演算子はプログラミング(ステートメントの実行を決定する際など)で使用してください。
-
!(論理NOT):TRUEをFALSEに、FALSEをTRUEに変換します。単一の条件(例:!TRUEはFALSEになる)や、ベクトル全体(例:!c(TRUE, FALSE)はc(FALSE, TRUE)になる)を否定できます。
また、単一のベクトル内の要素を比較するために、all(すべての要素が
TRUE の場合に TRUE を返す)や
any(1つ以上の要素が TRUE の場合に
TRUE を返す)関数を使用できます。
チャレンジ 2
以下のコードを用いて:
R
x <- c(5.4, 6.2, 7.1, 4.8, 7.5)
names(x) <- c('a', 'b', 'c', 'd', 'e')
print(x)
出力
a b c d e
5.4 6.2 7.1 4.8 7.5 x から 4 より大きく 7
未満の値を返すサブセットコマンドを書いてください。
R
x_subset <- x[x<7 & x>4]
print(x_subset)
出力
a b d
5.4 6.2 4.8 ヒント: 重複する名前
1つのベクトル内で複数の要素が同じ名前を持つことも可能です(データフレームでは、列名が重複することはありますが、行名は一意である必要があります)。以下の例を考えてみてください:
R
x <- 1:3
x
出力
[1] 1 2 3R
names(x) <- c('a', 'a', 'a')
x
出力
a a a
1 2 3 R
x['a'] # 最初の値のみを返す
出力
a
1 R
x[names(x) == 'a'] # すべての値を返す
出力
a a a
1 2 3 ヒント: 演算子に関するヘルプを得る方法
演算子を引用符で囲むことで、ヘルプを検索できます:help("%in%") または ?"%in%"。
名前付き要素をスキップする
名前付き要素をスキップまたは削除するのは少し難しいです。文字列を否定してスキップしようとすると、R は「文字列を否定する方法が分からない」というやや分かりにくいエラーを出します:
R
x <- c(a=5.4, b=6.2, c=7.1, d=4.8, e=7.5) # もう一度その場でベクトルに名前を付ける
x[-"a"]
エラー
Error in -"a": invalid argument to unary operatorしかし、!=(等しくない)演算子を使用して論理ベクトルを構築すれば、望む動作を実現できます:
R
x[names(x) != "a"]
出力
b c d e
6.2 7.1 4.8 7.5 複数の名前付きインデックスをスキップするのはさらに難しいです。例えば、"a"
と "c" を削除しようとして次のように試みます:
R
x[names(x)!=c("a","c")]
警告
Warning in names(x) != c("a", "c"): longer object length is not a multiple of
shorter object length出力
b c d e
6.2 7.1 4.8 7.5 R は 何か をしましたが、警告を出しており、それが示す通り
間違った結果 を返しました("c"
要素がまだベクトルに含まれています)。
!=
がこの場合に何を実際にしているのかは、非常に良い質問です。
リサイクル
このコードの比較部分を見てみましょう:
R
names(x) != c("a", "c")
警告
Warning in names(x) != c("a", "c"): longer object length is not a multiple of
shorter object length出力
[1] FALSE TRUE TRUE TRUE TRUEnames(x)[3] != "c" は明らかに偽なのに、なぜ R
はこのベクトルの3番目の要素に TRUE
を返すのでしょうか?!= を使用すると、R
は左辺の各要素を右辺の対応する要素と比較しようとします。左辺と右辺の長さが異なる場合はどうなりますか?
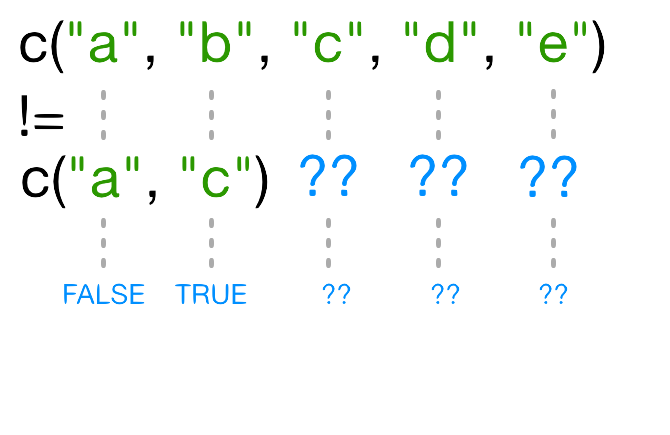
一方のベクトルがもう一方より短い場合、短いベクトルは リサイクル されます:
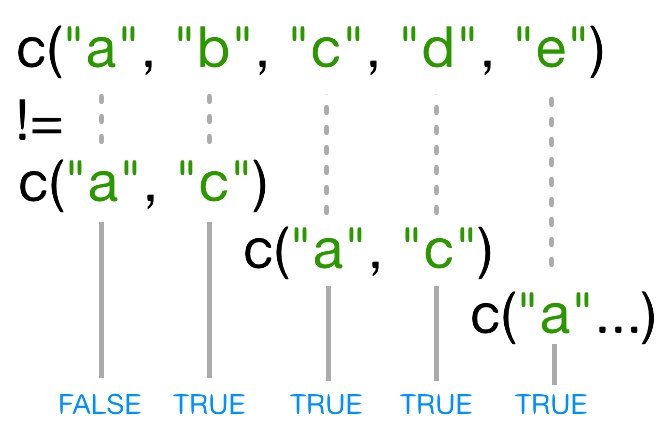
この場合、R は c("a", "c") を必要な回数だけ繰り返して
names(x)
と一致させます(例:c("a","c","a","c","a"))。
再利用された "a" が names(x)
の3番目の要素と一致しないため、!= の結果が
TRUE になります。
リサイクルによりこのような間違いが発生するのを防ぐには
%in%
演算子を使用します。この演算子は左辺の各要素について、右辺の中にその要素が存在するかどうかを確認します。今回は値を除外したいので、!
演算子も使用します:
R
x[! names(x) %in% c("a","c") ]
出力
b d e
6.2 4.8 7.5 チャレンジ 3
ベクトルの要素を、特定のリスト内のいずれかと一致させる操作は、データ解析で非常に一般的なタスクです。例えば、gapminder
データセットには country と continent
の変数がありますが、これらの間の情報は含まれていません。東南アジアの情報を抽出したいとします。このとき、どのようにして東南アジアのすべての国について
TRUE、それ以外を FALSE
とする論理ベクトルを作成しますか?
以下のデータを使用します:
R
seAsia <- c("Myanmar","Thailand","Cambodia","Vietnam","Laos")
## エピソード2でダウンロードした gapminder データを読み込む
gapminder <- read.csv("data/gapminder_data.csv", header=TRUE)
## データフレームから `country` 列を抽出(詳細は後述);
## factor を文字列に変換;
## 重複しない要素のみ取得
countries <- unique(as.character(gapminder$country))
以下の3つの方法を試し、それぞれがどのように(正しくない、または正しい方法で)動作するのか説明してください:
-
間違った方法(
==のみを使用)
-
不格好な方法(論理演算子
==と|を使用)
-
エレガントな方法(
%in%を使用)
間違った方法
countries==seAsia
この方法では、警告("In countries == seAsia : 長いオブジェクトの長さが短いオブジェクトの長さの倍数ではありません")が表示され、誤った結果(すべてFALSEのベクトル)が返されます。これは、seAsiaの再利用された値が正しい位置に一致しないためです。不格好な方法
以下のコードでは正しい値を得ることができますが、非常に冗長で扱いにくいです:
R
(countries=="Myanmar" | countries=="Thailand" |
countries=="Cambodia" | countries == "Vietnam" | countries=="Laos")
または、countries==seAsia[1] | countries==seAsia[2] | ...
のように記述します。
リストが長い場合、さらに複雑になります。
-
エレガントな方法
countries %in% seAsia
この方法は正確で、記述が簡単で可読性も高いです。
特殊値の扱い
R
では、欠損値、無限値、未定義値を処理できない関数に出会うことがあります。
そのようなデータをフィルタリングするために、以下の特殊な関数を使用できます:
-
is.na:ベクトル、行列、またはデータフレーム内のNA(またはNaN)を含む位置を返します。 - 同様に、
is.nanとis.infiniteは、それぞれNaNとInfに対応します。 -
is.finite:NA、NaN、Infを含まない位置を返します。 -
na.omit:ベクトルからすべての欠損値を除外します。
因子のサブセット
ベクトルのサブセット方法を学んだところで、他のデータ構造のサブセットについて考えてみましょう。
因子のサブセットは、ベクトルのサブセットと同じ方法で行えます。
R
f <- factor(c("a", "a", "b", "c", "c", "d"))
f[f == "a"]
出力
[1] a a
Levels: a b c dR
f[f %in% c("b", "c")]
出力
[1] b c c
Levels: a b c dR
f[1:3]
出力
[1] a a b
Levels: a b c d要素をスキップしても、そのカテゴリが因子レベルから削除されるわけではありません:
R
f[-3]
出力
[1] a a c c d
Levels: a b c d行列のサブセット
行列は [
関数を使用してサブセットします。この場合、2つの引数を取り、1つ目は行、2つ目は列に適用されます:
R
set.seed(1)
m <- matrix(rnorm(6*4), ncol=4, nrow=6)
m[3:4, c(3,1)]
出力
[,1] [,2]
[1,] 1.12493092 -0.8356286
[2,] -0.04493361 1.5952808行または列全体を取得する場合は、1つ目または2つ目の引数を空白のままにします:
R
m[, c(3,4)]
出力
[,1] [,2]
[1,] -0.62124058 0.82122120
[2,] -2.21469989 0.59390132
[3,] 1.12493092 0.91897737
[4,] -0.04493361 0.78213630
[5,] -0.01619026 0.07456498
[6,] 0.94383621 -1.989351701つの行または列のみを取得すると、結果が自動的にベクトルに変換されます:
R
m[3,]
出力
[1] -0.8356286 0.5757814 1.1249309 0.9189774結果を行列として保持するには、第3引数 を指定して
drop = FALSE を設定します:
R
m[3, , drop=FALSE]
出力
[,1] [,2] [,3] [,4]
[1,] -0.8356286 0.5757814 1.124931 0.9189774行または列の外側をアクセスしようとすると、R はエラーをスローします:
R
m[, c(3,6)]
エラー
Error in m[, c(3, 6)]: subscript out of boundsヒント: 高次元配列
多次元配列の場合、[
の各引数が次元に対応します。例えば、3次元配列の場合、最初の3つの引数が行、列、および深さ次元に対応します。
行列はベクトルであるため、1つの引数だけを使用してサブセットを取ることもできます:
R
m[5]
出力
[1] 0.3295078これは通常あまり有用ではなく、読み取りづらい場合があります。ただし、行列がデフォルトで 列優先フォーマット に配置されていることを理解するのに役立ちます。つまり、ベクトルの要素は列ごとに配置されます:
R
matrix(1:6, nrow=2, ncol=3)
出力
[,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6行ごとに行列を埋めたい場合は、byrow=TRUE
を使用します:
R
matrix(1:6, nrow=2, ncol=3, byrow=TRUE)
出力
[,1] [,2] [,3]
[1,] 1 2 3
[2,] 4 5 6行列は、行および列のインデックスの代わりに、その行名および列名を使用してサブセットを取ることもできます。
チャレンジ 4
以下のコードを用いて:
R
m <- matrix(1:18, nrow=3, ncol=6)
print(m)
出力
[,1] [,2] [,3] [,4] [,5] [,6]
[1,] 1 4 7 10 13 16
[2,] 2 5 8 11 14 17
[3,] 3 6 9 12 15 18- 以下のコマンドのうち、11 と 14 を抽出するものはどれでしょうか?
A. m[2,4,2,5]
B. m[2:5]
C. m[4:5,2]
D. m[2,c(4,5)]
D
リストのサブセット
ここでは新しいサブセット演算子を紹介します。リストをサブセットするためには、3つの関数を使用します。これらは、原子ベクトルや行列を学ぶ際にも登場しました:[,
[[, $ です。
[
を使用すると、常にリストが返されます。リストから要素を「抽出」するのではなく「サブセット」したい場合に使用します。
R
xlist <- list(a = "Software Carpentry", b = 1:10, data = head(mtcars))
xlist[1]
出力
$a
[1] "Software Carpentry"このコードは、1つの要素を含むリスト を返します。
リストの要素は、原子ベクトルと同じ方法でサブセットできます。ただし、比較演算は再帰的ではなく、リスト内のデータ構造に基づいて条件が評価されるため、リスト内の個々の要素には適用されません。
R
xlist[1:2]
出力
$a
[1] "Software Carpentry"
$b
[1] 1 2 3 4 5 6 7 8 9 10リストの個々の要素を抽出するには、二重角括弧関数 [[
を使用する必要があります。
R
xlist[[1]]
出力
[1] "Software Carpentry"この結果はリストではなくベクトルであることに注意してください。
複数の要素を一度に抽出することはできません:
R
xlist[[1:2]]
エラー
Error in xlist[[1:2]]: subscript out of boundsまた、要素をスキップすることもできません:
R
xlist[[-1]]
エラー
Error in xlist[[-1]]: invalid negative subscript in get1index <real>ただし、名前を使用して要素をサブセットおよび抽出することは可能です:
R
xlist[["a"]]
出力
[1] "Software Carpentry"$
演算子は、名前で要素を抽出するための簡潔な記法を提供します:
R
xlist$data
出力
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1チャレンジ 5
以下のリストが与えられています:
R
xlist <- list(a = "Software Carpentry", b = 1:10, data = head(mtcars))
リストとベクトルのサブセット方法を用いて、xlist
から数字の 2 を抽出してください。
ヒント:数字の 2 はリスト内の "b" に含まれています。
R
xlist$b[2]
出力
[1] 2R
xlist[[2]][2]
出力
[1] 2R
xlist[["b"]][2]
出力
[1] 2チャレンジ 6
以下の線形モデルが与えられています:
R
mod <- aov(pop ~ lifeExp, data=gapminder)
残差の自由度を抽出してください(ヒント:attributes()
が役立ちます)。
R
attributes(mod) ## `df.residual` は `mod` の名前の1つです
R
mod$df.residual
データフレーム
データフレームは内部的にはリストであることを覚えておきましょう。そのため、同様のルールが適用されます。ただし、データフレームは2次元のオブジェクトでもあります:
[
に1つの引数を与える場合、リストと同様に動作し、それぞれのリスト要素が列に対応します。結果として得られるオブジェクトはデータフレームになります:
R
head(gapminder[3])
出力
pop
1 8425333
2 9240934
3 10267083
4 11537966
5 13079460
6 14880372同様に、[[ を使用すると、単一の列
を抽出します:
R
head(gapminder[["lifeExp"]])
出力
[1] 28.801 30.332 31.997 34.020 36.088 38.438$
演算子は、列を名前で抽出するための便利なショートカットを提供します:
R
head(gapminder$year)
出力
[1] 1952 1957 1962 1967 1972 19772つの引数を与えると、[
は行列と同じように動作します:
R
gapminder[1:3,]
出力
country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007単一行をサブセットすると、結果はデータフレームになります(要素が混合型のためです):
R
gapminder[3,]
出力
country year pop continent lifeExp gdpPercap
3 Afghanistan 1962 10267083 Asia 31.997 853.1007ただし、単一列をサブセットすると結果はベクトルになります(第3引数
drop = FALSE を指定することで変更可能)。
チャレンジ 7
以下の一般的なデータフレームサブセットエラーを修正してください:
- 年 1957 の観測値を抽出する
- 1列目から4列目以外のすべての列を抽出する
R
gapminder[,-1:4]
- 寿命が80年以上の行を抽出する
R
gapminder[gapminder$lifeExp > 80]
- 1行目と4列目、5列目(
continentとlifeExp)を抽出する
R
gapminder[1, 4, 5]
- 応用:年 2002 年と 2007 年の情報を含む行を抽出する
R
gapminder[gapminder$year == 2002 | 2007,]
以下の一般的なデータフレームサブセットエラーを修正:
- 年 1957 の観測値を抽出する
R
# gapminder[gapminder$year = 1957,]
gapminder[gapminder$year == 1957,]
- 1列目から4列目以外のすべての列を抽出する
R
# gapminder[,-1:4]
gapminder[,-c(1:4)]
- 寿命が80年以上の行を抽出する
R
# gapminder[gapminder$lifeExp > 80]
gapminder[gapminder$lifeExp > 80,]
- 1行目と4列目、5列目(
continentとlifeExp)を抽出する
R
# gapminder[1, 4, 5]
gapminder[1, c(4, 5)]
- 応用:年 2002 年と 2007 年の情報を含む行を抽出する
R
# gapminder[gapminder$year == 2002 | 2007,]
gapminder[gapminder$year == 2002 | gapminder$year == 2007,]
gapminder[gapminder$year %in% c(2002, 2007),]
チャレンジ 8
なぜ
gapminder[1:20]はエラーを返すのでしょうか?gapminder[1:20, ]とはどう異なるのでしょうか?行 1 から 9 と 19 から 23 のみを含む新しい
data.frameを作成し、それをgapminder_smallと名付けてください。この操作は 1 ステップまたは 2 ステップで行うことができます。
gapminderは data.frame なので、2 次元でサブセット化する必要があります。gapminder[1:20, ]は最初の 20 行とすべての列をサブセット化して返します。
R
gapminder_small <- gapminder[c(1:9, 19:23),]
まとめ
- R のインデックスは 0 ではなく 1 から始まります。
-
[]を使用して位置による個々の値にアクセスします。 -
[low:high]を使用してデータのスライスにアクセスします。 -
[c(...)]を使用して任意のデータセットにアクセスします。 - 論理演算や論理ベクトルを使用してデータのサブセットにアクセスします。
Content from 制御フロー
最終更新日:2024-11-22 | ページの編集
概要
質問
- Rでデータに依存した選択を行うにはどうすればよいですか?
- Rで操作を繰り返すにはどうすればよいですか?
目的
-
if...else文やifelse()を使って条件分岐文を書く。 -
for()ループを理解し、記述する。
コードを書く際に、特定の条件が満たされた場合にのみアクションを実行したり、特定の回数アクションを実行したりすることがよくあります。
Rでは、制御フローを設定するいくつかの方法があります。条件分岐文の場合、最も一般的に使用される構文は次のとおりです。
R
# if
if (条件が真の場合) {
アクションを実行
}
# if ... else
if (条件が真の場合) {
アクションを実行
} else { # つまり、条件が偽の場合、
代替のアクションを実行
}
例えば、変数 x
が特定の値を持つ場合にメッセージを表示するようにしたいとします。
R
x <- 8
if (x >= 10) {
print("xは10以上です")
}
x
出力
[1] 8この場合、x
が10以上ではないため、コンソールにメッセージは表示されません。else
文を追加することで、10未満の数値の場合に異なるメッセージを表示できます。
R
x <- 8
if (x >= 10) {
print("xは10以上です")
} else {
print("xは10未満です")
}
出力
[1] "xは10未満です"複数の条件をテストする場合は、else if
を使用できます。
R
x <- 8
if (x >= 10) {
print("xは10以上です")
} else if (x > 5) {
print("xは5より大きく、10未満です")
} else {
print("xは5以下です")
}
出力
[1] "xは5より大きく、10未満です"重要: if()
文内で条件を評価するとき、Rは論理要素(TRUE または
FALSE)を探します。これにより、初心者にとって頭痛の種になる場合があります。例えば:
R
x <- 4 == 3
if (x) {
"4は3と等しい"
} else {
"4は3と等しくない"
}
出力
[1] "4は3と等しくない"この場合、x が FALSE
であるため、「等しくない」メッセージが表示されます。
R
x <- 4 == 3
x
出力
[1] FALSEチャレンジ 1
if() 文を使用して、gapminder データセットに
2002
年のレコードがあるかどうかを報告する適切なメッセージを表示してください。
次に、2012 年の場合も同様に行ってください。
チャレンジ 1 の解答は、any()
関数を使用しない方法から見ていきます。 まず、gapminder$year
の要素のうち 2002
と等しいものを記述する論理ベクトルを取得します。
R
gapminder[(gapminder$year == 2002),]
次に、gapminder data.frame の 2002
年に対応する行数をカウントします。
R
rows2002_number <- nrow(gapminder[(gapminder$year == 2002),])
2002 年のレコードが存在するかどうかは、rows2002_number
が 1 以上であることに等しいです。
R
rows2002_number >= 1
これらをまとめると次のようになります:
R
if(nrow(gapminder[(gapminder$year == 2002),]) >= 1){
print("2002年のレコードが見つかりました。")
}
このすべては、any()
を使うことでより簡潔に記述できます。論理条件は次のように表せます:
R
if(any(gapminder$year == 2002)){
print("2002年のレコードが見つかりました。")
}
次のような警告メッセージが出た人はいますか?
エラー
Error in if (gapminder$year == 2012) {: the condition has length > 1if() 関数は単一の(長さ 1
の)入力のみを受け付けるため、ベクトルを使用するとエラーを返します。
そのため、if() を使用する際は、入力が単一(長さ
1)であることを確認する必要があります。
ヒント: 組み込みの ifelse()
関数
R は上述のように構成された if() や
else if() 文を受け付けますが、ifelse()
関数を使用する方法もあります。
この関数は単一およびベクトル入力の両方を受け入れ、次の構文で構成されます:
R
# ifelse 関数
ifelse(条件が真の場合, アクションを実行, 代替アクションを実行)
例えば:
R
y <- -3
ifelse(y < 0, "yは負の数です", "yは正の数または0です")
出力
[1] "yは負の数です"ヒント: any() と
all()
any() 関数は、ベクトル内に少なくとも 1 つの
TRUE 値がある場合に TRUE
を返します。それ以外の場合は FALSE を返します。 これは
%in% 演算子と同様の方法で使用できます。 all()
関数はその名前が示す通り、ベクトル内のすべての値が TRUE
の場合にのみ TRUE を返します。
繰り返し操作
値のセットを反復処理し、順序が重要で各値に対して同じ操作を実行したい場合、for()
ループが役立ちます。 for() ループは シェルのレッスン
でも紹介されました。 for()
ループは最も柔軟な繰り返し操作のひとつですが、その分、正しく使用するのが難しい場合もあります。
多くの R ユーザーは、for()
ループを学ぶことを勧めますが、繰り返しの順序が重要でない場合には使用を避けるようアドバイスしています。
順序が重要でない場合には、purrr
パッケージなどのベクトル化された代替手段を学ぶべきです。
これにより、計算効率が向上します。
for() ループの基本構造は次のとおりです:
例えば:
R
for (i in 1:10) {
print(i)
}
出力
[1] 1
[1] 2
[1] 3
[1] 4
[1] 5
[1] 6
[1] 7
[1] 8
[1] 9
[1] 10ここで、1:10
はその場で作成されたベクトルです。他のベクトルを使って反復処理することもできます。
2つのことを同時に反復処理するには、別の for()
ループをネストして使用できます。
R
for (i in 1:5) {
for (j in c('a', 'b', 'c', 'd', 'e')) {
print(paste(i,j))
}
}
出力
[1] "1 a"
[1] "1 b"
[1] "1 c"
[1] "1 d"
[1] "1 e"
[1] "2 a"
[1] "2 b"
[1] "2 c"
[1] "2 d"
[1] "2 e"
[1] "3 a"
[1] "3 b"
[1] "3 c"
[1] "3 d"
[1] "3 e"
[1] "4 a"
[1] "4 b"
[1] "4 c"
[1] "4 d"
[1] "4 e"
[1] "5 a"
[1] "5 b"
[1] "5 c"
[1] "5 d"
[1] "5 e"出力を見ると、最初のインデックス (i) が 1
に設定されているとき、2 番目のインデックス (j)
がその全範囲を反復処理することがわかります。 j
のインデックスがすべて処理されると、i
がインクリメントされます。このプロセスは各 for()
ループで最後のインデックスが使用されるまで続きます。
結果を表示する代わりに、ループの出力を新しいオブジェクトに書き込むこともできます。
R
output_vector <- c()
for (i in 1:5) {
for (j in c('a', 'b', 'c', 'd', 'e')) {
temp_output <- paste(i, j)
output_vector <- c(output_vector, temp_output)
}
}
output_vector
出力
[1] "1 a" "1 b" "1 c" "1 d" "1 e" "2 a" "2 b" "2 c" "2 d" "2 e" "3 a" "3 b"
[13] "3 c" "3 d" "3 e" "4 a" "4 b" "4 c" "4 d" "4 e" "5 a" "5 b" "5 c" "5 d"
[25] "5 e"このアプローチは便利ですが、結果を「増やしていく」(結果オブジェクトを段階的に構築する)ことは計算効率が悪いため、大量の値を反復処理する場合は避けるべきです。
ヒント: 結果を増やさないでください
初心者や経験豊富な R
ユーザーがつまずく最大の原因のひとつは、for
ループが進むにつれて結果オブジェクト(ベクトル、リスト、マトリックス、データフレームなど)を構築していくことです。
コンピュータはこれを非常に苦手とするため、計算速度が著しく低下する可能性があります。
そのため、適切な次元を持つ空の結果オブジェクトを事前に定義し、各反復で結果を適切な場所に格納するほうがはるかに効率的です。
より効率的な方法は、値を入力する前に(空の)出力オブジェクトを定義することです。 この例では少し複雑に見えますが、それでも効率的です。
R
output_matrix <- matrix(nrow = 5, ncol = 5)
j_vector <- c('a', 'b', 'c', 'd', 'e')
for (i in 1:5) {
for (j in 1:5) {
temp_j_value <- j_vector[j]
temp_output <- paste(i, temp_j_value)
output_matrix[i, j] <- temp_output
}
}
output_vector2 <- as.vector(output_matrix)
output_vector2
出力
[1] "1 a" "2 a" "3 a" "4 a" "5 a" "1 b" "2 b" "3 b" "4 b" "5 b" "1 c" "2 c"
[13] "3 c" "4 c" "5 c" "1 d" "2 d" "3 d" "4 d" "5 d" "1 e" "2 e" "3 e" "4 e"
[25] "5 e"ヒント: While ループ
特定の条件が満たされている間、操作を繰り返す必要がある場合があります。
これには while() ループを使用します。
R
while(条件が真の間){
アクションを実行
}
R は条件が満たされていることを「TRUE」として解釈します。
例えば、以下のような while ループは、
runif() 関数を使用して 0 から 1
の範囲のランダムな数を生成し、その数が 0.1
未満になるまで繰り返します。
R
z <- 1
while(z > 0.1){
z <- runif(1)
cat(z, "\n")
}
出力
0.5074782
0.3067685
0.4269077
0.6931021
0.08513597 while()
ループは常に適切とは限りません。条件が常に満たされる場合、無限ループに陥ってしまう可能性があるため注意が必要です。
チャレンジ 2
オブジェクト output_vector と
output_vector2 を比較してください。
それらは同じでしょうか?もし違う場合、その理由は何でしょうか?
最後のコードブロックをどのように変更すれば、output_vector2
を output_vector と同じにできますか?
2つのベクトルが同一であるかどうかは、all()
関数を使って確認できます:
R
all(output_vector == output_vector2)
ただし、output_vector のすべての要素が
output_vector2 内に存在することは確認できます:
R
all(output_vector %in% output_vector2)
逆も同様です:
R
all(output_vector2 %in% output_vector)
したがって、output_vector と output_vector2
の要素は異なる順序で並んでいるだけです。
これは、as.vector()
が入力マトリックスの列を基に要素を出力するためです。
output_matrix
を確認すると、要素を行ごとに取得する必要があることがわかります。
解決策は、output_matrix
を転置することです。これには、転置関数 t()
を使用するか、要素を適切な順序で入力する方法があります。
最初の解決策では、次のコードを変更します:
R
output_vector2 <- as.vector(output_matrix)
以下のように変更します:
R
output_vector2 <- as.vector(t(output_matrix))
2つ目の解決策では、次のコードを変更します:
R
output_matrix[i, j] <- temp_output
以下のように変更します:
R
output_matrix[j, i] <- temp_output
チャレンジ 3
gapminder
データを大陸ごとにループし、平均寿命が50年より小さいか大きいかを出力するスクリプトを書いてください。
ステップ 1: 大陸ベクトルのすべてのユニークな値を抽出できることを確認します。
R
gapminder <- read.csv("data/gapminder_data.csv")
unique(gapminder$continent)
ステップ 2: 各大陸ごとにループして、データの各
subset について平均寿命を計算する必要があります。
次のように行えます:
- ‘continent’ のユニークな値ごとにループする。
- 各大陸の値について、そのサブセットを一時変数に格納する。
- 計算された平均寿命をユーザーに出力として返す:
R
for (iContinent in unique(gapminder$continent)) {
tmp <- gapminder[gapminder$continent == iContinent, ]
cat(iContinent, mean(tmp$lifeExp, na.rm = TRUE), "\n")
rm(tmp)
}
ステップ 3:
この課題では、平均寿命が50未満または50以上の場合のみ出力する必要があります。
そのため、計算された平均寿命が閾値より上か下かを評価し、結果に応じて出力を行う
if() 条件を追加します。
以下に(3)を修正したものを示します:
3a. 計算された平均寿命が閾値 (50年) より小さい場合は大陸名と「50未満」を、閾値以上の場合は大陸名と「50以上」を返します。
R
thresholdValue <- 50
for (iContinent in unique(gapminder$continent)) {
tmp <- mean(gapminder[gapminder$continent == iContinent, "lifeExp"])
if (tmp < thresholdValue){
cat("平均寿命は", iContinent, "で", thresholdValue, "年未満です\n")
} else {
cat("平均寿命は", iContinent, "で", thresholdValue, "年以上です\n")
} # if else 条件の終了
rm(tmp)
} # for ループの終了
チャレンジ 4
チャレンジ 3 のスクリプトを変更して、今度は各国ごとにループを行います。 この際、平均寿命が50年未満、50年から70年の間、または70年を超えているかを出力してください。
チャレンジ 3 の解答を修正し、lowerThreshold と
upperThreshold という2つの閾値を追加し、if-else
文を拡張します:
R
lowerThreshold <- 50
upperThreshold <- 70
for (iCountry in unique(gapminder$country)) {
tmp <- mean(gapminder[gapminder$country == iCountry, "lifeExp"])
if(tmp < lowerThreshold) {
cat("平均寿命は", iCountry, "で", lowerThreshold, "年未満です\n")
} else if(tmp > lowerThreshold && tmp < upperThreshold) {
cat("平均寿命は", iCountry, "で", lowerThreshold, "年から", upperThreshold, "年の間です\n")
} else {
cat("平均寿命は", iCountry, "で", upperThreshold, "年以上です\n")
}
rm(tmp)
}
チャレンジ 5 - 応用
gapminder
データセットの各国ごとにループし、その国が「B」で始まるかどうかをテストし、
平均寿命が50年未満の場合には、時間に対する寿命の折れ線グラフを作成するスクリプトを書いてください。
「B」で始まる国を見つけるには、Unix
シェルのレッスン で紹介された grep()
コマンドを使用します。 まず以下を試します:
R
grep("^B", unique(gapminder$country))
ただし、このコマンドは「B」で始まる country
のファクター変数のインデックスを返します。
値を取得するには、value=TRUE オプションを
grep() コマンドに追加する必要があります:
R
grep("^B", unique(gapminder$country), value = TRUE)
これらの国を candidateCountries
という変数に格納し、その変数内の各エントリをループします。
ループ内で各国の平均寿命を評価し、平均寿命が50未満の場合、with()
と subset() を使用して寿命の推移をプロットします:
R
thresholdValue <- 50
candidateCountries <- grep("^B", unique(gapminder$country), value = TRUE)
for (iCountry in candidateCountries) {
tmp <- mean(gapminder[gapminder$country == iCountry, "lifeExp"])
if (tmp < thresholdValue) {
cat("平均寿命は", iCountry, "で", thresholdValue, "年未満です。寿命グラフを描画中...\n")
with(subset(gapminder, country == iCountry),
plot(year, lifeExp,
type = "o",
main = paste(iCountry, "の寿命の推移"),
ylab = "平均寿命",
xlab = "年"
) # plot の終了
) # with の終了
} # if の終了
rm(tmp)
} # for ループの終了
まとめ
-
ifとelseを使用して選択肢を作る。 -
forを使用して操作を繰り返す。
Content from ggplot2 を使用した出版品質のグラフィック作成
最終更新日:2024-11-22 | ページの編集
概要
質問
- R で出版品質のグラフィックを作成するにはどうすればよいですか?
目的
- ggplot2 を使用して出版品質のグラフィックを作成できるようになる。
- ggplot のプロットに幾何学(geometry)、美的要素(aesthetic)、統計レイヤー(statistics layers)を適用する。
- 異なる色、形状、線を使用してプロットの美的要素を操作する。
- スケールを変換したり、グループごとにパネル分けすることでデータ可視化を改善する。
- ggplot を使用して作成したプロットをディスクに保存する。
データをプロットすることは、データや変数間の関係を素早く探る最良の方法の一つです。
R には 3 つの主なプロットシステムがあります。 基本プロットシステム、lattice パッケージ、そして ggplot2 パッケージです。
今回は ggplot2 パッケージを学びます。ggplot2 は、出版品質のグラフィックを作成するために最も効果的です。
ggplot2 は “グラフィックの文法 (grammar of graphics)” に基づいています。この概念は、任意のプロットが次のような同じ構成要素のセットから構築できるというものです:データセット、美的要素のマッピング、およびグラフィックのレイヤー。
データセットは、ユーザーが提供するデータです。
美的要素のマッピング (Mapping aesthetics) は、データをグラフィックに結びつけます。 これにより、X軸やY軸に何をプロットするか、データポイントのサイズや色をどうするかなど、グラフの見た目にデータを反映させます。
レイヤーは、ggplot2 の実際のグラフィック出力です。 レイヤーはプロットの種類(散布図、ヒストグラムなど)、座標系(長方形、極座標など)、およびその他の重要なプロットの側面を決定します。 このようなグラフィックのレイヤーの概念は、Photoshop、Illustrator、Inkscape などの画像編集プログラムを使用した経験があれば馴染みがあるかもしれません。
では、以前使用した gapminder データを使って例を構築してみましょう。
最も基本的な関数は ggplot
で、これにより新しいプロットを作成していることを R に知らせます。
ggplot
関数に渡した引数は、プロット全体に適用される「グローバル」オプションです。
R
library("ggplot2")
ggplot(data = gapminder)

ここでは ggplot
を呼び出し、プロットに表示するデータを指定しました。
これはプロットを実際に描画するのに十分な情報ではなく、他の要素を追加するための白紙の状態を作成するだけです。
次に、aes
関数を使用して美的要素のマッピングを追加します。
aes
は、データ内の変数が図の美的プロパティ(例:X軸やY軸の位置)にどのようにマップされるかを
ggplot に伝えます。
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp))
ここでは、gapminder データフレームの “gdpPercap” 列を X
軸に、“lifeExp” 列を Y 軸にプロットするよう ggplot
に指示しました。 aes
にこれらの列を明示的に渡す必要がないのは、ggplot が
data から自動的に列を見つけるためです。
プロットを完成させる最後のステップは、レイヤーを追加してデータを視覚的に表現する方法を
ggplot に指示することです。
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point()
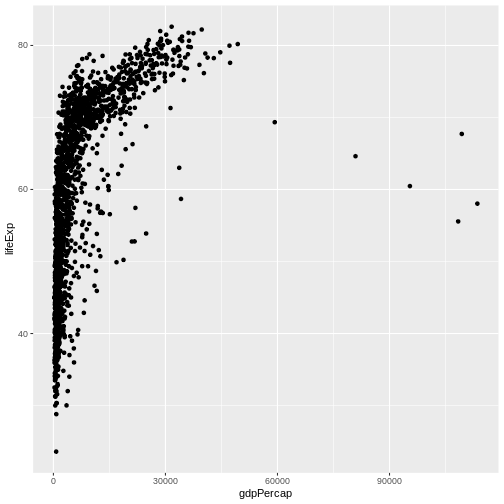
ここでは geom_point を使用して、X と
Y
の関係を散布図として視覚的に表現するように指示しました。
チャレンジ 1
例を修正して、平均寿命が時間とともにどのように変化したかを示す図を作成してください:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) + geom_point()
ヒント:gapminder データセットには “year” という列があり、これを X 軸に表示する必要があります。
次のような解答例があります:
R
ggplot(data = gapminder, mapping = aes(x = year, y = lifeExp)) + geom_point()
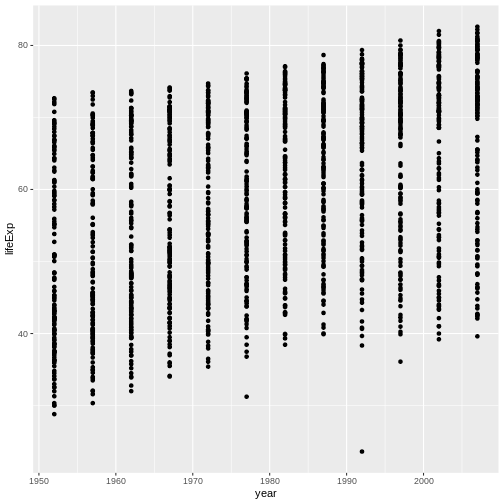
レイヤー (Layers)
散布図は、時間経過による変化を可視化するための最適な方法ではないかもしれません。 代わりに、データを折れ線グラフとして視覚化してみましょう:
R
ggplot(data = gapminder, mapping = aes(x=year, y=lifeExp, color=continent)) +
geom_line()
geom_point
レイヤーを追加する代わりに、geom_line
レイヤーを追加しました。
…
ヒント: 美的要素に値を設定する
これまで、美的要素(例:色)をデータ内の変数にマッピングする方法を見てきました。
たとえば、geom_line(mapping = aes(color=continent))
を使用すると、各大陸に異なる色が割り当てられます。
しかし、すべての線の色を青に変更したい場合はどうでしょう?
geom_line(mapping = aes(color="blue"))
が機能すると考えるかもしれませんが、そうではありません。
特定の変数にマッピングしたくない場合は、aes()
の外側で色を指定します: geom_line(color="blue")
のように記述します。
チャレンジ 3
前の例からポイントレイヤーとラインレイヤーの順序を入れ替えてみてください。何が起こるでしょうか?
ラインがポイントの上に描画されるようになります!
R
ggplot(data = gapminder, mapping = aes(x=year, y=lifeExp, group=country)) +
geom_point() + geom_line(mapping = aes(color=continent))
変換と統計
ggplot2 を使用すると、データの上に統計モデルを簡単にオーバーレイできます。 例を示すために、最初の例に戻ります:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point()
GDP の極端な値が原因で、ポイント間の関係を確認するのが難しいです。 X軸の単位スケールを変更するには scale 関数を使用します。これにより、データ値と美的要素の視覚値の間のマッピングが制御されます。 また、大量のクラスター化されたデータを扱う際に役立つ alpha 関数を使用してポイントの透過性を変更することもできます。
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(alpha = 0.5) + scale_x_log10()
scale_x_log10 関数はプロットの座標系に変換を適用します。
これにより、10 の倍数が左から右に均等に配置されます。たとえば、1,000 の
GDP は 10,000 の値から 100,000
の値までと同じ水平距離を持つようになります。
これにより、X軸に沿ったデータの分布を視覚化しやすくなります。
ヒント:美的要素をマッピングではなく値に設定するリマインダー
geom_point(alpha = 0.5)
を使用したことに注意してください。前回のヒントで述べたように、aes()
関数の外側で設定を行うと、この値がすべてのポイントに適用されます。
ただし、他の美的要素設定と同様に、alpha
をデータ内の変数にマッピングすることも可能です。
たとえば、geom_point(mapping = aes(alpha = continent))
を使用すると、大陸ごとに異なる透過性を指定できます。
データに簡単な関係を適合させるには、geom_smooth
レイヤーを追加します:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(alpha = 0.5) + scale_x_log10() + geom_smooth(method="lm")
出力
`geom_smooth()` using formula = 'y ~ x'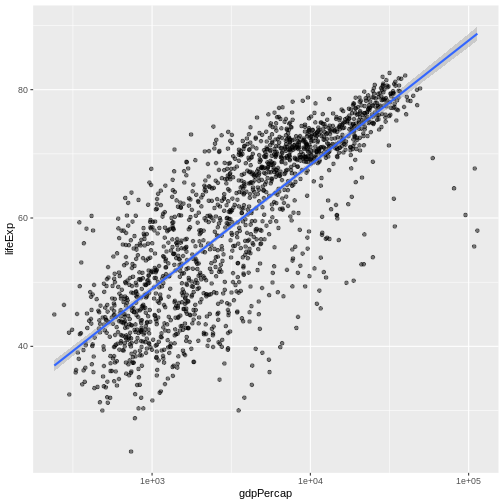
ラインを太くするには、geom_smooth レイヤー内で
linewidth 美的要素を設定します:
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(alpha = 0.5) + scale_x_log10() + geom_smooth(method="lm", linewidth=1.5)
出力
`geom_smooth()` using formula = 'y ~ x'
美的要素を指定する方法には2つあります。ここでは、geom_smooth
に引数として linewidth
を渡して美的要素を設定し、geom 全体に適用しました。
以前のレッスンでは、データ変数とその視覚表現の間にマッピングを定義するために
aes 関数を使用しました。
チャレンジ 4a
前の例のポイントレイヤーで、ポイントの色とサイズを変更してください。
ヒント:aes 関数を使用しないでください。
ヒント:ポイントに対する linewidth に相当するものは
size です。
次のような解答例があります: color 引数が
aes() 関数の外側で指定されていることに注意してください。
これは、グラフ上のすべてのデータポイントに適用され、特定の変数に関連付けられていません。
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp)) +
geom_point(size=3, color="orange") + scale_x_log10() +
geom_smooth(method="lm", linewidth=1.5)
出力
`geom_smooth()` using formula = 'y ~ x'
チャレンジ 4b
チャレンジ 4a
の解答を修正し、ポイントの形状を変更し、大陸ごとに異なる色と新しいトレンドラインを追加してください。
ヒント:color 引数は美的マッピング内で使用できます。
次のような解答例があります: color 引数を
aes() 関数内で指定することで、特定の変数に接続できます。
一方で、shape 引数は aes()
呼び出しの外側で指定されており、すべてのデータポイントを同じ形状に変更します。
R
ggplot(data = gapminder, mapping = aes(x = gdpPercap, y = lifeExp, color = continent)) +
geom_point(size=3, shape=17) + scale_x_log10() +
geom_smooth(method="lm", linewidth=1.5)
出力
`geom_smooth()` using formula = 'y ~ x'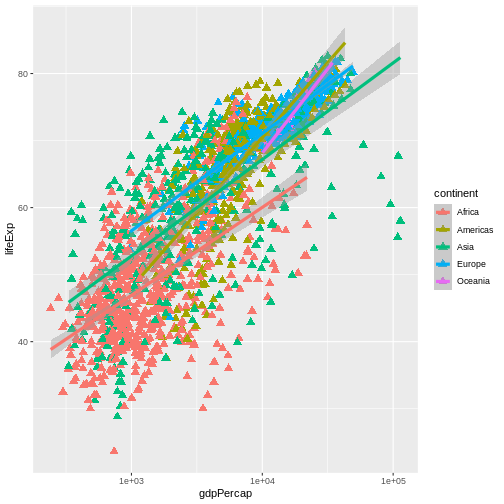
マルチパネル図
以前、すべての国における時間経過による平均寿命の変化を1つのプロットで視覚化しました。 代わりに、facet パネルのレイヤーを追加して、複数のパネルに分割することもできます。
ヒント
アメリカ大陸にある国のみを含むデータのサブセットを作成します。 これには25カ国が含まれますが、これにより図が煩雑になり始めます。 X軸ラベルを回転させて読みやすさを維持するために「テーマ」定義を適用します。 ggplot2ではほぼすべてがカスタマイズ可能です。
R
americas <- gapminder[gapminder$continent == "Americas",]
ggplot(data = americas, mapping = aes(x = year, y = lifeExp)) +
geom_line() +
facet_wrap( ~ country) +
theme(axis.text.x = element_text(angle = 45))
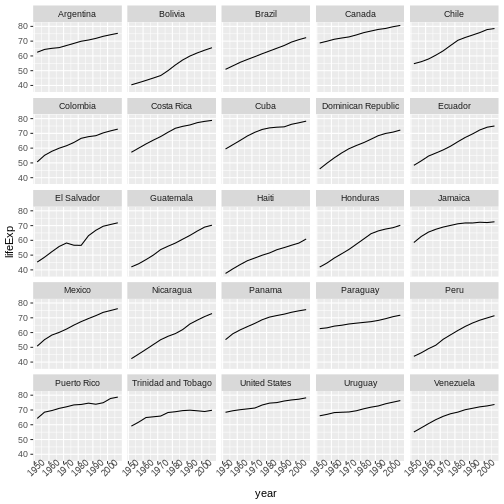
facet_wrap
レイヤーには、チルダ(~)で表される「式」が引数として渡されます。
これにより、Rは gapminder データセットの country
列内のユニークな値ごとにパネルを描画します。
テキストの変更
この図を出版用にクリーンアップするには、いくつかのテキスト要素を変更する必要があります。 X軸は煩雑すぎ、Y軸にはデータフレーム内の列名ではなく「Life expectancy」と記載する必要があります。
これを行うには、いくつかの異なるレイヤーを追加します。テーマ
(theme)
レイヤーは軸テキストや全体的なテキストサイズを制御します。
軸ラベル、プロットタイトル、および凡例のタイトルは labs
関数を使用して設定できます。 凡例タイトルは、aes
仕様で使用した名前を使って設定します。 以下では、色の凡例タイトルを
color = "Continent" として設定しています。
同様に、塗りつぶしの凡例タイトルは fill = "MyTitle"
として設定します。
R
ggplot(data = americas, mapping = aes(x = year, y = lifeExp, color=continent)) +
geom_line() + facet_wrap( ~ country) +
labs(
x = "Year", # X軸のタイトル
y = "Life expectancy", # Y軸のタイトル
title = "Figure 1", # 図のメインタイトル
color = "Continent" # 凡例のタイトル
) +
theme(axis.text.x = element_text(angle = 90, hjust = 1))
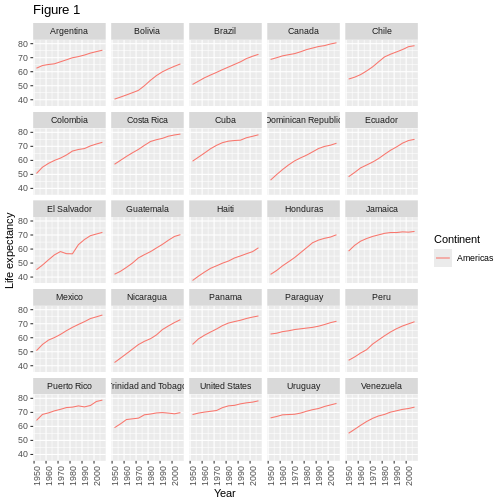
プロットのエクスポート
ggsave() 関数を使用すると、ggplot
で作成したプロットをエクスポートできます。
width、height、および dpi
引数を調整することで、出版用の高品質グラフィックを作成できます。
上記のプロットを保存するには、まずそれを lifeExp_plot
という変数に割り当て、その後 ggsave を使用してプロットを
png 形式で results ディレクトリに保存します。
(作業ディレクトリ内に results/
フォルダを作成してください。)
R
lifeExp_plot <- ggplot(data = americas, mapping = aes(x = year, y = lifeExp, color=continent)) +
geom_line() + facet_wrap( ~ country) +
labs(
x = "Year", # X軸のタイトル
y = "Life expectancy", # Y軸のタイトル
title = "Figure 1", # 図のメインタイトル
color = "Continent" # 凡例のタイトル
) +
theme(axis.text.x = element_text(angle = 90, hjust = 1))
ggsave(filename = "results/lifeExp.png", plot = lifeExp_plot, width = 12, height = 10, dpi = 300, units = "cm")
ggsave の便利な点は2つあります。
1つ目は、最後に作成したプロットをデフォルトとして保存することです。そのため、plot
引数を省略すると、自動的に最後に作成したプロットが保存されます。
2つ目は、指定したファイル名の拡張子(例:.png や
.pdf)から保存形式を自動的に判断することです。
必要に応じて、device
引数で形式を明示的に指定することもできます。
これは ggplot2 でできることの一部です。RStudio は利用可能なさまざまなレイヤーの概要を示した非常に便利なチートシートを提供しています。 また、ggplot2 のウェブサイト にはより詳細なドキュメントがあります。 すべての RStudio チートシートはRStudio のウェブサイトから利用できます。 最後に、変更方法がわからない場合、Google 検索を使用すると、Stack Overflow 上の関連する質問と再利用可能なコードが見つかることがよくあります。
チャレンジ 5
異なる大陸間で利用可能な年における平均寿命を比較するボックスプロットを生成してください。
応用:
- Y軸の名前を「Life Expectancy」に変更する。
- X軸のラベルを削除する。
次のような解答例があります: xlab() および
ylab() はそれぞれ X軸と Y軸のラベルを設定します。
軸タイトル、テキスト、目盛りは theme()
呼び出し内で変更する必要があります。
R
ggplot(data = gapminder, mapping = aes(x = continent, y = lifeExp, fill = continent)) +
geom_boxplot() + facet_wrap(~year) +
ylab("Life Expectancy") +
theme(axis.title.x=element_blank(),
axis.text.x = element_blank(),
axis.ticks.x = element_blank())
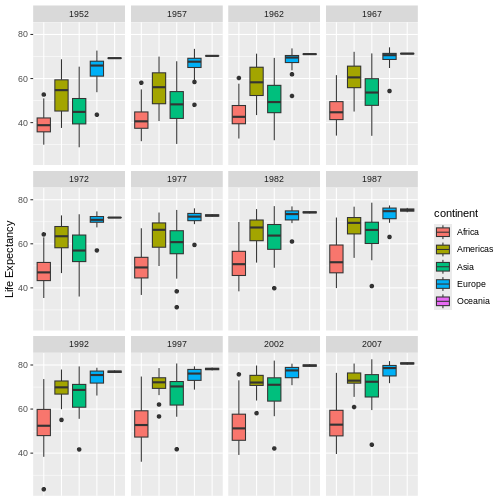
まとめ
-
ggplot2を使用してプロットを作成する。 - グラフィックをレイヤーとして考える: 美的要素、幾何学、統計、スケール変換、グループ化。
Content from ベクトル化
最終更新日:2024-11-22 | ページの編集
概要
質問
- ベクトルのすべての要素に一度に操作を行うにはどうすればよいですか?
目的
- Rにおけるベクトル化された操作を理解する。
Rの関数のほとんどはベクトル化されており、ループを使用して各要素に対して1つずつ処理を行う必要なく、ベクトルのすべての要素に対して操作を実行します。 これにより、コードが簡潔で読みやすく、エラーが少なくなります。
R
x <- 1:4
x * 2
出力
[1] 2 4 6 8乗算がベクトルの各要素に対して行われました。
2つのベクトルを加えることもできます:
R
y <- 6:9
x + y
出力
[1] 7 9 11 13x の各要素が対応する y
の要素と加算されました:
次に、ループを使用して2つのベクトルを加算する方法を示します:
R
output_vector <- c()
for (i in 1:4) {
output_vector[i] <- x[i] + y[i]
}
output_vector
出力
[1] 7 9 11 13ベクトル化された操作を使用した出力と比較してください。
R
sum_xy <- x + y
sum_xy
出力
[1] 7 9 11 13チャレンジ 1
gapminder データセットの pop
列を使用してみましょう。
gapminder データフレームに新しい列を作成し、
人口を100万人単位で表示してください。
データフレームの先頭または末尾を確認して、正しく動作したか確認してください。
gapminder データセットの pop
列を使用してみましょう。
gapminder データフレームに新しい列を作成し、
人口を100万人単位で表示してください。
データフレームの先頭または末尾を確認して、正しく動作したか確認してください。
R
gapminder$pop_millions <- gapminder$pop / 1e6
head(gapminder)
出力
country year pop continent lifeExp gdpPercap pop_millions
1 Afghanistan 1952 8425333 Asia 28.801 779.4453 8.425333
2 Afghanistan 1957 9240934 Asia 30.332 820.8530 9.240934
3 Afghanistan 1962 10267083 Asia 31.997 853.1007 10.267083
4 Afghanistan 1967 11537966 Asia 34.020 836.1971 11.537966
5 Afghanistan 1972 13079460 Asia 36.088 739.9811 13.079460
6 Afghanistan 1977 14880372 Asia 38.438 786.1134 14.880372チャレンジ 2
単一のグラフ上で、すべての国の人口(単位は100万人)を年に対してプロットしてください。 どの国がどれかを識別する必要はありません。
次に、中国、インド、インドネシアに対してのみグラフ化を行ってください。 こちらもどの国がどれかを識別する必要はありません。
人口(単位は100万人)を年に対してプロットして、プロットスキルをリフレッシュしましょう。
R
ggplot(gapminder, aes(x = year, y = pop_millions)) +
geom_point()
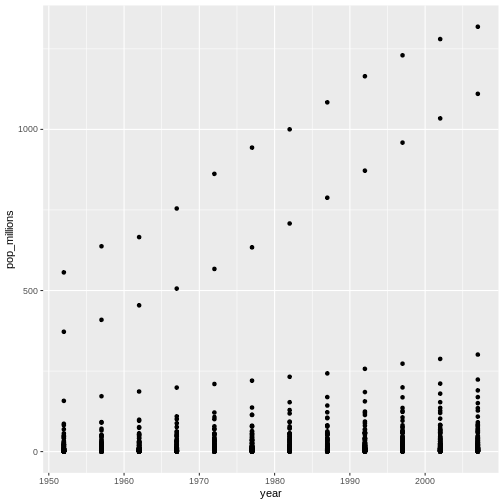
R
countryset <- c("China","India","Indonesia")
ggplot(gapminder[gapminder$country %in% countryset,],
aes(x = year, y = pop_millions)) +
geom_point()
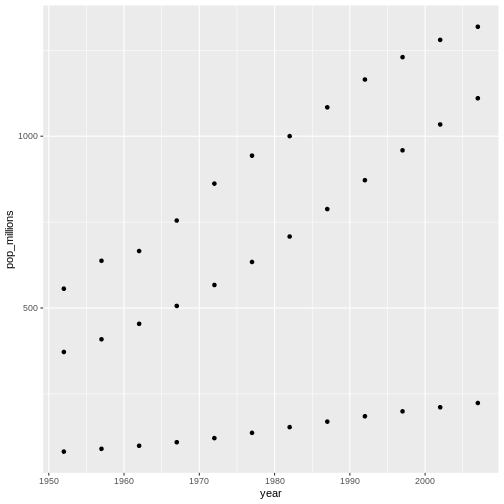
比較演算子、論理演算子、多くの関数もまたベクトル化されています:
比較演算子
R
x > 2
出力
[1] FALSE FALSE TRUE TRUE論理演算子
R
a <- x > 3 # または、明確にするために a <- (x > 3)
a
出力
[1] FALSE FALSE FALSE TRUEヒント:論理ベクトルの便利な関数
any() はベクトル内に1つでも TRUE
が含まれていれば TRUE を返します。all() はベクトル内のすべての要素が TRUE
の場合にのみ TRUE を返します。
ほとんどの関数はベクトルに対して要素ごとに操作を行います:
関数
R
x <- 1:4
log(x)
出力
[1] 0.0000000 0.6931472 1.0986123 1.3862944ベクトル化された操作は行列の要素ごとにも動作します:
R
m <- matrix(1:12, nrow=3, ncol=4)
m * -1
出力
[,1] [,2] [,3] [,4]
[1,] -1 -4 -7 -10
[2,] -2 -5 -8 -11
[3,] -3 -6 -9 -12ヒント:要素ごとの積と行列積の違い
重要:* 演算子は要素ごとの積を行います!
行列積を行うには、%*% 演算子を使用する必要があります:
R
m %*% matrix(1, nrow=4, ncol=1)
出力
[,1]
[1,] 22
[2,] 26
[3,] 30R
matrix(1:4, nrow=1) %*% matrix(1:4, ncol=1)
出力
[,1]
[1,] 30行列代数についての詳細は、Quick-R のリファレンスガイド を参照してください。
チャレンジ 3
次の行列を使用して:
R
m <- matrix(1:12, nrow=3, ncol=4)
m
出力
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12以下を実行するとどうなるか予想してください:
m ^ -1m * c(1, 0, -1)m > c(0, 20)m * c(1, 0, -1, 2)
出力が予想と違った場合は、ヘルパーに尋ねてください!
次の行列を使用して:
R
m <- matrix(1:12, nrow=3, ncol=4)
m
出力
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 2 5 8 11
[3,] 3 6 9 12以下を実行するとどうなるか予想してください:
m ^ -1
出力
[,1] [,2] [,3] [,4]
[1,] 1.0000000 0.2500000 0.1428571 0.10000000
[2,] 0.5000000 0.2000000 0.1250000 0.09090909
[3,] 0.3333333 0.1666667 0.1111111 0.08333333m * c(1, 0, -1)
出力
[,1] [,2] [,3] [,4]
[1,] 1 4 7 10
[2,] 0 0 0 0
[3,] -3 -6 -9 -12m > c(0, 20)
出力
[,1] [,2] [,3] [,4]
[1,] TRUE FALSE TRUE FALSE
[2,] FALSE TRUE FALSE TRUE
[3,] TRUE FALSE TRUE FALSEチャレンジ 4
次の分数列の合計を計算したいとします:
R
x = 1/(1^2) + 1/(2^2) + 1/(3^2) + ... + 1/(n^2)
これを手で書き出すのは面倒で、高い値の n に対しては不可能です。 ベクトル化を使用して、n=100 の場合の x を計算してください。 n=10,000 の場合の合計はどうなりますか?
次の分数列の合計を計算したいとします:
R
x = 1/(1^2) + 1/(2^2) + 1/(3^2) + ... + 1/(n^2)
これを手で書き出すのは面倒で、高い値の n に対しては不可能です。 ベクトル化を使用して、n=100 の場合の x を計算してください。 n=10,000 の場合はどうでしょうか?
R
sum(1/(1:100)^2)
出力
[1] 1.634984R
sum(1/(1:1e04)^2)
出力
[1] 1.644834R
n <- 10000
sum(1/(1:n)^2)
出力
[1] 1.644834同じ結果を関数を使用して得ることもできます:
R
inverse_sum_of_squares <- function(n) {
sum(1/(1:n)^2)
}
inverse_sum_of_squares(100)
出力
[1] 1.634984R
inverse_sum_of_squares(10000)
出力
[1] 1.644834R
n <- 10000
inverse_sum_of_squares(n)
出力
[1] 1.644834ヒント:長さが異なるベクトルに対する操作
長さが異なるベクトルに対しても操作を実行できます。 これはリサイクルと呼ばれるプロセスを通じて行われます。 このプロセスでは、小さいベクトルが自動的に繰り返されて大きいベクトルの長さに一致します。 大きいベクトルが小さいベクトルの倍数ではない場合、Rは警告を出します。
R
x <- c(1, 2, 3)
y <- c(1, 2, 3, 4, 5, 6, 7)
x + y
警告
Warning in x + y: longer object length is not a multiple of shorter object
length出力
[1] 2 4 6 5 7 9 8ベクトル x はベクトル y
の長さに一致するようにリサイクルされました:
まとめ
- ループの代わりにベクトル化された操作を使用する。
Content from 関数の説明
最終更新日:2024-11-22 | ページの編集
概要
質問
- Rで新しい関数を書くにはどうすればよいですか?
目的
- 引数を受け取る関数を定義する。
- 関数から値を返す。
- 関数内で
stopifnot()を使って引数の条件を確認する。 - 関数をテストする。
- 関数引数のデフォルト値を設定する。
- プログラムを小さく、単一目的の関数に分割する理由を説明する。
1つのデータセットだけを分析するのであれば、スプレッドシートにデータをロードして単純な統計をプロットする方が速いかもしれません。 しかし、gapminder データは定期的に更新されるため、将来的に新しい情報を取得して分析を再実行する必要があるかもしれません。 また、別のソースから類似のデータを取得することも考えられます。
このレッスンでは、関数の書き方を学び、複数の操作を1つのコマンドで繰り返せるようにします。
関数とは?
関数は一連の操作をまとめ、継続的に使用できるようにします。 関数には以下の利点があります:
- 覚えやすい名前で呼び出すことができる。
- 個々の操作を覚える必要がなくなる。
- 定義された入力と期待される出力がある。
- プログラミング環境全体との豊かな接続が得られる。
ほとんどのプログラミング言語の基本的な構成要素として、ユーザー定義関数は「プログラミング」を代表する抽象概念と言えます。 関数を書いたことがあれば、あなたはコンピュータープログラマーです。
関数の定義
functions/ ディレクトリ内に新しい R
スクリプトファイルを開き、functions-lesson.R
という名前を付けます。
関数の一般的な構造は次のようになります:
R
my_function <- function(parameters) {
# 操作を実行
# 値を返す
}
Fahrenheit から Kelvin に温度を変換する関数
fahr_to_kelvin() を定義してみましょう:
R
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
fahr_to_kelvin() を function
の出力として割り当てることで定義します。
引数名のリストは括弧内に含まれています。 次に、関数の本体である実行される文を波括弧
({}) 内に記述します。
本体内の文はインデント(スペース2つ)されています。これによりコードが読みやすくなりますが、コードの動作には影響しません。
関数を作成することはレシピを書くことに似ています。 最初に関数が必要とする「材料」を定義します。この場合、材料は「temp」の1つだけです。 次に、材料をどのように処理するかを記述します。この例では、材料に数値操作を適用しています。
関数を呼び出すとき、引数として渡された値は関数内で使用できる変数に割り当てられます。 関数内ではreturn文を使用して、呼び出し元に結果を返します。
ヒント
Rの特有の機能として、return文は必須ではありません。 Rは関数本体の最後の行にある変数を自動的に返します。 ただし、明示的に return 文を定義することでコードがより明確になります。
関数を実行してみましょう。 自分で定義した関数を呼び出すのは、他の関数を呼び出すのと同じです:
R
# 水の凝固点
fahr_to_kelvin(32)
出力
[1] 273.15R
# 水の沸点
fahr_to_kelvin(212)
出力
[1] 373.15チャレンジ 1
Kelvin の温度を受け取り、Celsius に変換して返す関数
kelvin_to_celsius() を作成してください。
ヒント: Kelvin を Celsius に変換するには、273.15 を引きます。
Kelvin の温度を受け取り、Celsius に変換して返す関数
kelvin_to_celsius を作成します:
R
kelvin_to_celsius <- function(temp) {
celsius <- temp - 273.15
return(celsius)
}
関数の組み合わせ
関数の本当の力は、目的に応じて組み合わせたり再利用したりすることで得られます。
Fahrenheit を Kelvin に、Kelvin を Celsius に変換する2つの関数を定義してみましょう:
R
fahr_to_kelvin <- function(temp) {
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
kelvin_to_celsius <- function(temp) {
celsius <- temp - 273.15
return(celsius)
}
チャレンジ 2
上記の2つの関数を再利用して、Fahrenheit を直接 Celsius に変換する関数を定義してください。
Fahrenheit を直接 Celsius に変換する関数を定義します:
R
fahr_to_celsius <- function(temp) {
temp_k <- fahr_to_kelvin(temp)
result <- kelvin_to_celsius(temp_k)
return(result)
}
インタールード: 防御的プログラミング
関数を書くことで R コードを再利用可能かつモジュール化できる効率的な方法を理解したところで、関数が意図した用途でのみ動作するようにすることが重要です。 引数の条件を確認することは、防御的プログラミングの概念に関連しています。 防御的プログラミングでは、頻繁に条件を確認し、何か問題があればエラーをスローします。
stopifnot() を使った条件の確認
例えば、fahr_to_kelvin()
を以下のように改良できます:
R
fahr_to_kelvin <- function(temp) {
stopifnot(is.numeric(temp))
kelvin <- ((temp - 32) * (5 / 9)) + 273.15
return(kelvin)
}
適切な入力では正常に動作しますが、不適切な入力では即座にエラーをスローします。
チャレンジ 3
fahr_to_celsius()
関数で、防御的プログラミングを使用して、引数 temp
が不適切に指定された場合にエラーをスローするようにしてください。
stopifnot()
を追加して、引数を明示的に確認するようにします:
R
fahr_to_celsius <- function(temp) {
stopifnot(is.numeric(temp))
temp_k <- fahr_to_kelvin(temp)
result <- kelvin_to_celsius(temp_k)
return(result)
}
関数の組み合わせについてさらに詳しく
次に、データセットから国の国内総生産(GDP)を計算する関数を定義します:
R
# データセットを受け取り、人口列と
# 1人当たりGDP列を掛け合わせます。
calcGDP <- function(dat) {
gdp <- dat$pop * dat$gdpPercap
return(gdp)
}
calcGDP() は function
の出力として定義されます。 引数名のリストは括弧内に含まれています。
次に、関数を呼び出したときに実行される文(関数の本体)は波括弧
({}) 内に記述されます。
本体内の文をインデント(スペース2つ)しました。これにより、コードが読みやすくなりますが、動作には影響しません。
関数を呼び出すと、渡された値が引数に割り当てられ、それが関数の本体内の変数になります。
関数内では、return() 関数を使用して結果を返します。
ただし、return()
関数は任意です。Rは関数本体の最後の行にあるコマンドの結果を自動的に返します。
R
calcGDP(head(gapminder))
出力
[1] 6567086330 7585448670 8758855797 9648014150 9678553274 11697659231あまり情報量が多くありませんね。では、引数を追加して、年や国ごとにデータを抽出できるようにしましょう。
R
# データセットを受け取り、人口列と
# 1人当たりGDP列を掛け合わせます。
calcGDP <- function(dat, year=NULL, country=NULL) {
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}
gdp <- dat$pop * dat$gdpPercap
new <- cbind(dat, gdp=gdp)
return(new)
}
これらの関数を別の R
スクリプトに書き出している場合(おすすめです!)、source()
関数を使用してRセッションに読み込むことができます:
R
source("functions/functions-lesson.R")
では、この関数で行われる処理について簡単に説明します。 英語でいうと、次のような流れです:
- 提供されたデータを年で絞り込みます(
year引数が空でない場合)。 - その結果を国でさらに絞り込みます(
country引数が空でない場合)。 - 上記の2つの手順から得られたサブセットに対してGDPを計算します。
- サブセットにGDP列を新しい列として追加し、これを最終結果として返します。
この出力は、単なる数値ベクトルよりもはるかに有用です。
年を指定するとどうなるか見てみましょう:
R
head(calcGDP(gapminder, year=2007))
出力
country year pop continent lifeExp gdpPercap gdp
12 Afghanistan 2007 31889923 Asia 43.828 974.5803 31079291949
24 Albania 2007 3600523 Europe 76.423 5937.0295 21376411360
36 Algeria 2007 33333216 Africa 72.301 6223.3675 207444851958
48 Angola 2007 12420476 Africa 42.731 4797.2313 59583895818
60 Argentina 2007 40301927 Americas 75.320 12779.3796 515033625357
72 Australia 2007 20434176 Oceania 81.235 34435.3674 703658358894特定の国の場合:
R
calcGDP(gapminder, country="Australia")
出力
country year pop continent lifeExp gdpPercap gdp
61 Australia 1952 8691212 Oceania 69.120 10039.60 87256254102
62 Australia 1957 9712569 Oceania 70.330 10949.65 106349227169
63 Australia 1962 10794968 Oceania 70.930 12217.23 131884573002
64 Australia 1967 11872264 Oceania 71.100 14526.12 172457986742
65 Australia 1972 13177000 Oceania 71.930 16788.63 221223770658
66 Australia 1977 14074100 Oceania 73.490 18334.20 258037329175
67 Australia 1982 15184200 Oceania 74.740 19477.01 295742804309
68 Australia 1987 16257249 Oceania 76.320 21888.89 355853119294
69 Australia 1992 17481977 Oceania 77.560 23424.77 409511234952
70 Australia 1997 18565243 Oceania 78.830 26997.94 501223252921
71 Australia 2002 19546792 Oceania 80.370 30687.75 599847158654
72 Australia 2007 20434176 Oceania 81.235 34435.37 703658358894または、両方を指定する場合:
R
calcGDP(gapminder, year=2007, country="Australia")
出力
country year pop continent lifeExp gdpPercap gdp
72 Australia 2007 20434176 Oceania 81.235 34435.37 703658358894関数の本体を詳しく見てみましょう:
ここでは、year と country
の2つの引数を追加しました。 これらにはデフォルト引数として
NULL
を設定しています。これは、ユーザーが別の値を指定しない限り、引数がこの値を取ることを意味します。
R
if(!is.null(year)) {
dat <- dat[dat$year %in% year, ]
}
if (!is.null(country)) {
dat <- dat[dat$country %in% country,]
}
ここでは、各追加引数が NULL
かどうかを確認し、NULL でない場合は、dat
に格納されているデータセットを該当する引数に基づいて絞り込みます。
条件を組み込むことで、関数を柔軟に使用できるようにしました。 これで以下の用途に対応できます:
- データセット全体の計算
- 単一の年の計算
- 単一の国の計算
- 年と国の組み合わせの計算
さらに %in%
を使用することで、複数の年や国を引数に渡すこともできます。
ヒント:値渡し (Pass by value)
R の関数はほぼ常に、関数本体内で操作するためにデータをコピーします。
関数内で dat を変更しても、それは関数に渡されたオリジナルの
gapminder データセットではなく、コピーされた
dat に対してのみ適用されます。
これは「値渡し」と呼ばれ、コードを書く際に安全性が高まります。 関数本体内で行った変更は、本体内にとどまることが保証されます。
ヒント:関数スコープ (Function scope)
もう1つの重要な概念はスコープです。
関数本体内で作成または変更された変数(または関数)は、その関数の実行中にのみ存在します。
たとえば、calcGDP() を呼び出すと、変数
dat、gdp、new
は関数本体内にのみ存在します。
同じ名前の変数がインタラクティブRセッション内にあっても、それらは実行中の関数に影響を受けません。
最後に、新しいサブセットに基づいてGDPを計算し、その列を追加した新しいデータフレームを作成しました。 このようにすると、関数を後で呼び出したときに、返されたGDP値の文脈を確認でき、最初の試行で得られた数値ベクトルよりもはるかに有用です。
チャレンジ 4
1987年のニュージーランドのGDPを計算してみましょう。 1952年のニュージーランドのGDPとどう違いますか?
R
calcGDP(gapminder, year = c(1952, 1987), country = "New Zealand")
1987年のニュージーランドのGDP: 65,050,008,703
1952年のニュージーランドのGDP: 21,058,193,787
チャレンジ 5
paste()
関数は、テキストを結合するために使用できます。例:
R
best_practice <- c("Write", "programs", "for", "people", "not", "computers")
paste(best_practice, collapse=" ")
出力
[1] "Write programs for people not computers"2つの引数 text と wrapper を取る関数
fence() を作成し、text を wrapper
で囲んだテキストを出力してください:
R
fence(text=best_practice, wrapper="***")
注: paste() 関数には sep
という引数があり、テキスト間の区切り文字を指定します。
デフォルトはスペース " " です。paste0()
のデフォルトはスペースなしの "" です。
2つの引数 text と wrapper を取る関数
fence() を作成し、text を wrapper
で囲んだテキストを出力します:
R
fence <- function(text, wrapper){
text <- c(wrapper, text, wrapper)
result <- paste(text, collapse = " ")
return(result)
}
best_practice <- c("Write", "programs", "for", "people", "not", "computers")
fence(text=best_practice, wrapper="***")
出力
[1] "*** Write programs for people not computers ***"ヒント
Rには、複雑な操作を行う際に活用できる独自の機能があります。 ここでは、これらの高度な概念を必要とする内容を書くことはありません。 将来的にRで関数を書くことに慣れたら、R言語マニュアル や Hadley Wickham の Advanced R Programming にあるこの章を読んで学びを深めてください。
ヒント:テストとドキュメント
関数をテストしてドキュメント化することは非常に重要です: ドキュメントは、関数の目的、使い方を理解する助けとなり、関数が意図した通りに動作するか確認することも重要です。
初めのうちは、ワークフローは以下のようになるでしょう:
- 関数を書く
- 関数の動作をドキュメント化するためにコメントを追加する
- ソースファイルを読み込む
- コンソールで関数を試して期待通りに動作するか確認する
- 必要なバグ修正を行う
- 繰り返す
関数の正式なドキュメントは、.Rd
ファイルに記述され、ヘルプファイルで見ることができるドキュメントに変換されます。
roxygen2
パッケージを使用すると、関数コードに沿ってドキュメントを記述し、適切な
.Rd ファイルに処理できます。
より複雑なRプロジェクトを書くようになったら、この形式的なドキュメント作成方法に切り替えることを検討してください。
実際、パッケージとは、形式的なドキュメント付きの関数の集合です。
自分の関数を source("functions.R")
を使用して読み込むのは、他人の関数(または将来的には自分のパッケージ!)を
library("package")
を通じて読み込むのと本質的に同じです。
正式な自動化テストは、testthat パッケージを使用して記述できます。
まとめ
- Rで新しい関数を定義するには
functionを使用する。 - 関数に値を渡すにはパラメータを使用する。
- Rで
stopifnot()を使って関数引数を柔軟にチェックする。 -
source()を使用してプログラムに関数を読み込む。
Content from データの書き出し
最終更新日:2024-11-22 | ページの編集
概要
質問
- Rで作成したプロットやデータをどのように保存できますか?
目的
- Rからプロットやデータを保存できるようになる。
プロットの保存
すでに ggplot2 で作成した最新のプロットを
ggsave コマンドを使って保存する方法を学びました。
復習として:
R
ggsave("My_most_recent_plot.pdf")
RStudio の「Plot」ウィンドウで「Export」ボタンを使用してプロットを保存することもできます。 これにより、.pdf や .png、.jpg などの形式で保存するオプションが表示されます。
プロットを事前に「Plot」ウィンドウに作成せずに保存したい場合もあります。 たとえば、複数ページのPDFドキュメントを作成し、それぞれ異なるプロットを表示したい場合や、 ループでファイルの複数のサブセットを処理して、各サブセットからデータをプロットし、それぞれのプロットを保存したい場合があります。
この場合、より柔軟なアプローチを使用できます。 pdf
関数を使用して新しいPDFデバイスを作成できます。この関数の引数を使用してサイズや解像度を制御できます。
R
pdf("Life_Exp_vs_time.pdf", width=12, height=4)
ggplot(data=gapminder, aes(x=year, y=lifeExp, colour=country)) +
geom_line() +
theme(legend.position = "none")
# PDFデバイスをオフにすることを忘れないでください!
dev.off()
このドキュメントを開いて確認してみてください。
チャレンジ 1
‘pdf’ コマンドを変更して、PDFの2ページ目を作成し、 同じデータを使って
facet_grid
を使用して大陸ごとに1つのパネルを表示するプロットを追加してください。
R
pdf("Life_Exp_vs_time.pdf", width = 12, height = 4)
p <- ggplot(data = gapminder, aes(x = year, y = lifeExp, colour = country)) +
geom_line() +
theme(legend.position = "none")
p
p + facet_grid(~continent)
dev.off()
jpeg や png
などのコマンドも同様に使用して、異なる形式のドキュメントを作成できます。
データの書き出し
Rからデータを書き出したい場合があります。
write.table 関数を使用してこれを行うことができます。
この関数は以前学んだ read.table に非常に似ています。
データクリーニングスクリプトを作成しましょう。 この分析では、オーストラリアの gapminder データに焦点を当てます:
R
aust_subset <- gapminder[gapminder$country == "Australia",]
write.table(aust_subset,
file="cleaned-data/gapminder-aus.csv",
sep=","
)
シェルに戻り、データが正しく表示されるか確認しましょう:
出力
"country","year","pop","continent","lifeExp","gdpPercap"
"61","Australia",1952,8691212,"Oceania",69.12,10039.59564
"62","Australia",1957,9712569,"Oceania",70.33,10949.64959
"63","Australia",1962,10794968,"Oceania",70.93,12217.22686
"64","Australia",1967,11872264,"Oceania",71.1,14526.12465
"65","Australia",1972,13177000,"Oceania",71.93,16788.62948
"66","Australia",1977,14074100,"Oceania",73.49,18334.19751
"67","Australia",1982,15184200,"Oceania",74.74,19477.00928
"68","Australia",1987,16257249,"Oceania",76.32,21888.88903
"69","Australia",1992,17481977,"Oceania",77.56,23424.76683あれ、ちょっと想定と違いますね。なぜか引用符がついています。 また、行番号が意味を持たないようです。
ヘルプファイルを見て、この挙動を変更する方法を確認してみましょう。
R
?write.table
デフォルトでは、Rは文字ベクトルをファイルに書き出す際に引用符で囲みます。 また、行と列の名前も書き出します。
これを修正しましょう:
R
write.table(
gapminder[gapminder$country == "Australia",],
file="cleaned-data/gapminder-aus.csv",
sep=",", quote=FALSE, row.names=FALSE
)
もう一度シェルスキルを使ってデータを確認しましょう:
出力
country,year,pop,continent,lifeExp,gdpPercap
Australia,1952,8691212,Oceania,69.12,10039.59564
Australia,1957,9712569,Oceania,70.33,10949.64959
Australia,1962,10794968,Oceania,70.93,12217.22686
Australia,1967,11872264,Oceania,71.1,14526.12465
Australia,1972,13177000,Oceania,71.93,16788.62948
Australia,1977,14074100,Oceania,73.49,18334.19751
Australia,1982,15184200,Oceania,74.74,19477.00928
Australia,1987,16257249,Oceania,76.32,21888.88903
Australia,1992,17481977,Oceania,77.56,23424.76683これでよさそうです!
チャレンジ 2
gapminder データを1990年以降に収集されたデータポイントのみを含むようにサブセット化する データクリーニングスクリプトファイルを作成してください。
このスクリプトを使用して、新しいサブセットを
cleaned-data/ ディレクトリに保存してください。
R
write.table(
gapminder[gapminder$year > 1990, ],
file = "cleaned-data/gapminder-after1990.csv",
sep = ",", quote = FALSE, row.names = FALSE
)
まとめ
- RStudioでプロットを保存するには「Export」ボタンを使用する。
- タブ形式のデータを保存するには
write.tableを使用する。
Content from dplyr を使用したデータフレーム操作
最終更新日:2024-11-22 | ページの編集
概要
質問
- 自分を繰り返さずにデータフレームを操作するにはどうすればよいですか?
目的
-
dplyrの6つの主要なデータフレーム操作の「動詞」をパイプと共に使用できるようになる。 -
group_by()とsummarize()を組み合わせてデータセットを要約する方法を理解する。 - 論理フィルタリングを使用してデータのサブセットを分析できるようになる。
データフレームの操作とは、研究者にとって多くの意味を持ちます。 たとえば、特定の観測値(行)や変数(列)を選択したり、データを特定の変数でグループ化したり、要約統計量を計算したりします。 これらの操作は通常の R の操作で実行できます:
R
mean(gapminder$gdpPercap[gapminder$continent == "Africa"])
出力
[1] 2193.755R
mean(gapminder$gdpPercap[gapminder$continent == "Americas"])
出力
[1] 7136.11R
mean(gapminder$gdpPercap[gapminder$continent == "Asia"])
出力
[1] 7902.15しかし、この方法はあまり「効率的」ではありません。 繰り返し作業は、現在および将来の時間を浪費し、重大なバグを引き起こす可能性があります。
dplyr パッケージ
幸いなことに、dplyr
パッケージは、上記の繰り返しを減らし、エラーの確率を減らし、入力作業を削減する非常に便利な関数を提供します。
さらに、dplyr の文法は読みやすくなる場合もあります。
ヒント:Tidyverse
dplyr
パッケージは、データサイエンス用に設計された「Tidyverse」と呼ばれる一連の
R パッケージの一部です。
これらのパッケージは、互いに調和して動作するように特別に設計されています。
このコースではいくつかのパッケージについて説明しますが、詳細は次のリンクを参照してください:
https://www.tidyverse.org/
ここでは、最もよく使用される5つの関数と、パイプ (%>%)
を使用した組み合わせを紹介します。
select()filter()group_by()summarize()mutate()
まだこのパッケージをインストールしていない場合は、以下を実行してください:
R
install.packages('dplyr')
次に、パッケージをロードします:
R
library("dplyr")
select() の使用
たとえば、データフレームのいくつかの変数のみを使用したい場合は、select()
関数を使用できます。 これにより、選択した変数のみが保持されます。
R
year_country_gdp <- select(gapminder, year, country, gdpPercap)
gapminder データから continent
列を削除したい場合は次のようにします:
R
smaller_gapminder_data <- select(gapminder, -continent)
year_country_gdp を開くと、年、国、および gdpPercap
のみが含まれていることがわかります。
上記では通常の文法を使用しましたが、dplyr
の強みはパイプを使用して複数の関数を組み合わせることにあります。 R
では初めてのパイプ文法なので、上記の例をパイプを使って書き直してみます。
R
year_country_gdp <- gapminder %>% select(year, country, gdpPercap)
この書き方の理由を理解するために、ステップごとに説明します。
まず、gapminder データフレームを呼び出し、パイプ記号 %>%
を使用して次のステップである select() 関数に渡します。
この場合、select()
関数でどのデータオブジェクトを使用するか指定しません。前のパイプからそれを取得するためです。
豆知識:シェルでパイプに出会ったことがある可能性があります。
R ではパイプ記号は %>% ですが、シェルでは |
です。概念は同じです!
ヒント:dplyr を使った列名の変更
第4章では、names()
関数の出力に値を割り当てることで列名を変更する方法を学びました。
これは少し面倒ですが、幸いなことに dplyr には rename()
関数があります。
パイプライン内での構文は rename(new_name = old_name)
です。 たとえば、上記の select() 文から gdpPercap
列名を変更してみましょう。
R
tidy_gdp <- year_country_gdp %>% rename(gdp_per_capita = gdpPercap)
head(tidy_gdp)
出力
year country gdp_per_capita
1 1952 Afghanistan 779.4453
2 1957 Afghanistan 820.8530
3 1962 Afghanistan 853.1007
4 1967 Afghanistan 836.1971
5 1972 Afghanistan 739.9811
6 1977 Afghanistan 786.1134
filter() の使用
上記を進めて、ヨーロッパの国のみを対象にしたい場合、select
と filter を組み合わせることができます:
R
year_country_gdp_euro <- gapminder %>%
filter(continent == "Europe") %>%
select(year, country, gdpPercap)
2007年のヨーロッパの国の平均寿命だけを表示したい場合は次のようにします:
R
europe_lifeExp_2007 <- gapminder %>%
filter(continent == "Europe", year == 2007) %>%
select(country, lifeExp)
チャレンジ 1
パイプを含む複数行にわたる単一のコマンドを書いて、lifeExp、country、year
のアフリカの値だけを含むデータフレームを生成してください。
他の大陸のデータは含まないようにしてください。
このデータフレームには何行ありますか?また、その理由は?
R
year_country_lifeExp_Africa <- gapminder %>%
filter(continent == "Africa") %>%
select(year, country, lifeExp)
上記と同様に、最初に gapminder データフレームを filter()
関数に渡し、その後 filter()
でフィルタリングされたバージョンの gapminder データフレームを
select() 関数に渡します。 注意:
この場合、操作の順序が非常に重要です。 最初に select
を使用した場合、フィルタリングステップの前に continent
変数を削除してしまうため、filter は動作しなくなります。
group_by() の使用
ベース R
を使ったエラーの起きやすい繰り返しを減らすつもりでしたが、これまではまだ同じことをすべての大陸について繰り返す必要がありました。
filter() を使用すると、条件に一致する観測値(上記では
continent=="Europe")のみが通過しますが、代わりに
group_by()
を使用すると、フィルタリングに使える一意の条件ごとにグループ化できます。
R
str(gapminder)
出力
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...R
str(gapminder %>% group_by(continent))
出力
gropd_df [1,704 × 6] (S3: grouped_df/tbl_df/tbl/data.frame)
$ country : chr [1:1704] "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int [1:1704] 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num [1:1704] 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr [1:1704] "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num [1:1704] 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num [1:1704] 779 821 853 836 740 ...
- attr(*, "groups")= tibble [5 × 2] (S3: tbl_df/tbl/data.frame)
..$ continent: chr [1:5] "Africa" "Americas" "Asia" "Europe" ...
..$ .rows : list<int> [1:5]
.. ..$ : int [1:624] 25 26 27 28 29 30 31 32 33 34 ...
.. ..$ : int [1:300] 49 50 51 52 53 54 55 56 57 58 ...
.. ..$ : int [1:396] 1 2 3 4 5 6 7 8 9 10 ...
.. ..$ : int [1:360] 13 14 15 16 17 18 19 20 21 22 ...
.. ..$ : int [1:24] 61 62 63 64 65 66 67 68 69 70 ...
.. ..@ ptype: int(0)
..- attr(*, ".drop")= logi TRUEgroup_by() を使用したデータフレーム(`group
ed_df)の構造は、元のgapminder(data.frame)とは異なることに気づくでしょう。grouped_dfは、各リスト項目が特定のcontinentの値に対応する行のみを含むdata.frame`
で構成されたリストのように考えることができます。
## `summarize()` の使用
上記では少し退屈でしたが、`group_by()` を `summarize()` と組み合わせると非常に強力になります。
これにより、各大陸ごとのデータフレームに対して繰り返し処理を行う関数を使用して、新しい変数を作成できます。
つまり、`group_by()` を使用して元のデータフレームを複数の部分に分割し、その後、`summarize()` 内で関数(例:`mean()` や `sd()`)を実行できます。
``` r
gdp_bycontinents <- gapminder %>%
group_by(continent) %>%
summarize(mean_gdpPercap = mean(gdpPercap))
R
continent mean_gdpPercap
<fctr> <dbl>
1 Africa 2193.755
2 Americas 7136.110
3 Asia 7902.150
4 Europe 14469.476
5 Oceania 18621.609これにより、大陸ごとの gdpPercap の平均値を計算できましたが、さらに便利なことに発展します。
チャレンジ 2
各国の平均寿命を計算してください。平均寿命が最も長い国と最も短い国はどこですか?
R
lifeExp_bycountry <- gapminder %>%
group_by(country) %>%
summarize(mean_lifeExp = mean(lifeExp))
lifeExp_bycountry %>%
filter(mean_lifeExp == min(mean_lifeExp) | mean_lifeExp == max(mean_lifeExp))
出力
# A tibble: 2 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5
2 Sierra Leone 36.8別の方法として、dplyr の関数 arrange()
を使用して行を並べ替えることもできます。
これはデータフレームの1つ以上の変数に基づいて並べ替えを行います。
降順にソートするには arrange() 内で desc()
を使用します。
R
lifeExp_bycountry %>%
arrange(mean_lifeExp) %>%
head(1)
出力
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Sierra Leone 36.8R
lifeExp_bycountry %>%
arrange(desc(mean_lifeExp)) %>%
head(1)
出力
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Iceland 76.5アルファベット順でも並べ替え可能です:
R
lifeExp_bycountry %>%
arrange(desc(country)) %>%
head(1)
出力
# A tibble: 1 × 2
country mean_lifeExp
<chr> <dbl>
1 Zimbabwe 52.7group_by()
関数を使用すると、複数の変数でグループ化できます。たとえば、year
と continent でグループ化してみましょう。
R
gdp_bycontinents_byyear <- gapminder %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap))
出力
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.これだけでもかなり強力ですが、さらに便利なことに、summarize()
内で複数の新しい変数を定義することもできます。
R
gdp_pop_bycontinents_byyear <- gapminder %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop))
出力
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
count() と n() の使用
グループごとの観測値の数を数える操作は非常に一般的です。dplyr
パッケージにはこれを支援する2つの関連する関数があります。
たとえば、2002年のデータセットに含まれる国の数を確認したい場合、count()
関数を使用できます。
この関数は、グループ化したい列名を1つ以上指定し、sort=TRUE
を追加することで結果を降順に並べることができます:
R
gapminder %>%
filter(year == 2002) %>%
count(continent, sort = TRUE)
出力
continent n
1 Africa 52
2 Asia 33
3 Europe 30
4 Americas 25
5 Oceania 2計算に観測値の数を使用する必要がある場合、n()
関数が便利です。 これは現在のグループ内の観測値の総数を返します。
たとえば、大陸ごとの標準誤差を求めたい場合:
R
gapminder %>%
group_by(continent) %>%
summarize(se_le = sd(lifeExp)/sqrt(n()))
出力
# A tibble: 5 × 2
continent se_le
<chr> <dbl>
1 Africa 0.366
2 Americas 0.540
3 Asia 0.596
4 Europe 0.286
5 Oceania 0.775複数のサマリー操作を連鎖させることも可能です。
次の例では、各大陸の国ごとの平均寿命の
最小値、最大値、平均、および
標準誤差 を計算しています:
R
gapminder %>%
group_by(continent) %>%
summarize(
mean_le = mean(lifeExp),
min_le = min(lifeExp),
max_le = max(lifeExp),
se_le = sd(lifeExp)/sqrt(n()))
出力
# A tibble: 5 × 5
continent mean_le min_le max_le se_le
<chr> <dbl> <dbl> <dbl> <dbl>
1 Africa 48.9 23.6 76.4 0.366
2 Americas 64.7 37.6 80.7 0.540
3 Asia 60.1 28.8 82.6 0.596
4 Europe 71.9 43.6 81.8 0.286
5 Oceania 74.3 69.1 81.2 0.775
mutate() の使用
情報を要約する前(または後)に mutate()
を使用して新しい変数を作成することもできます。
R
gdp_pop_bycontinents_byyear <- gapminder %>%
mutate(gdp_billion = gdpPercap*pop/10^9) %>%
group_by(continent,year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))
出力
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
mutate と論理フィルタリングの組み合わせ:
ifelse
新しい変数を作成する際、論理条件を組み合わせることができます。
mutate() と ifelse()
を組み合わせることで、新しい変数の作成時にフィルタリングを簡単に行うことができます。
以下はその例です:
R
## 特定の条件でGDPを計算
gdp_pop_bycontinents_byyear_above25 <- gapminder %>%
mutate(gdp_billion = ifelse(lifeExp > 25, gdpPercap * pop / 10^9, NA)) %>%
group_by(continent, year) %>%
summarize(mean_gdpPercap = mean(gdpPercap),
sd_gdpPercap = sd(gdpPercap),
mean_pop = mean(pop),
sd_pop = sd(pop),
mean_gdp_billion = mean(gdp_billion),
sd_gdp_billion = sd(gdp_billion))
出力
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.
dplyr と ggplot2 の組み合わせ
最初に ggplot2 をインストールしてロードします:
R
install.packages('ggplot2')
R
library("ggplot2")
次に、%>% を使用してデータフレームを
ggplot() に直接渡す例です:
R
gapminder %>%
filter(continent == "Americas") %>%
ggplot(mapping = aes(x = year, y = lifeExp)) +
geom_line() +
facet_wrap( ~ country) +
theme(axis.text.x = element_text(angle = 45))
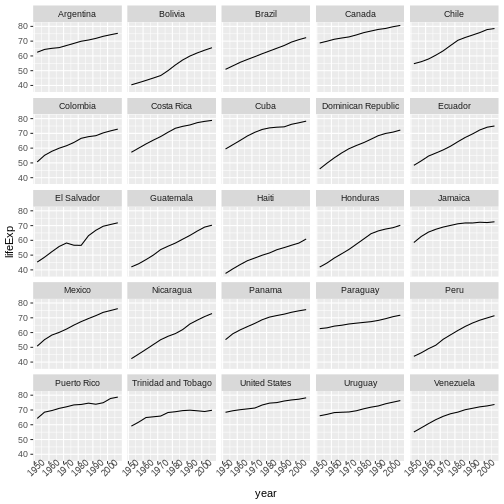
その他の例:
R
gapminder %>%
mutate(startsWith = substr(country, 1, 1)) %>%
filter(startsWith %in% c("A", "Z")) %>%
ggplot(aes(x = year, y = lifeExp, colour = continent)) +
geom_line() +
facet_wrap(vars(country)) +
theme_minimal()
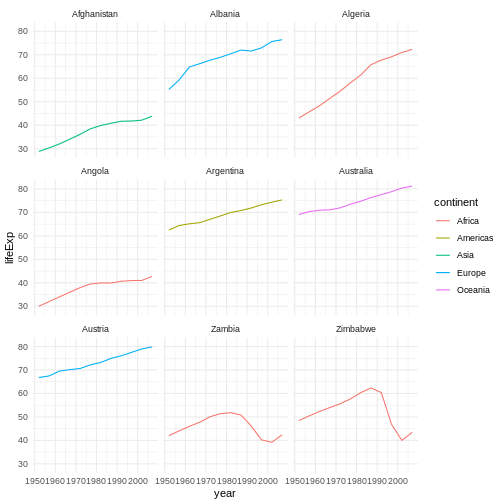
応用チャレンジ
2002年のデータから各大陸について2つの国をランダムに選び、平均寿命を計算してください。
その後、大陸名を逆順に並べ替えてください。 ヒント:
dplyr の関数 arrange() と
sample_n() を使用してください。これらは他の dplyr
関数と似た構文を持っています。
R
lifeExp_2countries_bycontinents <- gapminder %>%
filter(year==2002) %>%
group_by(continent) %>%
sample_n(2) %>%
summarize(mean_lifeExp=mean(lifeExp)) %>%
arrange(desc(mean_lifeExp))
その他の素晴らしいリソース
- R for Data Science (オンライン書籍)
- データ整形チートシート (PDFファイル)
- dplyr 入門 (オンラインドキュメント)
- R と RStudio を使用したデータ整形 (オンラインビデオ)
- データサイエンスのための Tidyverse スキル (オンライン書籍)
まとめ
- データフレームの操作に
dplyrパッケージを使用する。 -
select()を使用してデータフレームから変数を選択する。 -
filter()を使用して値に基づいてデータを選択する。 -
group_by()とsummarize()を使用してデータのサブセットを操作する。 -
mutate()を使用して新しい変数を作成する。
Content from tidyr を使用したデータフレームの操作
最終更新日:2024-11-22 | ページの編集
概要
質問
- データフレームのレイアウトをどのように変更できますか?
目的
- ’長い’データフレーム形式と’広い’データフレーム形式の概念を理解し、
tidyrを使ってそれらを変換できるようになる。
研究者はしばしば、データフレームの形式を「広い」レイアウトから「長い」レイアウトに変換したり、その逆を行ったりします。「長い」レイアウトまたは形式では以下のような特徴があります:
- 各列が変数
- 各行が観測値
純粋な「長い」形式では、通常、1列が観測値の変数であり、他の列がID変数です。
「広い」形式では、各行がサイト/被験者/患者を表し、同じ種類のデータを含む複数の観測変数があります。これには、時間経過での繰り返し観測や複数の変数の観測(またはその両方の混合)が含まれます。「広い」形式のほうがデータ入力が簡単である場合や、一部のアプリケーションが「広い」形式を好む場合があります。ただし、R
の多くの関数は「長い」形式のデータを想定して設計されています。このチュートリアルでは、元の形式に関係なく、データの形状を効率的に変換する方法を学びます。
長いデータフレームと広いデータフレームのレイアウトは主に可読性に影響します。人間にとって「広い」形式は、形状のために画面上でより多くのデータを確認できるため、より直感的な場合があります。しかし、「長い」形式は機械可読性が高く、データベースの形式に近いです。データフレームのID変数はデータベースのフィールドに、観測変数はデータベースの値に似ています。
開始方法
まず、まだインストールしていない場合は以下を実行してください(前回のレッスンでdplyrをインストールした可能性があります):
R
#install.packages("tidyr")
#install.packages("dplyr")
パッケージをロードします:
R
library("tidyr")
library("dplyr")
まず、元のgapminderデータフレームの構造を見てみましょう:
R
str(gapminder)
出力
'data.frame': 1704 obs. of 6 variables:
$ country : chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
$ year : int 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ pop : num 8425333 9240934 10267083 11537966 13079460 ...
$ continent: chr "Asia" "Asia" "Asia" "Asia" ...
$ lifeExp : num 28.8 30.3 32 34 36.1 ...
$ gdpPercap: num 779 821 853 836 740 ...チャレンジ 1
gapminder
は純粋に「長い」形式、「広い」形式、または中間形式のどれですか?
元の gapminder
データフレームは中間形式です。純粋に「長い」形式ではなく、複数の観測変数
(pop、lifeExp、gdpPercap)を持っています。
時々、gapminderデータセットのように、複数種類の観測データを持つことがあります。これは純粋な「長い」形式と「広い」形式の間のどこかに位置します。このデータセットには3つの「ID変数」
(continent、country、year)と3つの「観測変数」(pop、lifeExp、gdpPercap)があります。この中間形式は、すべての観測値を1列にする純粋に「長い」形式ではありませんが、それでも好まれる場合があります。これは、すべての観測変数が異なる単位を持っているためです。
多くのRの関数は「長い」形式を想定していますが、注意: 一部のプロット関数は「広い」形式のほうがうまく機能する場合があります。
pivot_longer() を使用して「広い」形式から「長い」形式に変換
これまで、適切にフォーマットされた元のgapminderデータセットを使用していましたが、「実際」のデータ(例:自分の研究データ)は通常、これほど整理されていません。ここでは、「広い」形式のgapminderデータセットを使用します。
「広い」形式の
gapminderデータをこちらのcsvファイルのリンクからダウンロードして、dataフォルダに保存してください。
データファイルをロードして確認しましょう。注意:continentとcountry列をファクターにしたくないため、read.csv()のstringsAsFactors引数を使用して無効にします。
R
gap_wide <- read.csv("data/gapminder_wide.csv", stringsAsFactors = FALSE)
str(gap_wide)
出力
'data.frame': 142 obs. of 38 variables:
$ continent : chr "Africa" "Africa" "Africa" "Africa" ...
$ country : chr "Algeria" "Angola" "Benin" "Botswana" ...
$ gdpPercap_1952: num 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num 3014 3828 960 918 617 ...
$ gdpPercap_1962: num 2551 4269 949 984 723 ...
$ gdpPercap_1967: num 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num 6223 4797 1441 12570 1217 ...
$ lifeExp_1952 : num 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num 72.3 42.7 56.7 50.7 52.3 ...
$ pop_1952 : num 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : int 31287142 10866106 7026113 1630347 12251209 7021078 15929988 4048013 8835739 614382 ...
$ pop_2007 : int 33333216 12420476 8078314 1639131 14326203 8390505 17696293 4369038 10238807 710960 ...
この非常に「広い」データフレームを「長い」レイアウトに戻すには、tidyrパッケージのpivot関数の1つであるpivot_longer()を使用します。この関数は、行数を増やして列数を減らすことによってデータセットを「長い」形式に変換します。
R
gap_long <- gap_wide %>%
pivot_longer(
cols = c(starts_with('pop'), starts_with('lifeExp'), starts_with('gdpPercap')),
names_to = "obstype_year", values_to = "obs_values"
)
str(gap_long)
出力
tibble [5,112 × 4] (S3: tbl_df/tbl/data.frame)
$ continent : chr [1:5112] "Africa" "Africa" "Africa" "Africa" ...
$ country : chr [1:5112] "Algeria" "Algeria" "Algeria" "Algeria" ...
$ obstype_year: chr [1:5112] "pop_1952" "pop_1957" "pop_1962" "pop_1967" ...
$ obs_values : num [1:5112] 9279525 10270856 11000948 12760499 14760787 ...ここでは、前回のdplyrレッスンで学んだパイピング構文を使用しています。実際、tidyrとdplyrの関数を組み合わせて使用することが可能です。
次に、separate()関数を使用して、文字列を複数の変数に分割します:
R
gap_long <- gap_long %>% separate(obstype_year, into = c('obs_type', 'year'), sep = "_")
gap_long$year <- as.integer(gap_long$year)
チャレンジ 2
gap_long を使用して、大陸ごとの平均寿命、人口、GDP per
capitaを計算してください。 ヒント:
dplyrレッスンで学んだgroup_by()とsummarize()関数を使用してください。
R
gap_long %>% group_by(continent, obs_type) %>%
summarize(means=mean(obs_values))
出力
`summarise()` has grouped output by 'continent'. You can override using the
`.groups` argument.出力
# A tibble: 15 × 3
# Groups: continent [5]
continent obs_type means
<chr> <chr> <dbl>
1 Africa gdpPercap 2194.
2 Africa lifeExp 48.9
3 Africa pop 9916003.
4 Americas gdpPercap 7136.
5 Americas lifeExp 64.7
6 Americas pop 24504795.
7 Asia gdpPercap 7902.
8 Asia lifeExp 60.1
9 Asia pop 77038722.
10 Europe gdpPercap 14469.
11 Europe lifeExp 71.9
12 Europe pop 17169765.
13 Oceania gdpPercap 18622.
14 Oceania lifeExp 74.3
15 Oceania pop 8874672. pivot_wider() を使用して長い形式から中間形式へ
作業を確認するのは常に良いことです。それでは、2つ目の
pivot 関数である pivot_wider()
を使用して、観測変数を再び「広げ」てみましょう。pivot_wider()
は pivot_longer()
の逆で、データセットの列数を増やし、行数を減らすことでデータを「広い」形式に変換します。pivot_wider()
を使用して、gap_long
を元の中間形式または最も広い形式に変換できます。まずは中間形式から始めましょう。
pivot_wider() 関数には names_from と
values_from 引数があります。
names_from
には、広げたデータフレームで新しい出力列としてピボットされる列名を指定します。対応する値は、values_from
引数で指定された列から追加されます。
R
gap_normal <- gap_long %>%
pivot_wider(names_from = obs_type, values_from = obs_values)
dim(gap_normal)
出力
[1] 1704 6R
dim(gapminder)
出力
[1] 1704 6R
names(gap_normal)
出力
[1] "continent" "country" "year" "pop" "lifeExp" "gdpPercap"R
names(gapminder)
出力
[1] "country" "year" "pop" "continent" "lifeExp" "gdpPercap"これで、元のgapminderと同じ次元を持つ中間形式のデータフレームgap_normalができましたが、変数の順序が異なります。これを修正してから、all.equal()
でチェックします。
R
gap_normal <- gap_normal[, names(gapminder)]
all.equal(gap_normal, gapminder)
出力
[1] "Attributes: < Component \"class\": Lengths (3, 1) differ (string compare on first 1) >"
[2] "Attributes: < Component \"class\": 1 string mismatch >"
[3] "Component \"country\": 1704 string mismatches"
[4] "Component \"pop\": Mean relative difference: 1.634504"
[5] "Component \"continent\": 1212 string mismatches"
[6] "Component \"lifeExp\": Mean relative difference: 0.203822"
[7] "Component \"gdpPercap\": Mean relative difference: 1.162302" R
head(gap_normal)
出力
# A tibble: 6 × 6
country year pop continent lifeExp gdpPercap
<chr> <int> <dbl> <chr> <dbl> <dbl>
1 Algeria 1952 9279525 Africa 43.1 2449.
2 Algeria 1957 10270856 Africa 45.7 3014.
3 Algeria 1962 11000948 Africa 48.3 2551.
4 Algeria 1967 12760499 Africa 51.4 3247.
5 Algeria 1972 14760787 Africa 54.5 4183.
6 Algeria 1977 17152804 Africa 58.0 4910.R
head(gapminder)
出力
country year pop continent lifeExp gdpPercap
1 Afghanistan 1952 8425333 Asia 28.801 779.4453
2 Afghanistan 1957 9240934 Asia 30.332 820.8530
3 Afghanistan 1962 10267083 Asia 31.997 853.1007
4 Afghanistan 1967 11537966 Asia 34.020 836.1971
5 Afghanistan 1972 13079460 Asia 36.088 739.9811
6 Afghanistan 1977 14880372 Asia 38.438 786.1134ほとんど完成ですが、元のデータはcountryとyearでソートされています。
R
gap_normal <- gap_normal %>% arrange(country, year)
all.equal(gap_normal, gapminder)
出力
[1] "Attributes: < Component \"class\": Lengths (3, 1) differ (string compare on first 1) >"
[2] "Attributes: < Component \"class\": 1 string mismatch >" 素晴らしいですね!最も長い形式から中間形式に戻り、コードにエラーがないことを確認できました。
次に、最も長い形式から最も広い形式まで変換してみましょう。この広い形式では、countryとcontinentをID変数として保持し、3つのメトリクス(pop、lifeExp、gdpPercap)と時間(year)にわたる観測値をピボットします。まず、新しい変数(時間*メトリクスの組み合わせ)に適切なラベルを作成し、ID変数を統合してgap_wideを定義しやすくします。
R
gap_temp <- gap_long %>% unite(var_ID, continent, country, sep = "_")
str(gap_temp)
出力
tibble [5,112 × 4] (S3: tbl_df/tbl/data.frame)
$ var_ID : chr [1:5112] "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" ...
$ obs_type : chr [1:5112] "pop" "pop" "pop" "pop" ...
$ year : int [1:5112] 1952 1957 1962 1967 1972 1977 1982 1987 1992 1997 ...
$ obs_values: num [1:5112] 9279525 10270856 11000948 12760499 14760787 ...R
gap_temp <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_")
str(gap_temp)
出力
tibble [5,112 × 3] (S3: tbl_df/tbl/data.frame)
$ ID_var : chr [1:5112] "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" "Africa_Algeria" ...
$ var_names : chr [1:5112] "pop_1952" "pop_1957" "pop_1962" "pop_1967" ...
$ obs_values: num [1:5112] 9279525 10270856 11000948 12760499 14760787 ...unite()
を使用して、continent、country
を統合した単一のID変数を作成し、変数名を定義しました。これで、pivot_wider()
に渡す準備が整いました。
R
gap_wide_new <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_") %>%
pivot_wider(names_from = var_names, values_from = obs_values)
str(gap_wide_new)
出力
tibble [142 × 37] (S3: tbl_df/tbl/data.frame)
$ ID_var : chr [1:142] "Africa_Algeria" "Africa_Angola" "Africa_Benin" "Africa_Botswana" ...
$ pop_1952 : num [1:142] 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num [1:142] 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num [1:142] 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num [1:142] 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num [1:142] 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num [1:142] 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num [1:142] 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num [1:142] 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num [1:142] 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num [1:142] 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num [1:142] 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num [1:142] 33333216 12420476 8078314 1639131 14326203 ...
$ lifeExp_1952 : num [1:142] 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num [1:142] 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num [1:142] 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num [1:142] 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num [1:142] 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num [1:142] 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num [1:142] 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num [1:142] 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num [1:142] 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num [1:142] 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num [1:142] 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num [1:142] 72.3 42.7 56.7 50.7 52.3 ...
$ gdpPercap_1952: num [1:142] 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num [1:142] 3014 3828 960 918 617 ...
$ gdpPercap_1962: num [1:142] 2551 4269 949 984 723 ...
$ gdpPercap_1967: num [1:142] 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num [1:142] 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num [1:142] 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num [1:142] 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num [1:142] 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num [1:142] 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num [1:142] 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num [1:142] 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num [1:142] 6223 4797 1441 12570 1217 ...チャレンジ 3
これをさらに一歩進めて、国、年、3つのメトリクスに基づいてピボットすることで、gap_ludicrously_wide
フォーマットデータを作成してください。 ヒント:
この新しいデータフレームには5行しかないはずです。
R
gap_ludicrously_wide <- gap_long %>%
unite(var_names, obs_type, year, country, sep = "_") %>%
pivot_wider(names_from = var_names, values_from = obs_values)
これで素晴らしい「広い」形式のデータフレームができましたが、ID_var
をより使いやすくするために、separate()
を使用して2つの変数に分割します。
R
gap_wide_betterID <- separate(gap_wide_new, ID_var, c("continent", "country"), sep="_")
gap_wide_betterID <- gap_long %>%
unite(ID_var, continent, country, sep = "_") %>%
unite(var_names, obs_type, year, sep = "_") %>%
pivot_wider(names_from = var_names, values_from = obs_values) %>%
separate(ID_var, c("continent","country"), sep = "_")
str(gap_wide_betterID)
出力
tibble [142 × 38] (S3: tbl_df/tbl/data.frame)
$ continent : chr [1:142] "Africa" "Africa" "Africa" "Africa" ...
$ country : chr [1:142] "Algeria" "Angola" "Benin" "Botswana" ...
$ pop_1952 : num [1:142] 9279525 4232095 1738315 442308 4469979 ...
$ pop_1957 : num [1:142] 10270856 4561361 1925173 474639 4713416 ...
$ pop_1962 : num [1:142] 11000948 4826015 2151895 512764 4919632 ...
$ pop_1967 : num [1:142] 12760499 5247469 2427334 553541 5127935 ...
$ pop_1972 : num [1:142] 14760787 5894858 2761407 619351 5433886 ...
$ pop_1977 : num [1:142] 17152804 6162675 3168267 781472 5889574 ...
$ pop_1982 : num [1:142] 20033753 7016384 3641603 970347 6634596 ...
$ pop_1987 : num [1:142] 23254956 7874230 4243788 1151184 7586551 ...
$ pop_1992 : num [1:142] 26298373 8735988 4981671 1342614 8878303 ...
$ pop_1997 : num [1:142] 29072015 9875024 6066080 1536536 10352843 ...
$ pop_2002 : num [1:142] 31287142 10866106 7026113 1630347 12251209 ...
$ pop_2007 : num [1:142] 33333216 12420476 8078314 1639131 14326203 ...
$ lifeExp_1952 : num [1:142] 43.1 30 38.2 47.6 32 ...
$ lifeExp_1957 : num [1:142] 45.7 32 40.4 49.6 34.9 ...
$ lifeExp_1962 : num [1:142] 48.3 34 42.6 51.5 37.8 ...
$ lifeExp_1967 : num [1:142] 51.4 36 44.9 53.3 40.7 ...
$ lifeExp_1972 : num [1:142] 54.5 37.9 47 56 43.6 ...
$ lifeExp_1977 : num [1:142] 58 39.5 49.2 59.3 46.1 ...
$ lifeExp_1982 : num [1:142] 61.4 39.9 50.9 61.5 48.1 ...
$ lifeExp_1987 : num [1:142] 65.8 39.9 52.3 63.6 49.6 ...
$ lifeExp_1992 : num [1:142] 67.7 40.6 53.9 62.7 50.3 ...
$ lifeExp_1997 : num [1:142] 69.2 41 54.8 52.6 50.3 ...
$ lifeExp_2002 : num [1:142] 71 41 54.4 46.6 50.6 ...
$ lifeExp_2007 : num [1:142] 72.3 42.7 56.7 50.7 52.3 ...
$ gdpPercap_1952: num [1:142] 2449 3521 1063 851 543 ...
$ gdpPercap_1957: num [1:142] 3014 3828 960 918 617 ...
$ gdpPercap_1962: num [1:142] 2551 4269 949 984 723 ...
$ gdpPercap_1967: num [1:142] 3247 5523 1036 1215 795 ...
$ gdpPercap_1972: num [1:142] 4183 5473 1086 2264 855 ...
$ gdpPercap_1977: num [1:142] 4910 3009 1029 3215 743 ...
$ gdpPercap_1982: num [1:142] 5745 2757 1278 4551 807 ...
$ gdpPercap_1987: num [1:142] 5681 2430 1226 6206 912 ...
$ gdpPercap_1992: num [1:142] 5023 2628 1191 7954 932 ...
$ gdpPercap_1997: num [1:142] 4797 2277 1233 8647 946 ...
$ gdpPercap_2002: num [1:142] 5288 2773 1373 11004 1038 ...
$ gdpPercap_2007: num [1:142] 6223 4797 1441 12570 1217 ...R
all.equal(gap_wide, gap_wide_betterID)
出力
[1] "Names: 24 string mismatches"
[2] "Attributes: < Component \"class\": Lengths (1, 3) differ (string compare on first 1) >"
[3] "Attributes: < Component \"class\": 1 string mismatch >"
[4] "Component 3: Mean relative difference: 4549.106"
[5] "Component 4: Mean relative difference: 4363.185"
[6] "Component 5: Mean relative difference: 4320.163"
[7] "Component 6: Mean relative difference: 4130.972"
[8] "Component 7: Mean relative difference: 3719.779"
[9] "Component 8: Mean relative difference: 3783.459"
[10] "Component 9: Mean relative difference: 4016.515"
[11] "Component 10: Mean relative difference: 4180.611"
[12] "Component 11: Mean relative difference: 4410.404"
[13] "Component 12: Mean relative difference: 4271.686"
[14] "Component 13: Mean relative difference: 4179.099"
[15] "Component 14: Mean relative difference: 3767.917"
[16] "Component 27: Mean relative difference: 0.9997802"
[17] "Component 28: Mean relative difference: 0.9997709"
[18] "Component 29: Mean relative difference: 0.9997686"
[19] "Component 30: Mean relative difference: 0.999758"
[20] "Component 31: Mean relative difference: 0.9997312"
[21] "Component 32: Mean relative difference: 0.9997358"
[22] "Component 33: Mean relative difference: 0.9997511"
[23] "Component 34: Mean relative difference: 0.9997609"
[24] "Component 35: Mean relative difference: 0.9997733"
[25] "Component 36: Mean relative difference: 0.999766"
[26] "Component 37: Mean relative difference: 0.9997608"
[27] "Component 38: Mean relative difference: 0.9997347" 行って戻るプロセスが完了しました!
その他の素晴らしいリソース
- R for Data Science (オンライン書籍)
- データ整形チートシート (PDFファイル)
- tidyr 入門 (オンラインドキュメント)
- R と RStudio を使用したデータ整形 (オンラインビデオ)
まとめ
- データフレームのレイアウトを変更するために
tidyrパッケージを使用する。 -
pivot_longer()を使用して「広い」レイアウトから「長い」レイアウトに変換する。 -
pivot_wider()を使用して「長い」レイアウトから「広い」レイアウトに変換する。
Content from knitrを使ったレポート作成
最終更新日:2024-11-22 | ページの編集
概要
質問
- ソフトウェアとレポートをどのように統合できますか?
目的
- 再現性のあるレポート作成の価値を理解する
- R Markdownファイルの基本構成を認識し、コンパイルする方法を学ぶ
- Rコードチャンクを理解し、その目的、構造、およびオプションを学ぶ
- 計算結果を議論する際に、テキストブロックにRの出力を埋め込むインラインチャンクの使用方法を実演する
- R Markdownファイルを他の形式にエクスポートする際の代替出力形式を把握する
データ解析レポート
データ解析者は通常、コラボレーター向けや将来の参照のために、自分の解析や結果を記述したレポートを多く作成します。
初心者の多くは、まず解析をすべて1つのRスクリプトに書き込み、スクリプトやさまざまなグラフを添付ファイルとしてメールで共有することから始めます。しかし、これは手間がかかり、どの添付ファイルがどの結果なのかを説明するのに長い議論を要する場合があります。
WordやLaTeXを使って正式なレポートを書くと、このプロセスが簡単になります。解析レポートと出力グラフを1つの文書にまとめることができます。ただし、図の形式を整えたり、煩わしい改ページを修正したりするのに手間がかかり、一度の形式変更で新たな問題が発生する「モグラ叩きゲーム」のような状況になりがちです。
R Markdownを使ってWebページ(htmlファイル)としてレポートを作成すると、これらの作業が簡単になります。この形式では、レポートを長いストリームとして作成できるため、通常は1ページに収まらない大きな図も完全なサイズで保持でき、読み手は単にスクロールするだけで内容を確認できます。また、R Markdown文書の形式はシンプルで、簡単に変更できるため、レポート作成ではなく解析に時間を費やすことができます。
文芸的プログラミング(Literate Programming)
理想的には、このような解析レポートは再現性のある文書です。エラーが見つかったり、データに新しい被験者が追加されたりした場合でも、レポートを再コンパイルするだけで、新しい結果や修正された結果を得ることができます。図を再作成し、Word文書に貼り付け、詳細な結果を手作業で編集する必要はありません。
ここで重要なRパッケージはknitrです。このパッケージを使うと、テキストとコードチャンクを混在させた文書を作成できます。knitrが文書を処理すると、コードチャンクが実行され、グラフやその他の結果が最終文書に挿入されます。
このようなアイデアは「文芸的プログラミング」と呼ばれています。
knitrでは、ほぼすべての種類のテキストとさまざまなプログラミング言語のコードを混在させることができますが、R Markdownを使用することをお勧めします。R MarkdownはMarkdownとRを組み合わせたものです。Markdownは、Webページを作成するための軽量マークアップ言語です。
R Markdownファイルの作成
RStudio内で、[File] → [New File] → [R Markdown] をクリックすると、次のようなダイアログボックスが表示されます:
デフォルト設定(HTML出力)をそのまま使用しても構いませんが、タイトルを入力してください。
R Markdownの基本構成
冒頭のテキストチャンク(ヘッダー)は、どの種類の文書を作成するか、選択されたオプションとともにRに指示を与えます。このヘッダーを使用して、文書にタイトル、著者、日付を指定し、作成したい出力形式を指定できます。この例では、HTML文書を作成します。
---
title: "Initial R Markdown document"
author: "Karl Broman"
date: "April 23, 2015"
output: html_document
---これらのフィールドは、不要であれば削除できます。ダブルクォートは必須ではありません。必要になるのは、タイトルにコロンを含めたい場合などです。
RStudioは、始めるためのサンプルテキスト付きで文書を作成します。以下のようなチャンクに注意してください:
```{r}
summary(cars)
```
これらはRコードチャンクで、knitrによって実行され、その結果で置き換えられます。詳細は後ほど説明します。
Markdown
Markdownは、メールを書くようにテキストをマークアップすることでWebページを作成するシステムです。マークアップされたテキストはHTMLコードに変換され、適切なHTMLコードに置き換えられます。
チャレンジ 1
新しいR Markdown文書を作成してください。すべてのRコードチャンクを削除し、Markdownの一部(セクション、斜体のテキスト、箇条書きリスト)を記述します。
文書をWebページに変換してください。
RStudioで、[File] → [New File] → [R Markdown…] を選択します。
プレースホルダーテキストを削除し、以下を追加します:
# Introduction
## Background on Data
このレポートでは、*gapminder* データセットを使用しています。このデータセットには次の列が含まれます:
* country
* continent
* year
* lifeExp
* pop
* gdpPercap
## Background on Methods
その後、ツールバーの「Knit」ボタンをクリックして、HTMLドキュメント(Webページ)を生成します。
もう少しMarkdownについて
ハイパーリンクを作成するには、以下のように記述します:
[Carpentries ホームページ](https://carpentries.org/)。
画像ファイルを挿入するには、以下のように記述します:

添字(例:F2)は
F~2~、上付き文字(例:F2)は F^2^
と記述します。
LaTeXで方程式を書く方法を知っていれば、$ $
や $$ $$ を使って数式を挿入できます。
例えば、$E = mc^2$ または次のような形で記述できます:
$$y = \mu + \sum_{i=1}^p \beta_i x_i + \epsilon$$Markdownの構文は、RStudioのツールバーの「Help」フィールドにある “Markdown Quick Reference” で確認できます。
Rコードチャンク
Markdownの真の力は、マークダウンにコードチャンクを組み合わせるところにあります。これがR Markdownです。処理されると、Rコードは実行され、生成される図などが最終文書に挿入されます。
基本的なコードチャンクは以下のようになります:
```{r load_data}
gapminder
つまり、```{r chunk_name} と ```
の間にRコードを記述します。各チャンクには一意の名前を付けるべきです。これによりエラーの修正が容易になり、図が生成される場合、生成されるファイル名はそのコードチャンクの名前に基づきます。RStudioでは、ショートカット
Ctrl+Alt+I(WindowsおよびLinux)、または
Cmd+Option+I(Mac)を使用してコードチャンクを素早く作成できます。
コンパイルの仕組み
「Knit」ボタンを押すと、R Markdown文書はknitrによって処理され、プレーンなMarkdown文書(および必要に応じて一連の図ファイル)が生成されます。Rコードは実行され、その入力と出力に置き換えられます。図が生成される場合、それらのリンクが文書に含まれます。
その後、Markdown文書と図ファイルはpandocツールによって処理され、図が埋め込まれたHTMLファイルに変換されます。
チャンクオプション
コードチャンクの処理方法を変更するためのオプションがさまざま用意されています。例:
-
echo=FALSEを使用してコード自体を非表示にする。 -
results="hide"を使用して結果の印刷を抑制する。 -
eval=FALSEを使用してコードを表示するが評価しない。 -
warning=FALSEやmessage=FALSEを使用して警告やメッセージを非表示にする。 -
fig.heightやfig.widthを使用して生成される図のサイズ(インチ単位)を制御する。
たとえば、以下のように記述します:
```{r load_libraries, echo=FALSE, message=FALSE}
library("dplyr")
library("ggplot2")
```
R Markdownのすべてのチャンクオプションは、RStudioのツールバーの「Help」フィールド内「Cheatsheets」セクションにある「R Markdown Cheat Sheet」で確認できます。
インラインRコード
レポート内のすべての数値を再現可能にすることができます。`r と `
を使用してインラインコードチャンクを作成します。
例: `r round(some_value, 2)`.
このコードは実行され、結果の値に置き換えられます。
インラインチャンクが行をまたいで分割されないように注意してください。
計算や変数の定義を行う大きなコードチャンクを、include=FALSE
を指定して記述するのが良いでしょう
(これは echo=FALSE と results="hide"
を指定するのと同じです)。
丸め処理は、場合によって出力に差異を生じさせることがあります。たとえば
2.0 を期待していても、round(2.03, 1) は
2 だけを返します。
R/broman
パッケージのmyround
関数は、この問題を処理します。
チャレンジ 4
インラインRコードを少し試してみましょう。
以下は、2 + 2 = 4 を求めるインラインコードの例です。
他の出力オプション
R Markdown を PDF または Word
ドキュメントに変換することもできます。
“ニット”
ボタン横の小さな三角形をクリックすると、ドロップダウンメニューが表示されます。
または、ファイルの冒頭ヘッダーに pdf_document または
word_document を指定することも可能です。
ヒント: PDFドキュメントの作成
PDF
ドキュメント(.pdf)を作成するには、追加のソフトウェアが必要になる場合があります。
Rパッケージ tinytex
は、このプロセスを簡単にするツールを提供します。tinytex
をインストールした後、tinytex::install_tinytex()
を実行して必要なソフトウェアをインストールしてください(この作業は一度だけでOKです)。
その後、PDFにニットする際に tinytex が自動的に必要な LaTeX
パッケージを検出し、インストールします。
詳細については、tinytexの公式ウェブサイト
をご覧ください。
ヒント: RStudio のビジュアルマークダウン編集
RStudio バージョン 1.4
以降には、ビジュアルマークダウン編集モードが含まれています。
このモードでは、マークダウン表記(例:
**太字**)が入力中にフォーマットされた外観(太字)に変換されます。
また、このモードには上部に基本的なフォーマットボタンを含むツールバーがあり、一般的なワープロソフトに似た操作が可能です。
ビジュアル編集モードは、R Markdown ドキュメントの右上隅にある![]() ボタンを押すことでオン/オフを切り替えられます。
ボタンを押すことでオン/オフを切り替えられます。
リソース
- Knitr in a knutshell tutorial
- Dynamic Documents with R and knitr (書籍)
- R Markdown documentation
- R Markdown cheat sheet
- Getting started with R Markdown
- R Markdown: The Definitive Guide (Rstudio チームの書籍)
- Reproducible Reporting
- The Ecosystem of R Markdown
- Introducing Bookdown
まとめ
- R Markdown で作成したレポートに、R で書かれたソフトウェアを組み合わせる。
- チャンクオプションを指定してフォーマットを制御する。
-
knitrを使用して、これらのドキュメントを PDF や他の形式に変換する。
Content from 良いソフトウェアを書く方法
最終更新日:2024-11-22 | ページの編集
概要
質問
- 他の人が使えるソフトウェアをどのように書けばよいですか?
目的
- R を書く際のベストプラクティスを説明し、それぞれの正当性を説明する。
プロジェクトフォルダを構造化する
プロジェクトフォルダは、コードファイル、マニュアル、データ、バイナリ、出力プロットなどのサブフォルダを作成して、構造化され、整理された状態を保つようにしましょう。
これは完全に手動で行うことも、RStudio の New Project
機能や、ProjectTemplate
のような専用パッケージを利用することも可能です。
ヒント: ProjectTemplate - 可能な解決策
プロジェクトの管理を自動化する方法の一つとして、サードパーティパッケージ
ProjectTemplate をインストールすることがあります。
このパッケージはプロジェクト管理のための理想的なディレクトリ構造を設定します。
分析パイプラインやワークフローを整理し、構造化するのに非常に便利です。
RStudio の標準プロジェクト機能や Git
と組み合わせることで、自分の作業を追跡し、共同作業者と簡単に共有できるようになります。
-
ProjectTemplateをインストールします。 - ライブラリを読み込みます。
- プロジェクトを初期化します。
R
install.packages("ProjectTemplate")
library("ProjectTemplate")
create.project("../my_project_2", merge.strategy = "allow.non.conflict")
ProjectTemplate
とその機能に関する詳細は、公式ホームページ ProjectTemplate
を参照してください。
コードを読みやすくする
コードを書く際に最も重要なのは、それを読みやすく、理解しやすくすることです。
他の誰かがコードを見て、それが何をしているか理解できるようにする必要があります。
多くの場合、この「他の誰か」とは、6
ヶ月後の自分自身であり、そのときに過去の自分に腹を立てている可能性があります。
ドキュメント: 「どのように」ではなく、「何を」「なぜ」を伝える
最初は、コマンドが何をするかを説明するコメントを書くことが多いでしょう。
それは、自分自身が学んでいる最中で、概念を明確にしたり、後で思い出す助けになるからです。
しかし、このようなコメントは、後になってコードがどの問題を解決しようとしているのかを思い出せない場合には、あまり役に立ちません。
なぜ 問題を解決しようとしているのか、そして 何
の問題なのかを説明するコメントを含めるようにしてください。
どのように
はその後で良いです。それは実装の詳細であり、理想的には気にする必要がないものです。
コードをモジュール化する
おすすめの方法は、関数を分析スクリプトから分離し、別のファイルに保存することです。
プロジェクト内で R セッションを開くときにそのファイルを
source します。
このアプローチは、分析スクリプトをすっきりさせるだけでなく、便利な関数のリポジトリを作成することができ、プロジェクト内のどの分析スクリプトにも簡単にロードできます。
また、関連する関数を簡単にグループ化することも可能です。
問題を小さな部分に分解する
問題解決や関数作成は、最初は難しく感じられ、コードの経験不足と切り離しにくいことがあります。
問題を消化しやすい部分に分解し、実装の詳細は後回しにしましょう。
問題をどんどん小さな関数に分解し、コードで解決できるレベルに達するまで続けてください。そしてそこから組み立て直しましょう。
コードが正しく動作していることを確認する
関数をテストすることを忘れないでください!
同じことを繰り返さない
関数はプロジェクト内での簡単な再利用を可能にします。
プロジェクト全体に似たようなコード行が繰り返されている場合、それらは関数にまとめる候補になります。
計算が一連の関数を通じて実行される場合、プロジェクトはよりモジュール化され、変更が容易になります。
特に、特定の入力が常に特定の出力を生成する場合には有効です。
スタイルを忘れずに
コードには一貫したスタイルを適用しましょう。
まとめ
- プロジェクトフォルダを構造化し、整理整頓を保つ。
- 「何を」「なぜ」を文書化し、「どのように」は後回しにする。
- プログラムを短く、単一目的の関数に分解する。
- 再実行可能なテストを書く。
- 同じことを繰り返さない。
- 命名規則、インデント、その他のスタイルの側面で一貫性を保つ。
