dplyrによるデータの操作と分析
最終更新日:2024-09-08 | ページの編集
概要
質問
- tidyverseメタパッケージを用いたRでのデータ分析
目的
- dplyr
** と **tidyr** パッケージの目的を説明する。 - データを操作するのに非常に便利な関数をいくつか説明する。
- ワイド表形式とロング表形式の概念を説明し、 、データ・フレームを一方の形式から他方の形式に変更する方法を見る。
- テーブルの結合方法を示す。
このエピソードは、Data Carpentriesの_Data Analysis and Visualisation in R for Ecologists_レッスンに基づいています。
dplyr**と**tidyr**を使ったデータ操作
ブラケット・サブセットは便利だが、煩雑で 、特に複雑な操作では読みにくい。
いくつかのパッケージは、データを操作する際に私たちの作業を大いに助けてくれる。
Rのパッケージは基本的に、
、より多くのことができるようにする追加関数のセットである。
いくつかのパッケージは、データを操作する際に私たちの作業を大いに助けてくれる。
Rのパッケージは基本的に、
、より多くのことができるようにする追加関数のセットである。
これまで使ってきた str() や data.frame()
などの関数は、Rに組み込まれています。パッケージをロードすることで、その他の
固有の関数にアクセスできるようになります。
初めてパッケージを使用する前に、
をマシンにインストールする必要がある。その後、 R
セッションでパッケージが必要になったら、毎回インポートする必要がある。
初めてパッケージを使用する前に、
をマシンにインストールする必要がある。その後、 R
セッションでパッケージが必要になったら、毎回インポートする必要がある。
dplyr\`** パッケージは、データ操作タスクのための強力なツールを提供します。 データフレームを直接操作できるように構築されており、多くの操作タスクが に最適化されている。 データフレームを直接操作できるように構築されており、多くの操作タスクが に最適化されている。
後述するように、 、特定の分析や視覚化を行うために、データフレームの形を変えたいことがある。 tidyr`**パッケージは、 、データの形を変えるというこの一般的な問題に対処し、 データを整然と操作するためのツールを提供する。
ワークショップの後、**dplyr**と**tidyr**についてもっと知りたい方は、
、こちらのhandy data transformation with をご覧ください。
- tidyverse
**パッケージは "umbrella-package "であり、 、データ解析のためのいくつかの便利なパッケージがインストールされます。 には、**tidyr**, dplyr**, **ggplot2, **tibble\`**などがあります。 これらのパッケージは、データを操作したり対話したりするのに役立ちます。 サブセット化、変換、 ビジュアライズなど、データを使ってさまざまなことができる。 サブセット化、変換、 ビジュアライズなど、データを使ってさまざまなことができる。
セットアップを行ったのであれば、すでにtidyverseパッケージがインストールされているはずです。 ライブラリから読み込んでみて、それがあるかどうか確認してください: ライブラリから読み込んでみて、それがあるかどうか確認してください:
R
## dplyr を含む tidyverse パッケージをロード
library("tidyverse")
tidyverse\`** パッケージをインストールするには、以下のようにタイプしてください:
R
BiocManager::install("tidyverse")
もし、**tidyverse**パッケージをインストールしなければならなかったなら、上記のlibrary()\`コマンドを使って、このRセッションでロードすることを忘れないでください!
tidyverseでデータをロードする
read.csv()の代わりに、tidyverseパッケージ **readr**の
read_csv()
関数(.の代わりに_があることに注意)を使ってデータを読み込みます。
R
rna <- read_csv("data/rnaseq.csv")
## データを見る
rna
出力
# A tibble: 32,428 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 1170 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 Apod GSM254… 36194 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
3 Cyp2d22 GSM254… 4060 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
4 Klk6 GSM254… 287 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
5 Fcrls GSM254… 85 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
6 Slc2a4 GSM254… 782 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
7 Exd2 GSM254… 1619 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 Gjc2 GSM254… 288 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
9 Plp1 GSM254… 43217 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
10 Gnb4 GSM254… 1071 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>データのクラスが “tibble”と呼ばれていることに注目してほしい。
Tibblesは、以前 で紹介したデータ・フレーム・オブジェクトの動作の一部を微調整している。 データ構造はデータフレームによく似ている。 我々の目的にとって、唯一の違いはそれだ:
各列のデータ型が列名の下に表示される。 <
dbl> は の小数点を持つ数値を保持するために定義されたデータ型である。 データで作業するとき、我々はしばしば、各因子または因子の組み合わせについて 見つかったオブザベーションの数を知りたい。 このタスクのために、**dplyr** はcount()\` を提供している。 例えば、感染したサンプルと感染していないサンプルそれぞれについて、 、データの行数をカウントしたい場合、次のようにする:これは、データの最初の数行と、 1画面に収まるだけの列数だけを印刷する。
これから、最も一般的な dplyr\` 関数のいくつかを学びます:
- select()\`: カラムのサブセット
-
filter(): 条件で行をサブセットする。 - mutate()\`: 他のカラムの情報を使って新しいカラムを作成する。
- group_by()
とsummarise()\`: グループ化されたデータの要約統計量を作成する。 - arrange()\`:結果の並べ替え
- count()\`: 離散値を数える
列の選択と行のフィルタリング
データフレームの列を選択するには select() を使う。
この関数の最初の引数 はデータフレーム (rna) で、続く
の引数は保持する列です。 Tibblesは、以前
で紹介したデータ・フレーム・オブジェクトの動作の一部を微調整している。
データ構造はデータフレームによく似ている。
我々の目的にとって、唯一の違いはそれだ:
R
select(rna, gene, sample, tissue, expression)
出力
# A tibble: 32,428 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545336 Cerebellum 1170
2 Apod GSM2545336 Cerebellum 36194
3 Cyp2d22 GSM2545336 Cerebellum 4060
4 Klk6 GSM2545336 Cerebellum 287
5 Fcrls GSM2545336 Cerebellum 85
6 Slc2a4 GSM2545336 Cerebellum 782
7 Exd2 GSM2545336 Cerebellum 1619
8 Gjc2 GSM2545336 Cerebellum 288
9 Plp1 GSM2545336 Cerebellum 43217
10 Gnb4 GSM2545336 Cerebellum 1071
# ℹ 32,418 more rows特定の列を除く*すべての列を選択するには、 その変数の前に”-“を付けて除外する。
R
select(rna, -tissue, -organism)
出力
# A tibble: 32,428 × 17
gene sample expression age sex infection strain time mouse ENTREZID
<chr> <chr> <dbl> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl>
1 Asl GSM2545… 1170 8 Fema… Influenz… C57BL… 8 14 109900
2 Apod GSM2545… 36194 8 Fema… Influenz… C57BL… 8 14 11815
3 Cyp2d22 GSM2545… 4060 8 Fema… Influenz… C57BL… 8 14 56448
4 Klk6 GSM2545… 287 8 Fema… Influenz… C57BL… 8 14 19144
5 Fcrls GSM2545… 85 8 Fema… Influenz… C57BL… 8 14 80891
6 Slc2a4 GSM2545… 782 8 Fema… Influenz… C57BL… 8 14 20528
7 Exd2 GSM2545… 1619 8 Fema… Influenz… C57BL… 8 14 97827
8 Gjc2 GSM2545… 288 8 Fema… Influenz… C57BL… 8 14 118454
9 Plp1 GSM2545… 43217 8 Fema… Influenz… C57BL… 8 14 18823
10 Gnb4 GSM2545… 1071 8 Fema… Influenz… C57BL… 8 14 14696
# ℹ 32,418 more rows
# ℹ 7 more variables: product <chr>, ensembl_gene_id <chr>,
# external_synonym <chr>, chromosome_name <chr>, gene_biotype <chr>,
# phenotype_description <chr>, hsapiens_homolog_associated_gene_name <chr>これは rna の中の、 tissue と
organism 以外のすべての変数を選択する。
特定の条件に基づいて行を選択するには、filter()
を使用する:
R
filter(rna, sex == "Male")
出力
# A tibble: 14,740 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 626 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
2 Apod GSM254… 13021 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
3 Cyp2d22 GSM254… 2171 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
4 Klk6 GSM254… 448 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
5 Fcrls GSM254… 180 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
6 Slc2a4 GSM254… 313 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
7 Exd2 GSM254… 2366 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
8 Gjc2 GSM254… 310 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
9 Plp1 GSM254… 53126 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
10 Gnb4 GSM254… 1355 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
# ℹ 14,730 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>R
filter(rna, sex == "Male" & infection == "NonInfected")
出力
# A tibble: 4,422 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 535 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
2 Apod GSM254… 13668 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
3 Cyp2d22 GSM254… 2008 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
4 Klk6 GSM254… 1101 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
5 Fcrls GSM254… 375 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
6 Slc2a4 GSM254… 249 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
7 Exd2 GSM254… 3126 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
8 Gjc2 GSM254… 791 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
9 Plp1 GSM254… 98658 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
10 Gnb4 GSM254… 2437 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
# ℹ 4,412 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>ここで、このデータセットで解析されたマウス
遺伝子のヒトホモログに興味があるとしよう。 この情報は、
hsapiens_homolog_associated_gene_name という名前の
rna tibbleの 最後のカラムにある。
ここで、このデータセットで解析されたマウス
遺伝子のヒトホモログに興味があるとしよう。 この情報は、
hsapiens_homolog_associated_gene_name という名前の
rna tibbleの 最後のカラムにある。 簡単に視覚化するために、
、2つの列geneと
hsapiens_homolog_associated_gene_nameだけを含む新しいテーブルを作成する。
R
genes <- select(rna, gene, hsapiens_homolog_associated_gene_name)
genes
出力
# A tibble: 32,428 × 2
gene hsapiens_homolog_associated_gene_name
<chr> <chr>
1 Asl ASL
2 Apod APOD
3 Cyp2d22 CYP2D6
4 Klk6 KLK6
5 Fcrls FCRL2
6 Slc2a4 SLC2A4
7 Exd2 EXD2
8 Gjc2 GJC2
9 Plp1 PLP1
10 Gnb4 GNB4
# ℹ 32,418 more rowsマウス遺伝子の中にはヒトにホモログがないものもある。 これらは、
filter() と、 何かが NA かどうかを判定する
is.na() 関数を使って取得することができる。
R
filter(genes, is.na(hsapiens_homolog_associated_gene_name))
出力
# A tibble: 4,290 × 2
gene hsapiens_homolog_associated_gene_name
<chr> <chr>
1 Prodh <NA>
2 Tssk5 <NA>
3 Vmn2r1 <NA>
4 Gm10654 <NA>
5 Hexa <NA>
6 Sult1a1 <NA>
7 Gm6277 <NA>
8 Tmem198b <NA>
9 Adam1a <NA>
10 Ebp <NA>
# ℹ 4,280 more rowsヒトのホモログを持つマウス遺伝子だけを保持したい場合、
、結果を否定する”!“記号を挿入することができる。したがって、
、hsapiens_homolog_associated_gene_name is not an
NA となるすべての行を求めることになる。
R
filter(genes, !is.na(hsapiens_homolog_associated_gene_name))
出力
# A tibble: 28,138 × 2
gene hsapiens_homolog_associated_gene_name
<chr> <chr>
1 Asl ASL
2 Apod APOD
3 Cyp2d22 CYP2D6
4 Klk6 KLK6
5 Fcrls FCRL2
6 Slc2a4 SLC2A4
7 Exd2 EXD2
8 Gjc2 GJC2
9 Plp1 PLP1
10 Gnb4 GNB4
# ℹ 28,128 more rowsパイプ
選択とフィルタを同時に行いたい場合は? これを行うには、 、中間ステップ、ネストされた関数、パイプの3つの方法がある。 これを行うには、 、中間ステップ、ネストされた関数、パイプの3つの方法がある。
中間ステップでは、一時的なデータフレームを作成し、 、次の関数の入力として使用する:
R
rna2 <- filter(rna, sex == "Male")
rna3 <- select(rna2, gene, sample, tissue, expression)
rna3
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsこれは読みやすいが、 、個別に名前を付けなければならない中間オブジェクトがたくさんあるため、ワークスペースが散らかる可能性がある。 複数の 、それを把握するのは難しいかもしれない。 複数の 、それを把握するのは難しいかもしれない。
、関数を入れ子にすることもできる:
R
rna3 <- select(filter(rna, sex == "Male", gene, sample, tissue, expression))
エラー
Error in `filter()`:
ℹ In argument: `gene`.
Caused by error:
! `..2` must be a logical vector, not a character vector.R
rna3
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsこれは便利だが、 Rは式を内側から外側へと評価する(この場合、フィルタリングしてから選択する)ため、関数が入れ子になりすぎると読みにくくなることがある。
最後のオプションである_パイプ_は、Rに最近追加されたものである。パイプを使うと、ある関数の出力を 、次の関数に直接送ることができる。これは、同じデータセットに対して多くの処理を行う必要がある場合に便利である 。
ミューテート R のパイプは %>%
(magrittr パッケージで利用可能) または
|> (ベース R で利用可能) のように見えます。
RStudioを使用する場合は、
PCをお持ちの場合はCtrl+Shift+M、
Macをお持ちの場合はCmd+Shift+Mでパイプを
。
上記のコードでは、パイプを使って rna データセットをまず
filter() を通して sex が Male
である行を残し、次に select() を通して gene,
sample, tissue, expressioncolumns
だけを残すように送っている。
パイプ %>% はその左側にあるオブジェクトを受け取り、
その右側にある関数の最初の引数として直接渡します。 filter()
と select()
関数の引数として明示的にデータフレームを含める必要はもうありません。
R
rna %>%
filter(sex == "Male") %>%
select(gene, sample, tissue, expression)
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsパイプを “then”のように読むことが役に立つと思う人もいるだろう。
パイプを “then”のように読むことが役に立つと思う人もいるだろう。 例えば、
上の例では、データフレーム rna
を取得し、sex=="Male" の行を で
フィルターし、gene, sample,
tissue, expression の列を 選択
した。
dplyr\`**関数はそれ自体ではやや単純だが、 、パイプを使った線形ワークフローに組み合わせることで、 、データフレームのより複雑な操作を行うことができる。
この小さいバージョンのデータで新しいオブジェクトを作りたい場合、 、新しい名前を割り当てることができる:
R
rna3<- rna %>%
filter(sex == "Male") %>%
select(gene, sample, tissue, expression)
rna3
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsR
rna %>%
filter(expression > 50000,
sex == "Female",
time == 0 ) %>%
select(gene, sample, time, expression, age)
出力
# A tibble: 9 × 5
gene sample time expression age
<chr> <chr> <dbl> <dbl> <dbl>
1 Plp1 GSM2545337 0 101241 8
2 Atp1b1 GSM2545337 0 53260 8
3 Plp1 GSM2545338 0 96534 8
4 Atp1b1 GSM2545338 0 50614 8
5 Plp1 GSM2545348 0 102790 8
6 Atp1b1 GSM2545348 0 59544 8
7 Plp1 GSM2545353 0 71237 8
8 Glul GSM2545353 0 52451 8
9 Atp1b1 GSM2545353 0 61451 8ミューテート
例えば、単位変換をしたり、2つの
列の値の比率を求めたりするために、既存の
列の値に基づいて新しい列を作成したいことがよくあります。 これには
mutate() を使う。 これには mutate()
を使う。
時間単位の新しい列を作成する:
R
rna %>%
mutate(time_hours = time * 24) %>%
select(time, time_hours)
出力
# A tibble: 32,428 × 2
time time_hours
<dbl> <dbl>
1 8 192
2 8 192
3 8 192
4 8 192
5 8 192
6 8 192
7 8 192
8 8 192
9 8 192
10 8 192
# ℹ 32,418 more rowsまた、mutate()の同じ呼び出しの中で、最初の新しいカラムに基づいて2番目の新しいカラムを作成することもできる:
R
rna %>%
mutate(time_hours = time * 24,
time_mn = time_hours * 60) %>%
select(time, time_hours, time_mn)
出力
# A tibble: 32,428 × 3
time time_hours time_mn
<dbl> <dbl> <dbl>
1 8 192 11520
2 8 192 11520
3 8 192 11520
4 8 192 11520
5 8 192 11520
6 8 192 11520
7 8 192 11520
8 8 192 11520
9 8 192 11520
10 8 192 11520
# ℹ 32,418 more rowsチャレンジ
後述するように、
、特定の分析や視覚化を行うために、データフレームの形を変えたいことがある。
tidyr`**パッケージは、
、データの形を変えるというこの一般的な問題に対処し、
データを整然と操作するためのツールを提供する。 の値は対数変換する。
以下の 条件を満たす
rnaデータから新しいデータフレームを作成する:gene、chromosome_name、
phenotype_description、sample、expression\
列のみを含む。 の値は対数変換する。 このデータフレームは、
、性染色体に位置し、 phenotype_descriptionに関連し、log
expressionが5より高い遺伝子のみを含んでいなければならない。
ヒント:このデータフレームを 、どのようにコマンドを並べるべきか考えてみよう!
R
rna %>%
mutate(expression = log(expression)) %>%
select(gene, chromosome_name, phenotype_description, sample, expression) %>%
filter(chromosome_name == "X" | chromosome_name == "Y") %>%
filter(!is.na(phenotype_description)) %>%
filter(expression > 5)
出力
# A tibble: 649 × 5
gene chromosome_name phenotype_description sample expression
<chr> <chr> <chr> <chr> <dbl>
1 Plp1 X abnormal CNS glial cell morphology GSM25… 10.7
2 Slc7a3 X decreased body length GSM25… 5.46
3 Plxnb3 X abnormal coat appearance GSM25… 6.58
4 Rbm3 X abnormal liver morphology GSM25… 9.32
5 Cfp X abnormal cardiovascular system phys… GSM25… 6.18
6 Ebp X abnormal embryonic erythrocyte morp… GSM25… 6.68
7 Cd99l2 X abnormal cellular extravasation GSM25… 8.04
8 Piga X abnormal brain development GSM25… 6.06
9 Pim2 X decreased T cell proliferation GSM25… 7.11
10 Itm2a X no abnormal phenotype detected GSM25… 7.48
# ℹ 639 more rows分割-適用-結合データ分析
多くのデータ分析タスクは、
_split-apply-combine_パラダイムを使ってアプローチすることができる:データをグループに分割し、各グループにいくつかの
分析を適用し、その結果を組み合わせる。
**dplyr** はgroup_by()`
関数を使って、これを非常に簡単にしている。
**dplyr\*\* はgroup_by()`
関数を使って、これを非常に簡単にしている。
R
rna %>%
group_by(gene)
出力
# A tibble: 32,428 × 19
# Groups: gene [1,474]
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 1170 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 Apod GSM254… 36194 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
3 Cyp2d22 GSM254… 4060 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
4 Klk6 GSM254… 287 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
5 Fcrls GSM254… 85 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
6 Slc2a4 GSM254… 782 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
7 Exd2 GSM254… 1619 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 Gjc2 GSM254… 288 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
9 Plp1 GSM254… 43217 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
10 Gnb4 GSM254… 1071 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>group_by()関数はデータ処理を行わず、 データをサブセットにグループ化する。上の例では、 32428オブザベーションの最初のtibbleは、1474グループにgene\`
変数に基づいて分割される。
同様に、ティブルをサンプルごとにグループ分けすることもできる:
R
rna %>%
group_by(sample)
出力
# A tibble: 32,428 × 19
# Groups: sample [22]
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 1170 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 Apod GSM254… 36194 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
3 Cyp2d22 GSM254… 4060 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
4 Klk6 GSM254… 287 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
5 Fcrls GSM254… 85 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
6 Slc2a4 GSM254… 782 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
7 Exd2 GSM254… 1619 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 Gjc2 GSM254… 288 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
9 Plp1 GSM254… 43217 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
10 Gnb4 GSM254… 1071 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>ここで、最初の 32428 オブザベーションの tibble
は、sample 変数に基づいて、 22 グループに分割される。
いったんデータがグループ化されると、その後の操作は各グループに独立して 。
summarise()`関数
group_by()はsummarise()\` と一緒に使われることが多く、
は各グループを1行の要約に折りたたむ。
group_by()は、 **カテゴリー** 変数を含むカラム名を引数として取り、 統計のサマリーを計算します。 group_by()\
は、 カテゴリー 変数を含むカラム名を引数として取り、
統計のサマリーを計算します。
そこで、遺伝子ごとの平均「発現」を計算する:
エラー
Error: <text>:4:0: unexpected end of input
2: group_by(gene %>%
3: summarise(mean_expression = mean(expression))
^また、各サンプルの全遺伝子の平均発現量を計算することもできる:
エラー
Error: <text>:4:0: unexpected end of input
2: group_by(sample %>%
3: summarise(mean_expression = mean(expression))
^しかし、複数の列でグループ化することもできる:
R
rna %>%
group_by(gene, infection, time) %>%
summarise(mean_expression = mean(expression))
出力
`summarise()` has grouped output by 'gene', 'infection'. You can override using
the `.groups` argument.出力
# A tibble: 4,422 × 4
# Groups: gene, infection [2,948]
gene infection time mean_expression
<chr> <chr> <dbl> <dbl>
1 AI504432 InfluenzaA 4 1104.
2 AI504432 InfluenzaA 8 1014
3 AI504432 NonInfected 0 1034.
4 AW046200 InfluenzaA 4 152.
5 AW046200 InfluenzaA 8 81
6 AW046200 NonInfected 0 155.
7 AW551984 InfluenzaA 4 302.
8 AW551984 InfluenzaA 8 342.
9 AW551984 NonInfected 0 238
10 Aamp InfluenzaA 4 4870
# ℹ 4,412 more rowsいったんデータがグループ化されると、同じ (必ずしも同じ変数でなくてもよい)時間に複数の変数を要約することもできる。 例えば、遺伝子別、条件別の「発現」の中央値を示す 列を追加することができる: 例えば、遺伝子別、条件別の「発現」の中央値を示す 列を追加することができる:
R
rna %>%
group_by(gene, infection, time) %>%
summarise(mean_expression = mean(expression),
median_expression = median(expression))
出力
`summarise()` has grouped output by 'gene', 'infection'. You can override using
the `.groups` argument.出力
# A tibble: 4,422 × 5
# Groups: gene, infection [2,948]
gene infection time mean_expression median_expression
<chr> <chr> <dbl> <dbl> <dbl>
1 AI504432 InfluenzaA 4 1104. 1094.
2 AI504432 InfluenzaA 8 1014 985
3 AI504432 NonInfected 0 1034. 1016
4 AW046200 InfluenzaA 4 152. 144.
5 AW046200 InfluenzaA 8 81 82
6 AW046200 NonInfected 0 155. 163
7 AW551984 InfluenzaA 4 302. 245
8 AW551984 InfluenzaA 8 342. 287
9 AW551984 NonInfected 0 238 265
10 Aamp InfluenzaA 4 4870 4708
# ℹ 4,412 more rowsR
rna %>%
filter(gene == "Dok3") %>%
group_by(time) %>%
summarise(mean = mean(expression))
出力
# A tibble: 3 × 2
time mean
<dbl> <dbl>
1 0 169
2 4 156.
3 8 61 カウント
データで作業するとき、我々はしばしば、各因子または因子の組み合わせについて
見つかったオブザベーションの数を知りたい。
このタスクのために、**dplyr** はcount()` を提供している。
例えば、感染したサンプルと感染していないサンプルそれぞれについて、
、データの行数をカウントしたい場合、次のようにする:
R
rna %>%
count(infection)
出力
# A tibble: 2 × 2
infection n
<chr> <int>
1 InfluenzaA 22110
2 NonInfected 10318count()関数は、すでに見たことのある、変数でグループ化し、そのグループ内のオブザベーションの数をカウントして要約する、ということの省略記法です。 言い換えれば、rna
%>% count(infection)`は次のものと等価である:
言い換えれば、rna %>% count(infection)\は次のものと等価である:
R
rna %>%
group_by(infection) %>%
summarise(n = n())
出力
# A tibble: 2 × 2
infection n
<chr> <int>
1 InfluenzaA 22110
2 NonInfected 10318先ほどの例では、count()
を使って、_1つの_要因(つまり感染)について
、行数/観察数を数えている。 先ほどの例では、count()
を使って、_1つの_要因(つまり感染)について
、行数/観察数を数えている。
もし、感染と時間のような_要因の組み合わせ_をカウントしたいのであれば、
、count()の引数として1つ目と2つ目の要因を指定することになる:
R
rna %>%
count(infection, time)
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 InfluenzaA 4 11792
2 InfluenzaA 8 10318
3 NonInfected 0 10318これと等価である:
R
rna %>%
group_by(infection, time) %>%
summarise(n = n())
出力
`summarise()` has grouped output by 'infection'. You can override using the
`.groups` argument.出力
# A tibble: 3 × 3
# Groups: infection [2]
infection time n
<chr> <dbl> <int>
1 InfluenzaA 4 11792
2 InfluenzaA 8 10318
3 NonInfected 0 10318比較を容易にするために、結果を並べ替えると便利なことがある。 arrange()`を使って表を並べ替えることができる。 例えば、上の表を時間順に並べたいとする:
R
rna %>%
count(infection, time) %>%
arrange(time)
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 NonInfected 0 10318
2 InfluenzaA 4 11792
3 InfluenzaA 8 10318あるいは回数で:
R
rna %>%
count(infection, time) %>%
arrange(n)
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 InfluenzaA 8 10318
2 NonInfected 0 10318
3 InfluenzaA 4 11792降順にソートするには、desc()関数を追加する必要がある:
R
rna %>%
count(infection, time) %>%
arrange(desc(n))
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 InfluenzaA 4 11792
2 InfluenzaA 8 10318
3 NonInfected 0 10318R
##
rna %>%
count(sample)
## 2.
rna %>%
group_by(sample) %>%
summary(seq_depth = sum(expression)) %>%
arrange(desc(seq_depth))
## 3.
rna %>%
filter(sample == "GSM2545336") %>%
count(gene_biotype) %>%
arrange(desc(n))
## 4.
rna %>%
filter(phenotype_description == "abnormal DNA methylation") %>%
group_by(gene, time) %>%
summary(mean_expression = mean(log(expression)) %>%
arrange()エラー
Error: <text>:20:0: unexpected end of input
18: summary(mean_expression = mean(log(expression)) %>%
19: arrange()
^データの再構築
rnatibble の行には、geneとsample\`
という2つの変数の組み合わせに関連付けられた発現値(単位)が格納されている。
比較を容易にするために、結果を並べ替えると便利なことがある。 arrange()\`を使って表を並べ替えることができる。 例えば、上の表を時間順に並べたいとする: または遺伝子(gene_biotype, ENTREZ_ID, product, …)。 その他の列はすべて、 (生物、年齢、性別、…)のいずれかを記述する変数に対応している。 または遺伝子(gene_biotype, ENTREZ_ID, product, …)。 遺伝子やサンプルによって変化しない変数は、すべての行で同じ値を持つ。
R
rna %>%
arrange(gene)
出力
# A tibble: 32,428 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 AI504432 GSM25… 1230 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 AI504432 GSM25… 1085 Mus mus… 8 Fema… NonInfec… C57BL… 0 Cereb…
3 AI504432 GSM25… 969 Mus mus… 8 Fema… NonInfec… C57BL… 0 Cereb…
4 AI504432 GSM25… 1284 Mus mus… 8 Fema… Influenz… C57BL… 4 Cereb…
5 AI504432 GSM25… 966 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
6 AI504432 GSM25… 918 Mus mus… 8 Male Influenz… C57BL… 8 Cereb…
7 AI504432 GSM25… 985 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 AI504432 GSM25… 972 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
9 AI504432 GSM25… 1000 Mus mus… 8 Fema… Influenz… C57BL… 4 Cereb…
10 AI504432 GSM25… 816 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>この構造はlong-formatと呼ばれ、1つのカラムにはすべての値、
、もう1つのカラムには値のコンテキストが列挙されている。
場合によっては、long-formatは実際には
“human-readable”ではなく、別のフォーマット、
wide-formatがよりコンパクトにデータを表現する方法として好まれる。
これは通常、科学者が
、行が遺伝子、列がサンプルを表す行列として見るのに慣れている遺伝子発現値の場合である。
これは通常、科学者が
、行が遺伝子、列がサンプルを表す行列として見るのに慣れている遺伝子発現値の場合である。
このフォーマットでは、 、サンプル内の遺伝子発現レベルとサンプル間の遺伝子発現レベル の関係を調べることができる。
出力
# A tibble: 1,474 × 23
gene GSM2545336 GSM2545337 GSM2545338 GSM2545339 GSM2545340 GSM2545341
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Asl 1170 361 400 586 626 988
2 Apod 36194 10347 9173 10620 13021 29594
3 Cyp2d22 4060 1616 1603 1901 2171 3349
4 Klk6 287 629 641 578 448 195
5 Fcrls 85 233 244 237 180 38
6 Slc2a4 782 231 248 265 313 786
7 Exd2 1619 2288 2235 2513 2366 1359
8 Gjc2 288 595 568 551 310 146
9 Plp1 43217 101241 96534 58354 53126 27173
10 Gnb4 1071 1791 1867 1430 1355 798
# ℹ 1,464 more rows
# ℹ 16 more variables: GSM2545342 <dbl>, GSM2545343 <dbl>, GSM2545344 <dbl>,
# GSM2545345 <dbl>, GSM2545346 <dbl>, GSM2545347 <dbl>, GSM2545348 <dbl>,
# GSM2545349 <dbl>, GSM2545350 <dbl>, GSM2545351 <dbl>, GSM2545352 <dbl>,
# GSM2545353 <dbl>, GSM2545354 <dbl>, GSM2545362 <dbl>, GSM2545363 <dbl>,
# GSM2545380 <dbl>rnaの遺伝子発現値をワイドフォーマットに変換するには、 、sample\`カラムの値が
、カラム変数の名前になる新しいテーブルを作成する必要がある。
ここでの重要なポイントは、我々はまだ 、整然としたデータ構造に従っているが、 、興味のある観察に従ってデータを整形したということである:遺伝子ごと、サンプルごとに記録する代わりに、遺伝子ごとの発現レベル 。
逆の変換は、列名を新しい変数の値( )に変換することである。
pivot_longer() と pivot_wider() の2つの
tidyr 関数を使って、これらの変換を行うことができます( こちら
を参照してください)( )。
より広いフォーマットへのデータのピボット
rnaの最初の3列を選択し、pivot_wider()\`
を使ってデータをワイドフォーマットに変換してみよう。
R
rna_exp <- rna %>%
select(gene, sample, expression)
rna_exp
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Cyp2d22 GSM2545336 4060
4 Klk6 GSM2545336 287
5 Fcrls GSM2545336 85
6 Slc2a4 GSM2545336 782
7 Exd2 GSM2545336 1619
8 Gjc2 GSM2545336 288
9 Plp1 GSM2545336 43217
10 Gnb4 GSM2545336 1071
# ℹ 32,418 more rowspivot_wider\`は主に3つの引数を取る:
- 変換されるデータ;
- the
names_from: その値が新しいカラム の名前になるカラム; - value_from\`: 新しいカラム を埋める値。
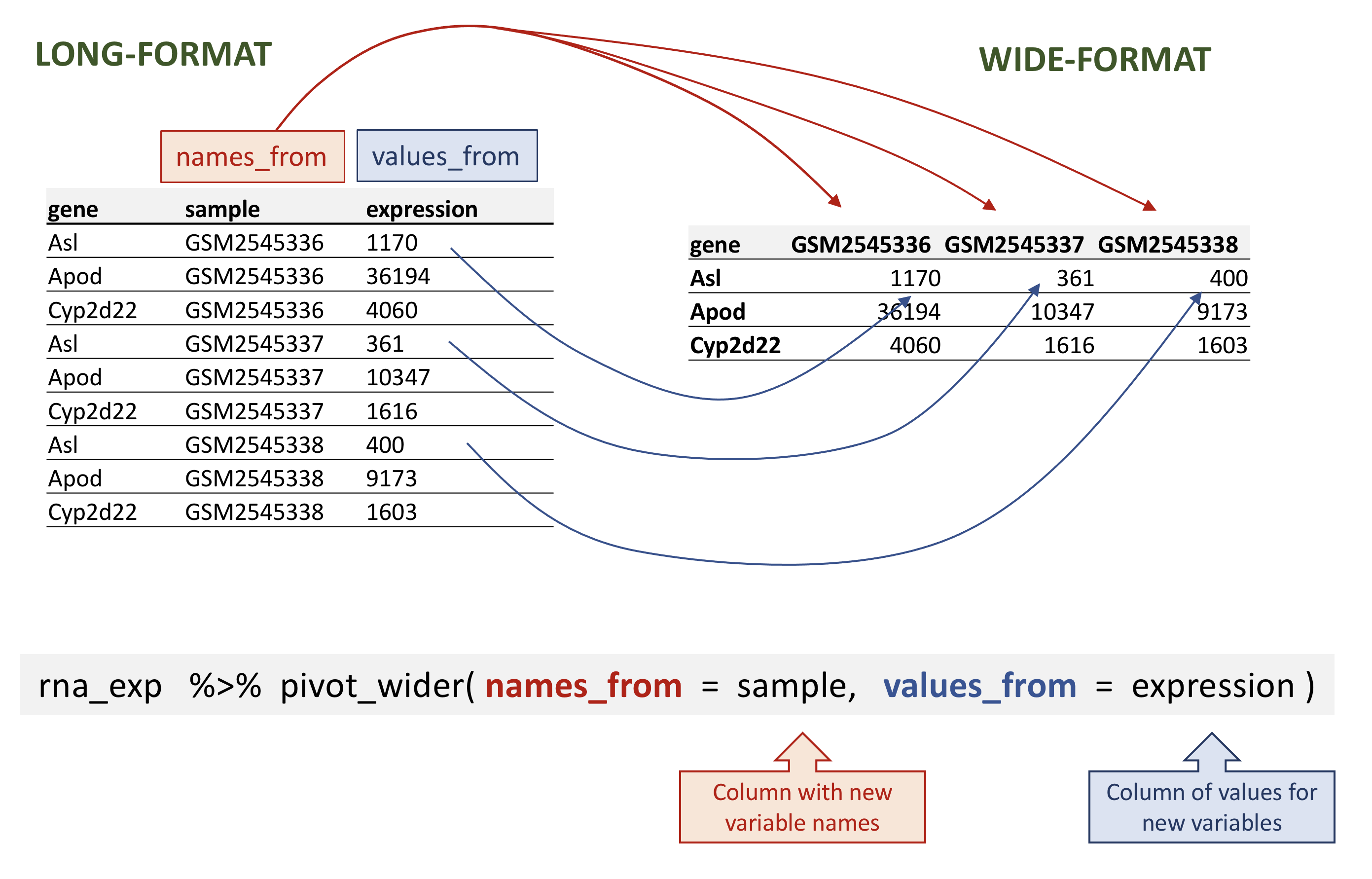
rna data.
R
rna_wide <- rna_exp %>%
pivot_wider(names_from = sample,
values_from = expression)
rna_wide
出力
# A tibble: 1,474 × 23
gene GSM2545336 GSM2545337 GSM2545338 GSM2545339 GSM2545340 GSM2545341
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Asl 1170 361 400 586 626 988
2 Apod 36194 10347 9173 10620 13021 29594
3 Cyp2d22 4060 1616 1603 1901 2171 3349
4 Klk6 287 629 641 578 448 195
5 Fcrls 85 233 244 237 180 38
6 Slc2a4 782 231 248 265 313 786
7 Exd2 1619 2288 2235 2513 2366 1359
8 Gjc2 288 595 568 551 310 146
9 Plp1 43217 101241 96534 58354 53126 27173
10 Gnb4 1071 1791 1867 1430 1355 798
# ℹ 1,464 more rows
# ℹ 16 more variables: GSM2545342 <dbl>, GSM2545343 <dbl>, GSM2545344 <dbl>,
# GSM2545345 <dbl>, GSM2545346 <dbl>, GSM2545347 <dbl>, GSM2545348 <dbl>,
# GSM2545349 <dbl>, GSM2545350 <dbl>, GSM2545351 <dbl>, GSM2545352 <dbl>,
# GSM2545353 <dbl>, GSM2545354 <dbl>, GSM2545362 <dbl>, GSM2545363 <dbl>,
# GSM2545380 <dbl>デフォルトでは、pivot_wider() 関数は欠損値に対して
NA を追加することに注意してください。
何らかの理由で、あるサンプルで 、いくつかの遺伝子の発現値が欠落していたとしよう。 何らかの理由で、あるサンプルで 、いくつかの遺伝子の発現値が欠落していたとしよう。 以下の架空の例では、遺伝子Cyp2d22の発現値はGSM2545338サンプルの 。
R
rna_with_missing_values <- rna %>%
select(gene, sample, expression) %>%
filter(gene %in% c("Asl", "Apod", "Cyp2d22")) %>%
filter(sample %in% c("GSM2545336", "GSM2545337", "GSM2545338")) %>%
arrange(sample) %>%
filter(!(gene == "Cyp2d22" & sample != "GSM2545338"))
rna_with_missing_values
出力
# A tibble: 7 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Asl GSM2545337 361
4 Apod GSM2545337 10347
5 Asl GSM2545338 400
6 Apod GSM2545338 9173
7 Cyp2d22 GSM2545338 1603デフォルトでは、pivot_wider()関数は、
の値が見つからない場合に NA を追加する。 これは、
pivot_wider() 関数の values_fill
引数でパラメータ化できる。 summarise()\`関数
R
rna_with_missing_values %>%
pivot_wider(names_from = sample,
values_from = expression)
出力
# A tibble: 3 × 4
gene GSM2545336 GSM2545337 GSM2545338
<chr> <dbl> <dbl> <dbl>
1 Asl 1170 361 400
2 Apod 36194 10347 9173
3 Cyp2d22 NA NA 1603R
rna_with_missing_values %>%
pivot_wider(names_from = sample,
values_from = expression,
values_fill = 0)
出力
# A tibble: 3 × 4
gene GSM2545336 GSM2545337 GSM2545338
<chr> <dbl> <dbl> <dbl>
1 Asl 1170 361 400
2 Apod 36194 10347 9173
3 Cyp2d22 0 0 1603データを長いフォーマットにピボットする
逆の状況では、カラム名を使い、 、新しい変数のペアに変えている。 一方の変数はカラム名を の値で表し、もう一方の変数にはカラム名に関連付けられている以前の値 が格納されている。 一方の変数はカラム名を の値で表し、もう一方の変数にはカラム名に関連付けられている以前の値 が格納されている。
pivot_longer()\`は主に4つの引数を取る:
- 変換されるデータ;
- names_to\`: の現在のカラム名で作成したい新しいカラム名;
- value_to\`: 作成したい新しいカラム名で、 の現在の値を格納する;
- 変数
names_toとvalues_toに格納する(または削除する)列の名前。

rnaデータのロングピボット。
rna_wideからrna_longを再作成するには、sampleというキーとexpressionという値を作成し、gene以外のすべてのカラム 。ここでは、gene`カラム
をマイナス記号で削除する。
ここで、新しい変数名がどのように引用されるかに注目してください。
R
rna_long<- rna_wide %>%
pivot_longer(names_to = "sample",
values_to = "expression",
-gene)
rna_long
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Asl GSM2545339 586
5 Asl GSM2545340 626
6 Asl GSM2545341 988
7 Asl GSM2545342 836
8 Asl GSM2545343 535
9 Asl GSM2545344 586
10 Asl GSM2545345 597
# ℹ 32,418 more rowsまた、 、どのようなカラムを含めるかという指定も使えたはずだ。
これは、 のカラムが多数あり、
のままにしておくよりも、何を集めるかを指定する方が簡単な場合に便利である。
また、 、どのようなカラムを含めるかという指定も使えたはずだ。 これは、
のカラムが多数あり、
のままにしておくよりも、何を集めるかを指定する方が簡単な場合に便利である。
ここで、starts_with()関数を使えば、
のサンプル名をすべてリストアップすることなく取得することができる!
もう一つの可能性は : 演算子を使うことである!
もう一つの可能性は : 演算子を使うことである!
R
rna_wide %>%
pivot_longer(names_to = "sample",
values_to = "expression",
cols = starts_with("GSM"))
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Asl GSM2545339 586
5 Asl GSM2545340 626
6 Asl GSM2545341 988
7 Asl GSM2545342 836
8 Asl GSM2545343 535
9 Asl GSM2545344 586
10 Asl GSM2545345 597
# ℹ 32,418 more rowsR
rna_wide %>%
pivot_longer(names_to = "sample",
values_to = "expression",
GSM2545336:GSM2545380)
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Asl GSM2545339 586
5 Asl GSM2545340 626
6 Asl GSM2545341 988
7 Asl GSM2545342 836
8 Asl GSM2545343 535
9 Asl GSM2545344 586
10 Asl GSM2545345 597
# ℹ 32,418 more rowsワイドフォーマットで欠損値があった場合、新しいロングフォーマットではNAが
。
前回の欠損値を含む架空のティブルを思い出してほしい:
R
rna_with_missing_values
出力
# A tibble: 7 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Asl GSM2545337 361
4 Apod GSM2545337 10347
5 Asl GSM2545338 400
6 Apod GSM2545338 9173
7 Cyp2d22 GSM2545338 1603R
wide_with_NA<- rna_with_missing_values %>%
pivot_wider(names_from = sample,
values_from = expression)
wide_with_NA
出力
# A tibble: 3 × 4
gene GSM2545336 GSM2545337 GSM2545338
<chr> <dbl> <dbl> <dbl>
1 Asl 1170 361 400
2 Apod 36194 10347 9173
3 Cyp2d22 NA NA 1603R
wide_with_NA %>%
pivot_longer(names_to = "sample",
values_to = "expression",
-gene)
出力
# A tibble: 9 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Apod GSM2545336 36194
5 Apod GSM2545337 10347
6 Apod GSM2545338 9173
7 Cyp2d22 GSM2545336 NA
8 Cyp2d22 GSM2545337 NA
9 Cyp2d22 GSM2545338 1603より幅の広い、より長いフォーマットへの移行は、データセットのバランスをとるのに有効な方法である。 、どの複製も同じ構成になる。
R
rna1<- rna %>%
select(gene, mouse, expression) %>%
pivot_wider(names_from = mouse, values_from = expression)
rna1
出力
# A tibble: 1,474 × 23
gene `14` `9` `10` `15` `18` `6` `5` `11` `22` `13` `23`
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Asl 1170 361 400 586 626 988 836 535 586 597 938
2 Apod 36194 10347 9173 10620 13021 29594 24959 13668 13230 15868 27769
3 Cyp2d22 4060 1616 1603 1901 2171 3349 3122 2008 2254 2277 2985
4 Klk6 287 629 641 578 448 195 186 1101 537 567 327
5 Fcrls 85 233 244 237 180 38 68 375 199 177 89
6 Slc2a4 782 231 248 265 313 786 528 249 266 357 654
7 Exd2 1619 2288 2235 2513 2366 1359 1474 3126 2379 2173 1531
8 Gjc2 288 595 568 551 310 146 186 791 454 370 240
9 Plp1 43217 101241 96534 58354 53126 27173 28728 98658 61356 61647 38019
10 Gnb4 1071 1791 1867 1430 1355 798 806 2437 1394 1554 960
# ℹ 1,464 more rows
# ℹ 11 more variables: `24` <dbl>, `8` <dbl>, `7` <dbl>, `1` <dbl>, `16` <dbl>,
# `21` <dbl>, `4` <dbl>, `2` <dbl>, `20` <dbl>, `12` <dbl>, `19` <dbl>R
rna1 %>%
pivot_longer(names_to = "mouse_id", values_to = "counts", -gene)
出力
# A tibble: 32,428 × 3
gene mouse_id counts
<chr> <chr> <dbl>
1 Asl 14 1170
2 Asl 9 361
3 Asl 10 400
4 Asl 15 586
5 Asl 18 626
6 Asl 6 988
7 Asl 5 836
8 Asl 11 535
9 Asl 22 586
10 Asl 13 597
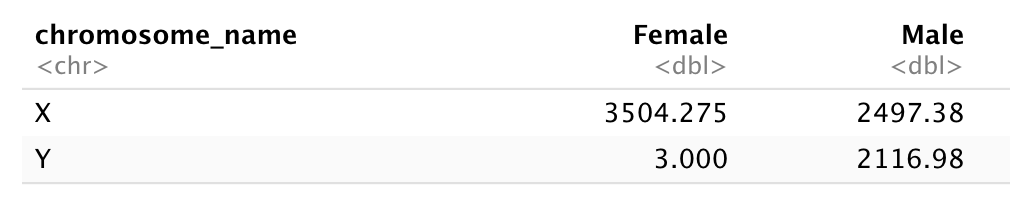
# ℹ 32,418 more rowsまず、 男性と女性のサンプルから、XとYの連鎖遺伝子の平均発現量を計算してみよう…
R
rna %>%
filter(chromosome_name == "Y" | chromosome_name == "X") %>%
group_by(sex, chromosome_name) %>%
summise(mean = mean(expression))
エラー
Error in summise(., mean = mean(expression)): could not find function "summise"そして、表をワイドフォーマットにピボットする
R
rna_1<- rna %>%
filter(chromosome_name == "Y" | chromosome_name == "X") %>%
group_by(sex, chromosome_name) %>%
summise(mean = mean(expression)) %>%
pivot_wider(names_from = sex,
values_from = mean)
エラー
Error in summise(., mean = mean(expression)): could not find function "summise"R
rna_1
エラー
Error in eval(expr, envir, enclos): object 'rna_1' not found各行が一意な chromosome_name と gender
の組み合わせになるように、このデータフレームを
pivot_longer() で変換する。
R
rna_1 %>%
pivot_longer(names_to = "gender",
values_to = "mean",
-chromosome_name)
エラー
Error in eval(expr, envir, enclos): object 'rna_1' not foundまず、遺伝子別、時間別の平均発現量を計算してみよう。
R
rna %>%
group_by(gene, time) %>%
summarise(mean_exp = mean(expression))
出力
`summarise()` has grouped output by 'gene'. You can override using the
`.groups` argument.出力
# A tibble: 4,422 × 3
# Groups: gene [1,474]
gene time mean_exp
<chr> <dbl> <dbl>
1 AI504432 0 1034.
2 AI504432 4 1104.
3 AI504432 8 1014
4 AW046200 0 155.
5 AW046200 4 152.
6 AW046200 8 81
7 AW551984 0 238
8 AW551984 4 302.
9 AW551984 8 342.
10 Aamp 0 4603.
# ℹ 4,412 more rowspivot_wider()関数を使用する前に
R
rna_time<- rna %>%
group_by(gene, time) %>%
summarise(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp)
出力
`summarise()` has grouped output by 'gene'. You can override using the
`.groups` argument.R
rna_time
出力
# A tibble: 1,474 × 4
# Groups: gene [1,474]
gene `0` `4` `8`
<chr> <dbl> <dbl> <dbl>
1 AI504432 1034. 1104. 1014
2 AW046200 155. 152. 81
3 AW551984 238 302. 342.
4 Aamp 4603. 4870 4763.
5 Abca12 5.29 4.25 4.14
6 Abcc8 2576. 2609. 2292.
7 Abhd14a 591. 547. 432.
8 Abi2 4881. 4903. 4945.
9 Abi3bp 1175. 1061. 762.
10 Abl2 2170. 2078. 2131.
# ℹ 1,464 more rowsこれにより、数字で始まるカラム名を持つティブルが生成されることに注意。 これにより、数字で始まるカラム名を持つティブルが生成されることに注意。 タイムポイントに対応するカラムを選択したい場合、 、カラム名を直接使うことはできない。 列4を選択するとどうなるか? 列4を選択するとどうなるか?
R
rna %>%
group_by(gene, time) %>%
summary(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp) %>%
select(gene, 4)
エラー
Error in UseMethod("pivot_wider"): no applicable method for 'pivot_wider' applied to an object of class "table"タイムポイント4を選択するには、“˶\`” というバックスティックを付けたカラム名を引用しなければならない。
R
rna %>%
group_by(gene, time) %>%
summarise(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp) %>%
select(gene, `4`)
出力
`summarise()` has grouped output by 'gene'. You can override using the
`.groups` argument.出力
# A tibble: 1,474 × 2
# Groups: gene [1,474]
gene `4`
<chr> <dbl>
1 AI504432 1104.
2 AW046200 152.
3 AW551984 302.
4 Aamp 4870
5 Abca12 4.25
6 Abcc8 2609.
7 Abhd14a 547.
8 Abi2 4903.
9 Abi3bp 1061.
10 Abl2 2078.
# ℹ 1,464 more rows、数字で始まらない名前を選択する:
R
rna %>%
group_by(gene, time) %>%
summary(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp) %>%
rename("time0" = `0`, "time4" = `4`, "time8" = `8`) %>%
select(gene, time4)
エラー
Error in UseMethod("pivot_wider"): no applicable method for 'pivot_wider' applied to an object of class "table"rna_time tibbleから開始する:
R
rna_time
出力
# A tibble: 1,474 × 4
# Groups: gene [1,474]
gene `0` `4` `8`
<chr> <dbl> <dbl> <dbl>
1 AI504432 1034. 1104. 1014
2 AW046200 155. 152. 81
3 AW551984 238 302. 342.
4 Aamp 4603. 4870 4763.
5 Abca12 5.29 4.25 4.14
6 Abcc8 2576. 2609. 2292.
7 Abhd14a 591. 547. 432.
8 Abi2 4881. 4903. 4945.
9 Abi3bp 1175. 1061. 762.
10 Abl2 2170. 2078. 2131.
# ℹ 1,464 more rowsフォールドチェンジを計算する:
R
rna_time %>%
mutate(time_8_vs_0 = `8` / `0`, time_8_vs_4 = `8` / `4`)
出力
# A tibble: 1,474 × 6
# Groups: gene [1,474]
gene `0` `4` `8` time_8_vs_0 time_8_vs_4
<chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 AI504432 1034. 1104. 1014 0.981 0.918
2 AW046200 155. 152. 81 0.522 0.532
3 AW551984 238 302. 342. 1.44 1.13
4 Aamp 4603. 4870 4763. 1.03 0.978
5 Abca12 5.29 4.25 4.14 0.784 0.975
6 Abcc8 2576. 2609. 2292. 0.889 0.878
7 Abhd14a 591. 547. 432. 0.731 0.791
8 Abi2 4881. 4903. 4945. 1.01 1.01
9 Abi3bp 1175. 1061. 762. 0.649 0.719
10 Abl2 2170. 2078. 2131. 0.982 1.03
# ℹ 1,464 more rowsそして、pivot_longer()関数を使用する:
R
rna_time %>%
mutate(time_8_vs_0 = `8` / `0`, time_8_vs_4 = `8` / `4`) %>%
pivot_longer(names_to = "comparisons",
values_to = "Fold_changes",
time_8_vs_0:time_8_vs_4)
出力
# A tibble: 2,948 × 6
# Groups: gene [1,474]
gene `0` `4` `8` comparisons Fold_changes
<chr> <dbl> <dbl> <dbl> <chr> <dbl>
1 AI504432 1034. 1104. 1014 time_8_vs_0 0.981
2 AI504432 1034. 1104. 1014 time_8_vs_4 0.918
3 AW046200 155. 152. 81 time_8_vs_0 0.522
4 AW046200 155. 152. 81 time_8_vs_4 0.532
5 AW551984 238 302. 342. time_8_vs_0 1.44
6 AW551984 238 302. 342. time_8_vs_4 1.13
7 Aamp 4603. 4870 4763. time_8_vs_0 1.03
8 Aamp 4603. 4870 4763. time_8_vs_4 0.978
9 Abca12 5.29 4.25 4.14 time_8_vs_0 0.784
10 Abca12 5.29 4.25 4.14 time_8_vs_4 0.975
# ℹ 2,938 more rowsテーブルの結合
実生活の多くの場面で、データは複数のテーブルにまたがっている。 通常このようなことが起こるのは、異なる情報源から異なるタイプの情報が 収集されるからである。 通常このようなことが起こるのは、異なる情報源から異なるタイプの情報が 収集されるからである。
分析によっては、2つ以上のテーブル( )のデータを、すべてのテーブルに共通するカラム( )に基づいて1つのデータフレームにまとめることが望ましい場合がある。
dplyr\` パッケージは、指定されたカラム内のマッチに基づいて、2つの データフレームを結合するための結合関数のセットを提供する。 ここでは、 、結合について簡単に紹介する。 詳しくは、 テーブル ジョインの章を参照されたい。 データ変換チート シート 、テーブル結合に関する簡単な概要も提供している。 ここでは、 、結合について簡単に紹介する。 詳しくは、 テーブル ジョインの章を参照されたい。 データ変換チート シート 、テーブル結合に関する簡単な概要も提供している。
、元のrnaテーブルをサブセットして作成し、
、3つのカラムと10行だけを残す。
R
rna_mini<- rna %>%
select(gene, sample, expression) %>%
head(10)
rna_mini
出力
# A tibble: 10 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Cyp2d22 GSM2545336 4060
4 Klk6 GSM2545336 287
5 Fcrls GSM2545336 85
6 Slc2a4 GSM2545336 782
7 Exd2 GSM2545336 1619
8 Gjc2 GSM2545336 288
9 Plp1 GSM2545336 43217
10 Gnb4 GSM2545336 10712番目のテーブルannot1には、遺伝子と
gene_descriptionの2つのカラムがある。
2番目のテーブルannot1には、遺伝子と
gene_descriptionの2つのカラムがある。 download
annot1.csv
リンクをクリックしてdata/フォルダに移動するか、
以下のRコードを使って直接フォルダにダウンロードすることができる。
R
download.file(url = "https://carpentries-incubator.github.io/bioc-intro/data/annot1.csv",
destfile = "data/annot1.csv")
annot1 <- read_csv(file = "data/annot1.csv")
annot1
出力
# A tibble: 10 × 2
gene gene_description
<chr> <chr>
1 Cyp2d22 cytochrome P450, family 2, subfamily d, polypeptide 22 [Source:MGI S…
2 Klk6 kallikrein related-peptidase 6 [Source:MGI Symbol;Acc:MGI:1343166]
3 Fcrls Fc receptor-like S, scavenger receptor [Source:MGI Symbol;Acc:MGI:19…
4 Plp1 proteolipid protein (myelin) 1 [Source:MGI Symbol;Acc:MGI:97623]
5 Exd2 exonuclease 3'-5' domain containing 2 [Source:MGI Symbol;Acc:MGI:192…
6 Apod apolipoprotein D [Source:MGI Symbol;Acc:MGI:88056]
7 Gnb4 guanine nucleotide binding protein (G protein), beta 4 [Source:MGI S…
8 Slc2a4 solute carrier family 2 (facilitated glucose transporter), member 4 …
9 Asl argininosuccinate lyase [Source:MGI Symbol;Acc:MGI:88084]
10 Gjc2 gap junction protein, gamma 2 [Source:MGI Symbol;Acc:MGI:2153060] ここで、dplyr パッケージの full_join()
関数を使用して、これら2つのテーブルを、すべての
変数を含む1つのテーブルに結合したいと思います。
関数は、最初のテーブルと2番目のテーブルの列
に一致する共通変数を自動的に見つける。
この場合、geneは共通の 。 このような変数をキーと呼ぶ。
キーは、
オブザベーションを異なるテーブル間でマッチさせるために使用される。
関数は、最初のテーブルと2番目のテーブルの列
に一致する共通変数を自動的に見つける。
この場合、geneは共通の 。 このような変数をキーと呼ぶ。
キーは、
オブザベーションを異なるテーブル間でマッチさせるために使用される。
R
full_join(rna_mini, annot1)
出力
Joining with `by = join_by(gene)`出力
# A tibble: 10 × 4
gene sample expression gene_description
<chr> <chr> <dbl> <chr>
1 Asl GSM2545336 1170 argininosuccinate lyase [Source:MGI Symbol;Acc…
2 Apod GSM2545336 36194 apolipoprotein D [Source:MGI Symbol;Acc:MGI:88…
3 Cyp2d22 GSM2545336 4060 cytochrome P450, family 2, subfamily d, polype…
4 Klk6 GSM2545336 287 kallikrein related-peptidase 6 [Source:MGI Sym…
5 Fcrls GSM2545336 85 Fc receptor-like S, scavenger receptor [Source…
6 Slc2a4 GSM2545336 782 solute carrier family 2 (facilitated glucose t…
7 Exd2 GSM2545336 1619 exonuclease 3'-5' domain containing 2 [Source:…
8 Gjc2 GSM2545336 288 gap junction protein, gamma 2 [Source:MGI Symb…
9 Plp1 GSM2545336 43217 proteolipid protein (myelin) 1 [Source:MGI Sym…
10 Gnb4 GSM2545336 1071 guanine nucleotide binding protein (G protein)…実生活では、遺伝子アノテーションのラベルが異なることがある。
annot2テーブルは、遺伝子名を含む 変数のラベルが異なる以外は、annot1と全く同じである。 この場合も、 [download annot2.csv](https://carpentries-incubator.github.io/bioc-intro/data/annot2.csv) 、自分でdata/`に移動するか、以下のRコードを使う。
この場合も、 download
annot2.csv
、自分でdata/\に移動するか、以下のRコードを使う。
R
download.file(url = "https://carpentries-incubator.github.io/bioc-intro/data/annot2.csv",
destfile = "data/annot2.csv")
annot2 <- read_csv(file = "data/annot2.csv")
annot2
出力
# A tibble: 10 × 2
external_gene_name description
<chr> <chr>
1 Cyp2d22 cytochrome P450, family 2, subfamily d, polypeptide 22 [S…
2 Klk6 kallikrein related-peptidase 6 [Source:MGI Symbol;Acc:MGI…
3 Fcrls Fc receptor-like S, scavenger receptor [Source:MGI Symbol…
4 Plp1 proteolipid protein (myelin) 1 [Source:MGI Symbol;Acc:MGI…
5 Exd2 exonuclease 3'-5' domain containing 2 [Source:MGI Symbol;…
6 Apod apolipoprotein D [Source:MGI Symbol;Acc:MGI:88056]
7 Gnb4 guanine nucleotide binding protein (G protein), beta 4 [S…
8 Slc2a4 solute carrier family 2 (facilitated glucose transporter)…
9 Asl argininosuccinate lyase [Source:MGI Symbol;Acc:MGI:88084]
10 Gjc2 gap junction protein, gamma 2 [Source:MGI Symbol;Acc:MGI:…どの変数名も一致しない場合、マッチングに使用する
変数を手動で設定することができる。
どの変数名も一致しない場合、マッチングに使用する
変数を手動で設定することができる。 これらの変数は、rna_mini
と annot2 テーブルを使用して以下に示すように、
by 引数を使用して設定することができる。
R
full_join(rna_mini, annot2, by = c("gene" = "external_gene_name"))
出力
# A tibble: 10 × 4
gene sample expression description
<chr> <chr> <dbl> <chr>
1 Asl GSM2545336 1170 argininosuccinate lyase [Source:MGI Symbol;Acc…
2 Apod GSM2545336 36194 apolipoprotein D [Source:MGI Symbol;Acc:MGI:88…
3 Cyp2d22 GSM2545336 4060 cytochrome P450, family 2, subfamily d, polype…
4 Klk6 GSM2545336 287 kallikrein related-peptidase 6 [Source:MGI Sym…
5 Fcrls GSM2545336 85 Fc receptor-like S, scavenger receptor [Source…
6 Slc2a4 GSM2545336 782 solute carrier family 2 (facilitated glucose t…
7 Exd2 GSM2545336 1619 exonuclease 3'-5' domain containing 2 [Source:…
8 Gjc2 GSM2545336 288 gap junction protein, gamma 2 [Source:MGI Symb…
9 Plp1 GSM2545336 43217 proteolipid protein (myelin) 1 [Source:MGI Sym…
10 Gnb4 GSM2545336 1071 guanine nucleotide binding protein (G protein)…上で見たように、最初のテーブルの変数名は、結合されたテーブルでも 。
チャレンジだ:
こちら
をクリックして annot3
テーブルをダウンロードし、そのテーブルをあなたの data/
リポジトリに置いてください。
full_join()関数を使用して、テーブルrna_miniとannot3`
を結合する。
、遺伝子_Klk6_、mt-Tf、mt-Rnr1、mt-Tv、mt-Rnr2、mt-Tl1_はどうなったのか?
full_join()関数を使用して、テーブルrna_miniとannot3`
を結合する。
、遺伝子_Klk6、mt-Tf、mt-Rnr1、mt-Tv、mt-Rnr2、_mt-Tl1_はどうなったのか?
R
annot3 <- read_csv("data/annot3.csv")
full_join(rna_mini, annot3)
出力
# A tibble: 15 × 4
gene sample expression gene_description
<chr> <chr> <dbl> <chr>
1 Asl GSM2545336 1170 argininosuccinate lyase [Source:MGI Symbol;Acc…
2 Apod GSM2545336 36194 apolipoprotein D [Source:MGI Symbol;Acc:MGI:88…
3 Cyp2d22 GSM2545336 4060 cytochrome P450, family 2, subfamily d, polype…
4 Klk6 GSM2545336 287 <NA>
5 Fcrls GSM2545336 85 Fc receptor-like S, scavenger receptor [Source…
6 Slc2a4 GSM2545336 782 solute carrier family 2 (facilitated glucose t…
7 Exd2 GSM2545336 1619 exonuclease 3'-5' domain containing 2 [Source:…
8 Gjc2 GSM2545336 288 gap junction protein, gamma 2 [Source:MGI Symb…
9 Plp1 GSM2545336 43217 proteolipid protein (myelin) 1 [Source:MGI Sym…
10 Gnb4 GSM2545336 1071 guanine nucleotide binding protein (G protein)…
11 mt-Tf <NA> NA mitochondrially encoded tRNA phenylalanine [So…
12 mt-Rnr1 <NA> NA mitochondrially encoded 12S rRNA [Source:MGI S…
13 mt-Tv <NA> NA mitochondrially encoded tRNA valine [Source:MG…
14 mt-Rnr2 <NA> NA mitochondrially encoded 16S rRNA [Source:MGI S…
15 mt-Tl1 <NA> NA mitochondrially encoded tRNA leucine 1 [Source…遺伝子_Klk6_はrna_miniにのみ存在し、遺伝子_mt-Tf_、mt-Rnr1、mt-Tv、
mt-Rnr2、_mt-Tl1_はannot3テーブルにのみ存在する。
表の 変数のそれぞれの値は、欠損として符号化されている。 表の
変数のそれぞれの値は、欠損として符号化されている。
データのエクスポート
dplyr\`を使って、 から情報を抽出したり、生データを要約したりする方法を学んだので、これらの新しいデータセットをエクスポートして、 を共同研究者と共有したり、アーカイブしたりしたいと思うかもしれない。
RにCSVファイルを読み込むために使用される read_csv()
関数と同様に、 、データフレームからCSVファイルを生成する
write_csv() 関数があります。
write_csv()を使う前に、生成されたデータセットを格納する新しいフォルダdata_outputを作業ディレクトリに作成する。 、生成されたデータセットを生データと同じディレクトリに書き込みたくない。 別々にするのは良い習慣だ。 dataフォルダーには、
、変更されていない生のデータだけを入れておく。
、削除したり変更したりしないように、そのままにしておく。
対照的に、このスクリプトはdata_output
ディレクトリの内容を生成するので、そこに含まれるファイルが削除されても、
再生成することができる。
、生成されたデータセットを生データと同じディレクトリに書き込みたくない。
別々にするのは良い習慣だ。
dataフォルダーには、 、変更されていない生のデータだけを入れておく。 、削除したり変更したりしないように、そのままにしておく。 対照的に、このスクリプトはdata_output`
ディレクトリの内容を生成するので、そこに含まれるファイルが削除されても、
再生成することができる。
write_csv()\`を使用して、以前に作成したrna_wideテーブルを保存しよう。
R
write_csv(rna_wide, file = "data_output/rna_wide.csv")