Content from スプレッドシートを使用したデータ整理
最終更新日:2024-09-08 | ページの編集
所要時間: 60分
概要
質問
- 表形式のデータを整理するにはどうすればよいですか?
目的
- スプレッドシートとその長所と短所について学びます。
- データを効果的に使用するには、スプレッドシート内のデータをどのようにフォーマットすればよいでしょうか?
- 一般的なスプレッドシートのエラーとその修正方法について説明します。
- きちんとしたデータの原則に従ってデータを整理します。
- カンマ区切り (CSV) 形式やタブ区切り (TSV) 形式などのテキストベースのスプレッドシート形式について説明します。
This episode is based on the Data Carpentries’s Data Analysis and Visualisation in R for Ecologists lesson.
表計算プログラム
質問
- 優れたデータ統合用にスプレッドシートを使用するための基本的な原則は何でしょうか?
客観的
- コンピューターがデータセットを に活用できるようにデータを整理するためのベスト プラクティスについて説明します。
キーポイント
- 適切なデータ構成は、あらゆる研究プロジェクトの基礎です。
適切なデータ構成は、研究 の基礎です。 ほとんどの研究者はデータを持っているか、 シートにデータ入力を行っていません。 スプレッドシート プログラムは、データ テーブルを設計し、非常に基本的 データ品質管理機能を処理するための非常に便利な インターフェイスです。 @Broman:2018 も参照してください。
スプレッドシートの概要
スプレッドシートはデータ入力に適しています。 したがって、スプレッドシートにはデータ がたくさんあります。 研究者としての時間の多くは、この「データの検討」段階 費やされることになります。 とても楽しいわけではありませんが、必要性は です。 データの編成について考える方法と、より効果的なデータ ラングリングのための かの実践方法を説明します。
このレッスンで教えられないこと
- スプレッドシートで 統計 を行う方法
- スプレッドシートで プロット を行う方法
- スプレッドシート プログラムで _コードを記述する_方法
これを実行したい場合は、O 発行の Head First Excel 参考になります。 「ライリー。
なぜスプレッドシートでのデータ分析を教えないのか
スプレッドシートでのデータ分析には通常、多くの 作業が必要です。 パラメーターを変更したり、 データセットを使用して分析を実行したりする場合は、通常、すべてを手動でやり直す必要があります。 (マクロを作成できることはわかりませ が、次の点を参照してください。)
また、 の作業に戻りたいときや、誰かが分析の詳細を尋ねたときに、スプレッドシート プログラムで行われた統計分析やプロット分析を追跡したり再現したりすること 困難です。
多くの表計算プログラムが利用可能です。 ほとんどの参加者は主なスプレッドシート プログラムとして を使用するため、このレッスンで Excel の例を使用します。 で使用できる表計算プログラムは LibreOffice です。 コマンドはプログラム間 少し異なる場合がありますが、一般的な考え方は同じです。
スプレッドシート プログラムには、研究者としてできる にする必要のある多くのことが含まれています。 それらは次の目的で使用できます。
- データ入力
- データの整理
- データのサブセット化と並べ替え
- 統計
- プロット
スプレッドシート プログラムはテーブルを使用してデータを表し、表示します。 テーブルとしてフォーマットされたデータ この章の主要テーマであり、効率的なダウンストリーム分析を にするために、標準化された方法でデータをテーブルに編成する方法 見ていきます。
スプレッドシートの問題
スプレッドシートはデータ入力には適していますが、実際にはデータ入力以外の目的で シート プログラムを使用する傾向があります。 これら を使用して、出版物のデータ テーブルを作成し、概要 統計を生成し、図を作成します。
スプレッドシートでパブリケーション用のテーブルを生成することは ではありません。多くの場合、パブリケーション用にデータ テーブルをフォーマットするとき、実際にはデータとして読み取ら ことを意図していない方法で重要な概要統計をレポートすることに 、特殊なフォーマットが必要になることがよくあります。 (セルを結合し、境界線を作成し、美しくする)。 この種の操作は文書編集ソフトウェア内で行う をお勧めします。
統計と数値を生成する後者の 2 つのアプリケーションは、 して使用する必要があります。1 スプレッド プログラムのグラフィカルなドラッグ アンド ドロップの性質のため、 手順を複製するのが不可能ではないにしても、非常に困難になる可能性があります (元に戻すことはさらに困難です)。特に の統計や数値により、より複雑な計算が必要な場合はそうです。 さらに、スプレッドシートで計算を行う場合、 わずかに異なる数式を の隣接するセルに誤って適用してしまうことがよくあります。 R や SAS などのコマンドライン ベースの統計プログラムを使用する場合、意図的に実行しない限り、データセット内の観測値には計算を適用し、別の観測値には計算を適用しないことは事実上不可能です。
スプレッドシートでのデータテーブルの書式設定
質問
- データを効果的に使用するには、スプレッドシート内のデータをどのようにフォーマットすればよいでしょうか?
目的
シートでのデータ入力と書式設定のベスト プラクティスについて説明します。
ベスト プラクティスを適用して、変数と観測値を シートに配置します。
キーポイント
生データは決して変更しないでください。 を加える前に必ずコピーを作成してください。
データをクリーンアップするために実行したすべての手順を テキスト ファイルに記録します。
きちんとしたデータの原則に従ってデータを整理します。
最もよくある間違いは、スプレッドシート プログラムを研究室の ブックのように扱うことです。つまり、情報を伝えるためにコンテキスト、余白のメモ、データとフィールドの空間 レイアウトに依存していることです。 人間として、これらのことを (通常は) 解釈できますが、コンピューターは情報 同じようには見ません。そして、すべての の意味をコンピューターに説明しない限り (それ 難しい場合があります!)、理解できません。 データがどのように組み合わされるかを確認できます。
コンピューターの力を利用すると、 効果的かつ高速な方法でデータを管理および分析できますが、その力を使用するには、コンピューターが理解できるようにデータを する必要があります ( コンピューターは非常に複雑です)。リテラル)。
このため、 の予備実験からデータの入力を開始する前に、適切にフォーマットされた をセットアップすることが非常に重要です。 データの整理は研究プロジェクトの です。 分析全体を通じて の操作が容易になるか困難になる可能性があるため、データ入力を行う や実験を設定するときに考慮する価値があります。 スプレッドシートではさまざまな方法で設定できますが、これらの 選択の一部によっては、 のプログラムでデータを操作する能力が制限されたり、6 か月後の自分や共同作業者が共同作業したりすることが制限される可能性があります。 データ。
注: データ入力とデータ分析に最適なレイアウト/形式 (およびソフトウェアと ) は異なる場合があります。 これを考慮し、理想的にはあるものから のものへの変換を自動化することが です。
分析を追跡する
スプレッドシートを使用しているとき、データのクリーンアップ 分析を行っているときに、最初のスプレッドシートとは 異なる外観のスプレッドシートが完成することがよくあります。 分析を したり、査読者や講師が別の分析を要求したときに何をしたかを把握したりするには、次のこと 行う必要があります。
クリーンアップまたは分析されたデータを含む新しいファイルを作成します。 元のデータセット 変更しないでください。変更すると、どこから始めたのかわからなくなります。
クリーンアップまたは分析で実行した手順を記録します。 実験の他のステップと同様に、これらのステップを追跡する必要があり 。 データ ファイルと同じフォルダーに保存された テキスト ファイルでこれを行うことをお勧め ます。
これはスプレッドシート設定の例です。
今日の演習中にこれらの原則を実践してください。
バージョン管理はこのコースの範囲外ですが、バージョンを維持 方法については、 ‘Git’ の Carpentries レッスンを参照してください。データを制御します。 簡単なチュートリアルについてはこの ブログ 投稿 を、より研究指向のユースケースについては @Perez-Riverol:2016 も参照してください。
スプレッドシートでのデータの構造化
データにスプレッドシート プログラムを使用する際の基本ルールは次のとおりです。
- すべての変数を列に入力します。測定対象は「重量」や「温度」 です。
- 各観測値を独自の行に配置します。
- 1 つのセルに複数の情報を組み合わせないでください。 場合によっては それは単なる つのことのように思えますが、それがそのデータを使用または並べ替えできるようにする唯一の方法であるかどうかを考えてください。
- 生データはそのままにしておきます。変更しないでください。
- クリーンアップされたデータを CSV (カンマ区切り値) 形式などのテキストベースの形式にエクスポートします。 これにより、誰でも を使用できるようになり、ほとんどのデータ リポジトリで必要になります。
たとえば、ベルギーのブリュッセルにあるいくつか 病院を訪れた患者からのデータがあります。 彼らは、訪問日、 、患者の性別、体重、血液型を記録しました。
次のようにデータを追跡するとします。
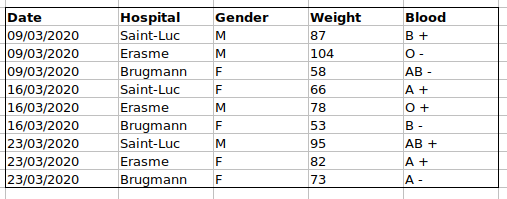
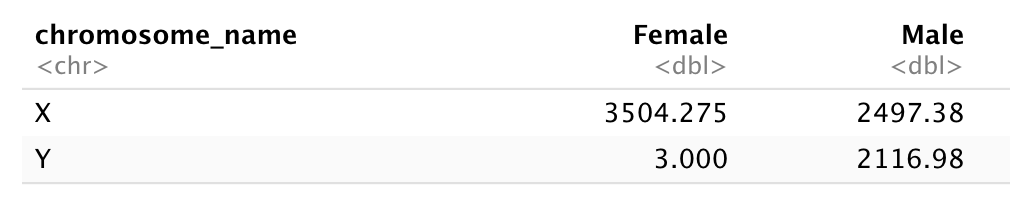
問題は、ABO グループと Rhesus グループが同じ「Blood」 タイプ列にあることです。 したがって、A グループのすべての観測値を調べたり、ABO グループごとの重み分布を調べたりしたい場合、このデータ設定を使用してこれを行うのは難しいでしょう 。 代わりに、ABO グループと Rhesus グループを別の列に配置すると、はるかに簡単になることがわかります。
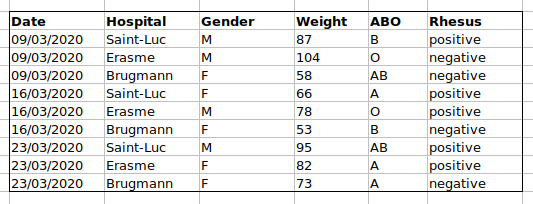
データシートを設定する際の重要なルールは、列は変数に され、行は観測に使用されるということです。
- 列は変数です
- 行は観測結果です
- セルは個別の値です
課題: 乱雑なデータセットを取り上げ、それをクリーンアップする方法を説明します。
ここ をクリックして、乱雑なデータセットをダウンロードします。
スプレッドシート プログラムでデータを開きます。
タブが 2 つあることがわかります。 このデータには、2020 年の新型コロナウイルス感染症 (COVID-19) の第 波と第 2 波の間にブリュッセルのさまざまな病院で記録されたさまざま 臨床変数が含まれています。 ご覧のとおり、 データは 3 月と 11 月 の波では異なる方法で記録されています。 あなたはこのプロジェクトの責任者となり、 データの分析を開始できるようにしたいと考えています。
隣にいる人と一緒に、この スプレッドシートのどこが間違っているのかを特定してください。 また、最初と 2 番目の Wave タブをクリーンアップし、それらをすべて つのスプレッドシートにまとめるために必要な手順について 説明します。
重要: 最初のアドバイスを忘れないでください。クリーンアップされたデータ用に ファイル (またはタブ) を作成する場合は、元の (生の) データを決して変更しないでください。
この演習を終えた後、このデータの が間違っていたのか、そしてそれをどのように修正するのかをグループで話し合います。
特に R スクリプト に関する 優れた参考文献は、Tidy Data 論文 @Wickham:2014 です。
よくあるスプレッドシートのエラー
質問
- スプレッドシート のデータの書式設定に関する一般的な課題は何ですか?また、それらを回避するにはどうすればよいですか?
目的
- 一般的なスプレッドシートの書式設定の問題を認識して解決します。
キーポイント
- 1 つのスプレッドシート内で複数のテーブルを使用しないでください。
- データが複数のタブに分散しないようにします。
- ゼロはゼロとして記録します。
- 欠落データを記録するには、適切な null 値を使用します。
- 情報を伝えたり、スプレッドシートを美しく見せるために書式設定を使用しないでください。
- コメントは別の列に配置します。
- 列ヘッダーに単位を記録します。
- セルには 1 つの情報のみを含めます。
- 列ヘッダーにはスペース、数字、特殊文字を使用しないでください。
- データ内では特殊文字を避けてください。
- メタデータを別のプレーン テキスト ファイルに記録します。
自分自身のデータ でなく、共同作業者やインターネットからのデータにも、注意すべき潜在的なエラーがいくつかあります。 エラーや、下流 データ分析 結果の解釈に悪影響が及ぶ可能性があることを認識 ていれば、自分やプロジェクト メンバーがエラーを回避しようとする動機になるかもしれません。 スプレッドシートでデータをフォーマットする方法に 変更を加えると、データのクリーニング と分析の効率と信頼性に大きな影響 を与える可能性があります。
- 複数のテーブルの使用
- 複数のタブの使用
- ゼロを埋めない
- 問題のある null 値の使用
- 情報を伝えるために書式設定を使用する
- 書式設定を使用してデータシートを美しく見せる
- セル内にコメントまたはユニットを配置する
- セルに複数の情報を入力する
- 問題のあるフィールド名の使用
- データ内での特殊文字の使用
- データテーブルへのメタデータの組み込み
複数のテーブルの使用
一般的な戦略は、1 つ スプレッドシート内に複数のデータ
テーブルを作成することです。
これはコンピュータを混乱させるので、行わないでください。
1
つのスプレッドシート内に複数のテーブルを作成すると、コンピュータにとっては、各行を観測
として認識するため、物事の間に た関連付けが描画されることになります。
また、同じフィールド名を
の場所で使用している可能性があり、データを使用可能な形式に
アップすることが困難になります。 以下の例は問題を示しています。
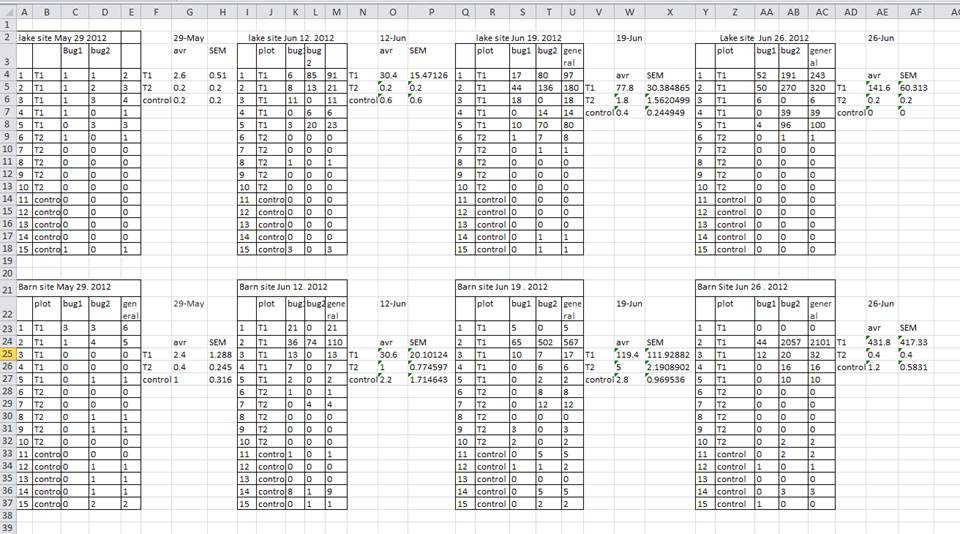
上の例では、コンピュータは、(たとえば) 行 4 と 、すべての列 A ~ A F が同じサンプルを参照しているとみなして表示します。 この行 、実際には 4 つの異なるサンプル (5 月 29 日、6 月 12 日、6 月 19 日、および 6 月 26 日の つの異なる収集日のそれぞれのサンプル 1) と、計算されたいくつかの概要統計 (平均 (avr) およびこれら のサンプルのうち 2 つの標準測定誤差 (SEM))。 他の行にも同様に問題があります。
複数のタブの使用
しかし、ワークブックのタブはどうでしょうか? データを整理する簡単な方法のように思えますよね? まあ、はい、いいえです。 追加のタブを作成すると、そこにあるデータの接続をコンピュータに認識させることができなく ます (この接続を確保するには、 スプレッドシート アプリケーション固有の関数を導入するか、 スクリプトを導入する必要があります)。 たとえば、測定を行う日ごとに のタブを作成するとします。
これは次の 2 つの理由から良い習慣ではありません。
測定を行うたびに新しいタブでデータ の記録を開始すると、誤って データに不一致が追加される可能性が高くなります。
たとえすべての不一致が忍び寄るのを防ぐことができたとしても、 これらのデータを単一 データテーブルに結合する必要があるため、 データを分析する前に余分な手順を追加することになります。 タブ 結合する方法をコンピュータに明示的に指示する必要があります。また、タブの形式が一貫していない場合は、 で結合する必要がある場合もあります。
次回データを入力するときに、別のタブ またはテーブルを作成するときは、元のスプレッドシートに別の列を 追加することで、このタブの追加を回避できるかどうか自問してください。 乱雑なデータ ファイルの では複数のタブを使用しましたが、データを 編成してタブ間で統合する方法がわかりました。
の過程でデータシートが非常に長くなる可能性があります。 これにより、スプレッドシートの上部に ヘッダーが表示されない場合、データの入力が困難になります。 ただし、ヘッダー 行を繰り返さないでください。 これらは簡単にデータに混入し、 的に問題が発生する可能性があります。 代わりに、列 ヘッダーを固定する ことができます。これにより、多くの 行を含むスプレッドシートがある場合でも、それらの行が表示されたままになります。
ゼロを埋めない
何かを測定するとき、調査でウサギが観察された回数は通常 である可能性があります。 その列にはほとんどゼロがあるのに、なぜわざわざ という数字のゼロを書き込むのでしょうか?
ただし、 スプレッドシートのゼロと空白のセルには違いがあります。 コンピューターにとって、ゼロは実際にはデータです。 あなたが測ったか、 なかった。 空白のセルは、測定されていないことを意味し、 はそれを未知の値 (ヌル または欠損値とも呼ばれます) として解釈します。
スプレッドシートや統計プログラムでは、ゼロであるつもりの の空白セルが誤って解釈される可能性があります。 観測値 入力しないことにより、そのデータ を不明または欠落 (null) として表すようにコンピュータに指示することになります。 これにより、後続の の計算または分析で問題が発生する可能性があります。 たとえば、単一の null 値を含む一連の数値 の平均は常に null です ( は欠落している観測値を推測できないため)。 このうち であるため、ゼロをゼロとして記録し、真に 欠損データをヌルとして記録することが非常に重要です。
問題のある null 値 {#null}の使用
例: -999 またはその他の数値 (またはゼロ) を に使用すると、欠損データを表します。
解決策:
データセット内で null 値が異なる で表現される理由はいくつかあります。 紛らわしいヌル値が測定装置から自動的に として記録される場合があります。 その場合、できることは ではありませんが、ツール を使用してデータ クリーニングで対処できます 1 OpenRefine 前分析。 また、データが存在しないさまざまな理由を伝えるために、 な null 値が使用されることもあります。 これは取得すべき重要 情報ですが、実際には 1 つの列を使用して 2 つの情報を取得することになります。 フォーマットを使用して 情報を伝える と同様に、ここでは「data_missing」のような新しい 列を作成し、その列を使用して の異なる理由をキャプチャすると良いでしょう。
理由が何であれ、不明または欠落しているデータが として -999、999、または 0 として記録されている場合は問題です。
多くの統計プログラムは、これらが欠損 (NULL) 値を表す であることを認識しません。 これらの値 がどのように解釈されるかは、データの分析に使用するソフトウェアによって異なります。 明確に定義された一貫性のある null インジケーターを使用することが です。
空白 (ほとんどのアプリケーション) と NA (R の場合) が 選択肢として適しています。 @White:2013 は、記事の中で、さまざまなソフトウェア アプリケーションに対して null 値 を示すための適切な選択肢について説明しています。
フォーマットを使用して情報を伝える
例: 分析から 必要があるセル、行、または列を強調表示し、空白の行を残してデータの 分離を示します。
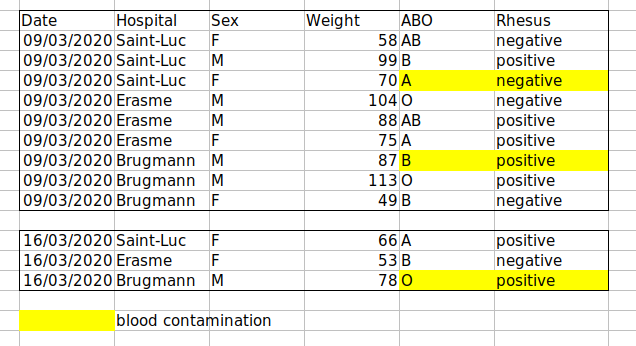
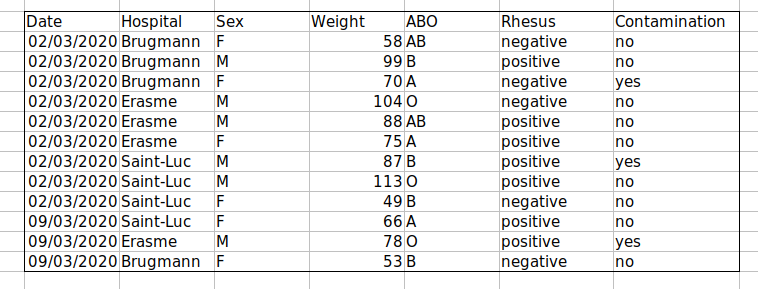
解決策: 新しいフィールドを作成して、 データをエンコードします。
書式設定を使用してデータシートを美しく見せる {#formatting_pretty}
例: セルを結合します。
解決策: 注意しないと、ワークシートを より美しく見えるように書式設定すると、データ内の 関連付けを認識するコンピュータの機能が損なわれる可能性があります。 セルを結合すると、統計ソフトウェアで を読み取ることができなくなります。 データを整理する にセルを結合する必要がないような方法でデータを再構築することを検討してください。
セル {#units}にコメントまたはユニットを配置する
ほとんどの分析ソフトウェアは Excel や LibreOffice のコメントを表示できないため、 データ セル内に配置されたコメントによって混乱する可能性があります。 書式設定について で説明したように、セルに を追加する必要がある場合は、別のフィールドを作成します。 同様に、セルに単位を含めないでください。理想的には、1 つの列に配置する の測定値が同じ 単位内にある必要がありますが、何らかの理由でそうでない場合は、別のフィールドを作成し、 セルの単位を指定します。で。
セル {#info}に複数の情報を入力する
例: A+、 B+、A- などの ABO グループとアカゲザル グループを 1 つのセルに記録する
解決策: セルに複数の情報を含めないでください。
これにより、データを分析できる方法が制限されます。
の場合、これらの測定値の両方が必要な場合は、
この情報を含めるようにデータシートを設計します。 たとえば、ABO
グループには 1 つの列を含め、Rhesus グループには つの列を含めます。
問題のあるフィールド名 {#field_name} の使用
説明的なフィールド名を選択しますが、スペース、 数字、またはいかなる種類の特殊文字も含めないように注意してください。 スペースは、空白を区切り文字として使用するパーサーによって て解釈される可能性があり、一部の プログラムは 数字で始まるテキスト文字列であるフィールド名を好みません。
アンダースコア (_) はスペースの代わりに使用できます。
読みやすさを向上させるために、
名前をキャメルケースで記述することを検討してください (例:
ExampleFileName)。 現時点では意味のある略語 も、6
か月後にはそれほど明確ではなくなる可能性があることに注意してください。ただし、
が長すぎる名前を付けすぎないでください。 フィールド名に
を含めることで混乱が回避され、他の人がフィールドを簡単に解釈できるようになります。
例
| いい名前 | 良い代替品 | 避ける |
|---|---|---|
| 最高_温度_C | 最大温度 | 最高温度 (°C) |
| 降水量_mm | 降水量 | プレcmm |
| 平均_年_成長 | 平均年成長 | 平均成長率/年 |
| sex | セックス | 男/女 |
| weight | 重さ | w。 |
| セル_タイプ | セルタイプ | 細胞の種類 |
| 観察_01 | 最初の_観察 | 1回目の観測 |
データ {#special}での特殊文字の使用
例: たとえば、Word または のアプリケーションからデータを直接コピーするなど、メモを書くときにスプレッドシート プログラムをワード プロセッサ として扱います。
解決策: これは一般的な戦略です。 たとえば、セルに の長いテキストを書き込む場合、スプレッドシートに改行、全角ダッシュ、 などを含めることがよくあります。 また、Word など アプリケーションからデータをコピーする場合、書式設定や派手な 標準文字 (左揃えと右揃えの引用符など) が含まれます。 このデータをコーディング/ 環境またはリレーショナル データベースにエクスポートすると、行が半分に切断されたり、エンコード エラーが発生したりする 、危険なことが発生する可能性があります。
一般的なベスト プラクティスは、改行、 タブ、垂直タブなどの文字の追加を避けることです。 言い換えれば、テキスト セルを、テキストとスペースのみを含めることができる単純な Web フォームで かのように扱います。
データテーブル {#metadata}へのメタデータの組み込み
例: データ テーブル の上部または下部に、列の意味、単位、例外などを説明する凡例を追加します。
解決策: データに関するデータ (「メタデータ」) を記録することは ではありません。 データセットを して分析している間は、データセットと親密な関係にあるかもしれませんが、変数「sglmemgp」がグループの単一のメンバー (たとえば を意味すること、または以前に使用した正確なアルゴリズムを意味することをまだ覚えている可能性は ありません。変数 変換するか、派生変数を作成すると、数か月後、1 年後、またはそれ以上かかります。
また、他の人があなたのデータを調べたり、使用したりする理由はたくさんあります。あなたの発見を理解するため、 を検証するため、 提出された出版物をレビューするため、結果を再現するため、 同様の研究を計画するため、さらには他の人がアクセスしたり 利用できるようにデータをアーカイブします。 デジタルデータは定義上、 可読ではありませんが、その意味を理解することは の仕事です。 研究の収集段階および分析段階でデータを文書化することの重要性は、特に研究が 記録の一部となる場合には 過大評価することはできません 。
ただし、データファイル 自体にはメタデータを含めないでください。 論文や補足ファイルの表とは異なり、メタデータ ( 形式) はデータ ファイルに含めるべきではありません。この情報はデータではなく、 データを含めるとコンピューター プログラムがデータ ファイルを解釈する が混乱する可能性があるためです。 むしろ、メタデータは、データ ファイルと同じディレクトリに別のファイルとして保存する必要があります。できればファイルと明確に関連付けられる名前を付けてプレーン テキスト形式で保存する必要があります。 メタデータ ファイルはフリー テキスト形式であるため、コメント、単位、 値のエンコード方法に関する情報などをエンコードすることも ます。これらの情報は文書化するには重要ですが、データ ファイルの 設定を混乱させる可能性があります。
さらに、ファイルまたはデータベース レベルのメタデータは、データセットを構成するファイルが相互に ように関連するかを記述します。どのような形式であるか。 は、以前のファイルに優先されるか、または以前のファイルによって置き換えられるか。 フォルダー レベルの readme.txt ファイルは、プロジェクト内のすべて ファイルとフォルダーを説明する古典的な方法です。
(メタデータに関するテキストは、EDINA および 大学データ ライブラリによるオンライン コース Research Data MANTRA から改変されました。 MANTRA は クリエイティブ コモンズ 表示 4.0 国際 ライセンス に基づいてライセンスされています。
データのエクスポート
質問
- ストリーム アプリケーションに役立つ方法でスプレッドシートからデータをエクスポートするにはどうすればよいでしょうか?
目的
- スプレッドシート データをユニバーサル ファイル形式で保存します。
- スプレッドシートから CSV ファイルにデータをエクスポートします。
キーポイント
一般的なスプレッドシート形式で保存されたデータは、データ分析ソフトウェアに 読み込まれないことが多く、 にエラーが生じます。
スプレッドシートから CSV や TSV などの形式にデータをエクスポートすると、ほとんどのプログラムで一貫して使用できる形式でデータが になります。
分析に使用するデータを Excel 既定のファイル形式 (Excel
バージョンに応じて *.xls または *.xlsx)
で保存することはお勧めできません。 なぜ?
これは独自の形式であり、 的にはファイルを開くことが不可能では にしても不便になる技術が存在しなくなる (または十分にまれになる) 可能性があるためです。
他の表計算ソフトウェアでは、 の Excel 形式で保存されたファイルを開くことができない場合があります。
Excel のバージョンが異なるとデータの処理方法が異なる場合があり、 整合が発生する可能性があります。 日付 は、データ ストレージにおける不整合の十分に文書化された例です。
最後に、データをデータ リポジトリに することを要求するジャーナルや補助金機関が増えています。また、そのほとんどは Excel 形式を受け入れ ん。 で説明する形式のいずれかである必要があります。
上記の点は、LibreOffice / Open Office で使用されるオープン データ 形式などの他の形式にも当てはまります。 これらの形式は ではなく、 ソフトウェア パッケージによって同じ方法で解析されません。
データを汎用的でオープンな静的形式で保存すると、この問題に するのに役立ちます。 タブ区切り (タブ区切り値または TSV) または カンマ区切り (カンマ区切り値または CSV) を試してください。 CSV ファイルは、列がカンマで区切られたプレーン テキスト ファイルです。したがって、「カンマ で区切られた値」または CSV と呼ばれます。 Excel/SPSS/などと した CSV ファイルの利点ファイルは、TextEdit や などのプレーン テキスト エディタを含む、ほぼすべてのソフトウェア を使用して CSV ファイルを開いて読み取ることができるということです。 CSV ファイル内のデータは、SQLite や R などの他 形式や環境にも簡単にインポートできます。CSV ファイルを使用する場合、特定の高価なプログラムの のバージョンに縛られることがないので、最大限の移植性と 性を実現するために使用するフォーマット。 ほとんどのスプレッドシート プログラムは などの区切りテキスト形式で簡単に保存できますが、ファイルのエクスポート に警告が表示される場合があります。
Excel で開いたファイルを CSV 形式で保存するには:
- 上部のメニューから「ファイル」と「名前を付けて保存」を選択します。
- [形式] フィールドのリストから、[カンマ区切りの 値]
(
*.csv) を選択します。 - ファイル名と する場所を再確認し、「保存」をクリックします。
下位互換性に関する重要な注意: ファイルは Excel で開くことができます。
エラー
Error in loadNamespace(x): there is no package called 'Knitr'R と xls に関するメモ: xls
ファイル (および Google スプレッドシート) を読み取ることができる R
パッケージがあります。 「xls」ドキュメント内の
ワークシートにアクセスすることも可能です。
しかし
- これらの中には Windows でのみ動作するものもあります。
- これは、データ分析 R コードの追加の複雑さ/依存性を
して、(単純だが手動の)
csvへのエクスポートを置き換えることに相当します。 - データ形式のベスト プラクティスは引き続き適用されます。
-
csv(または類似のもの) が では不十分である正当な理由は本当にあるのでしょうか?
カンマに関する注意事項
一部のデータセットでは、データ値自体にカンマ (,) が含まれる場合があります。 その場合、使用しているソフトウェア (Excel を含む) により、列内のデータが誤って表示される可能性が ます。 これは、データ値の一部であるカンマの が 区切り文字として解釈されるためです。
たとえば、データは次のようになります。
種 ID、属、種、分類群
AB、Amphispiza、bilineata、鳥類
AH、Ammospermophilus、harrisi、げっ歯類、国勢調査されていない
AS、Ammodramus、savannarum、鳥類
BA、Baiomys、taylori、げっ歯類レコード「AH,Ammospermophilus,harrisi,Rodent, not censused」では、「taxa」の値 コンマが含まれています (「Rodent, not censused」)。 上記を Excel (または他のスプレッドシート プログラム) に読み込むために を試みると、 のような結果が得られます。
エラー
Error in loadNamespace(x): there is no package called 'Knitr'taxa の値は (1 つの列 D に
を入れる代わりに) 2 つの列に分割されました。 これはさらに多数の
エラーに伝播する可能性があります。
たとえば、追加の列は、欠損値が多数ある (適切なヘッダーがない) 列
として解釈されます。 に加えて、行 3 のレコードの列 D の値
(つまり、‘taxa’ の値にカンマが含まれている の値)
も正しくなくなりました。
データを「csv」形式で保存し、 データ値にカンマが含まれる可能性があることが予想される場合は、値を引用符 (““) で囲むことで、上記 で説明した問題を回避できます。 このルールを適用すると、 データは次のようになります。
種 ID、属、種、分類群
"AB"、"Amphispiza"、"bilineata"、"Bird"
"AH"、"Ammospermophilus"、"harrisi"、"げっ歯類、国勢調査されていない"
"AS"、"Ammodramus" 、"サバンナルム"、"鳥"
"BA"、"バイオミス"、"テイロリ"、"げっ歯類"Excel では 引用符の外側にあるカンマのみが区切り文字として使用されるため、このファイルを Excel で「csv」として開いても、余分な 列は生成されません。
あるいは、カンマを含むデータを操作している場合、 シートで作業するときに別の区切り文字を使用する必要がある可能 があります1。 この場合、区切り文字としてタブを使用し、TSV ファイルを扱う場合は 使用することを検討してください。 TSV ファイルは、CSV ファイルと同じ方法でスプレッドシート プログラムからエクスポートできます。
データ の値が “” に含まれていないものの、区切り文字 とデータ値の一部としてカンマが含まれている既存のデータセットを操作している場合は、データ クリーニングに関する重大な問題 に直面する可能性があります。 扱っているデータセットに 百または数千のレコードが含まれている場合、それらを手動でクリーンアップする ( データ値からカンマを削除するか、値を 引用符 (““) で囲む) と、何時間もかかるだけではありません。ただし、誤って のエラーが発生する可能性があります。
データセットのクリーンアップは、多くの 分野における主要な問題の 1 つです。 このアプローチは、ほとんどの場合、特定 コンテキストに依存します。 ただし、スクリプトを作成して実行するなど、 化された方法でデータをクリーンアップすることをお勧めします。 Python と R のレッスンは、 するスクリプトを構築するためのスキルを開発するための基礎を提供します。
まとめ

典型的なデータ分析ワークフローは、上の図 に示されており、データは繰り返し変換、視覚化、モデル化されます。 この の繰り返しは、データが理解されるまで複数回繰り返されます。 ただし、 の実際のケースでは、実際にデータを分析して理解すること ではなく、データのクリーンアップと準備 にほとんどの時間が費やされます。
変換/視覚化/モデルのサイクルを高速で 回繰り返すアジャイルなデータ分析ワークフローは、データが予測可能な方法で されており、データを調べたり したりすることなく推論できる場合にのみ実現可能です。それ。
これは、カンマが小数点の として使用されるヨーロッパの 諸国に特に関係します。 このような場合、 csv ファイルのデフォルト値の区切り文字はセミコロン (;) になるか、値は体系的に引用符で囲まれた になります。↩︎
Content from RとRStudio
最終更新日:2024-09-08 | ページの編集
所要時間: 30分
概要
質問
- RとRStudioとは何ですか?
目的
- RStudio スクリプト、コンソール、環境、およびプロットペインの目的について説明します。
- Rプロジェクトとして一連の分析のためのファイルとディレクトリを整理し、作業ディレクトリの目的を理解する。
- RStudio 組み込みのヘルプインターフェイスを使用して、R 関数の詳細情報を検索します。
- Rのユーザーコミュニティとトラブルシューティングのために十分な情報を提供する方法を示す。
このエピソードは、Data Carpentriesの_Data Analysis and Visualisation in R for Ecologists_レッスンに基づいています。
Rとは? RStudioとは何ですか?
R](https://www.r-project.org/)という用語は、 プログラミング言語、統計計算_のための_環境 、それを使って書かれたスクリプトを解釈する_ソフトウェア_を指すのに使われる。
RStudioは現在、Rスクリプトを書くだけでなく、R ソフトウェア1と対話するための非常に人気のある方法です。 RStudio を正しく機能させるには、R と が必要です。
RStudio IDE Cheat Sheet](https://raw.githubusercontent.com/rstudio/cheatsheets/main/rstudio-ide.pdf) 、ここで説明するよりもはるかに多くの情報を提供していますが、キーボードショートカットを学んだり、新しい機能を発見したりするのに便利です。
なぜRを学ぶのか?
Rはポインティングやクリックを多用しない。
学習曲線は他のソフトウェアよりも急かもしれないが、 Rを使えば、分析結果は のポインティングとクリックの連続を覚えることに依存するのではなく、代わりに の一連のコマンドを書くことに依存する! そのため、より多くのデータを収集したため、 分析をやり直したい場合、 結果を得るためにどのボタンをどの順番でクリックしたかを覚えておく必要はない。スクリプトを再度実行するだけでよい。
スクリプトを使用することで、分析で使用したステップが明確になり、 、書いたコードを他の誰かが検査することができ、 フィードバックを与え、間違いを発見することができる。
スクリプトを使って仕事をすることで、自分がやっている の内容をより深く理解することになり、自分が使っている メソッドの学習と理解が容易になる。
Rコードは再現性に優れている
再現性とは、同じデータセットから同じ解析コード( )を使ったときに、他の誰か(未来の自分を含む)が 、同じ結果を得られることを意味する。
Rは他のツールと統合し、 のコードから原稿やレポートを作成することができる。 さらにデータを集めたり、データセットの誤りを修正したりすると、原稿や報告書の 図や統計検定が自動的に更新されます。 。
ジャーナルや研究助成機関では、 、再現性のある分析を求めるところが増えている。Rを知っていれば、このような 。
Rは学際的で拡張性がある
機能を拡張するためにインストールできる10000以上のパッケージ2により、Rは、 多くの科学分野からの統計的アプローチを組み合わせることができるフレームワークを提供し、 データの分析に必要な分析フレームワークに最適です。 例えば、 Rには画像分析、GIS、時系列、集団 遺伝学、その他多くのパッケージがある。
, the Comprehensive R Archive Network. From the R Journal, Volume 10/2, December 2018.](../fig/cran.png)
Rはあらゆる形や大きさのデータを扱う
Rで学ぶスキルは、 データセットの大きさに合わせて簡単にスケールアップできる。 データセットの行数が数百行であろうと数百万行であろうと、 、大差はないだろう。
Rはデータ分析用に設計されている。 欠損データや統計的 因子の取り扱いを便利にする特別なデータ構造 とデータ型が付属している。
Rは、スプレッドシート、データベース、その他多くのデータ形式、 、コンピュータ上またはウェブ上に接続することができます。
Rは大きく歓迎されるコミュニティ
何千人もの人々が毎日Rを利用している。 彼らの多くは、メーリングリストやStack Overflowのようなウェブサイト、またはRStudio communityを通じて、 。 こうした広範なユーザー・コミュニティは、 、バイオインフォマティクスのような専門分野にも広がっている。 Rコミュニティのそのようなサブセットの1つが、Bioconductorである。“現在および将来の生物学的アッセイからのデータの”分析と理解のための科学的プロジェクトである。 このワークショップは、Bioconductor コミュニティのメンバーによって開発されました。Bioconductor についての詳細は、関連ワークショップ “The Bioconductor Project” をご覧ください。
RStudioを使いこなす
まずはRStudioについて学んでみよう。 は R を扱うための統合開発環境(IDE)だ。
RStudio IDE オープンソース製品は、Affero General Public License (AGPL) v3の下でフリーです。 RStudio IDE は、Posit, Inc.の商用ライセンスおよび 優先メールサポートでもご利用いただけます。
RStudio IDE を使ってコードを書き、 コンピュータ上のファイルを操作し、これから作成する変数を検査し、 生成するプロットを視覚化する。 RStudioは他にも (例:バージョン管理、パッケージの開発、Shynyアプリの作成)にも使えます。 ワークショップでは取り上げません。
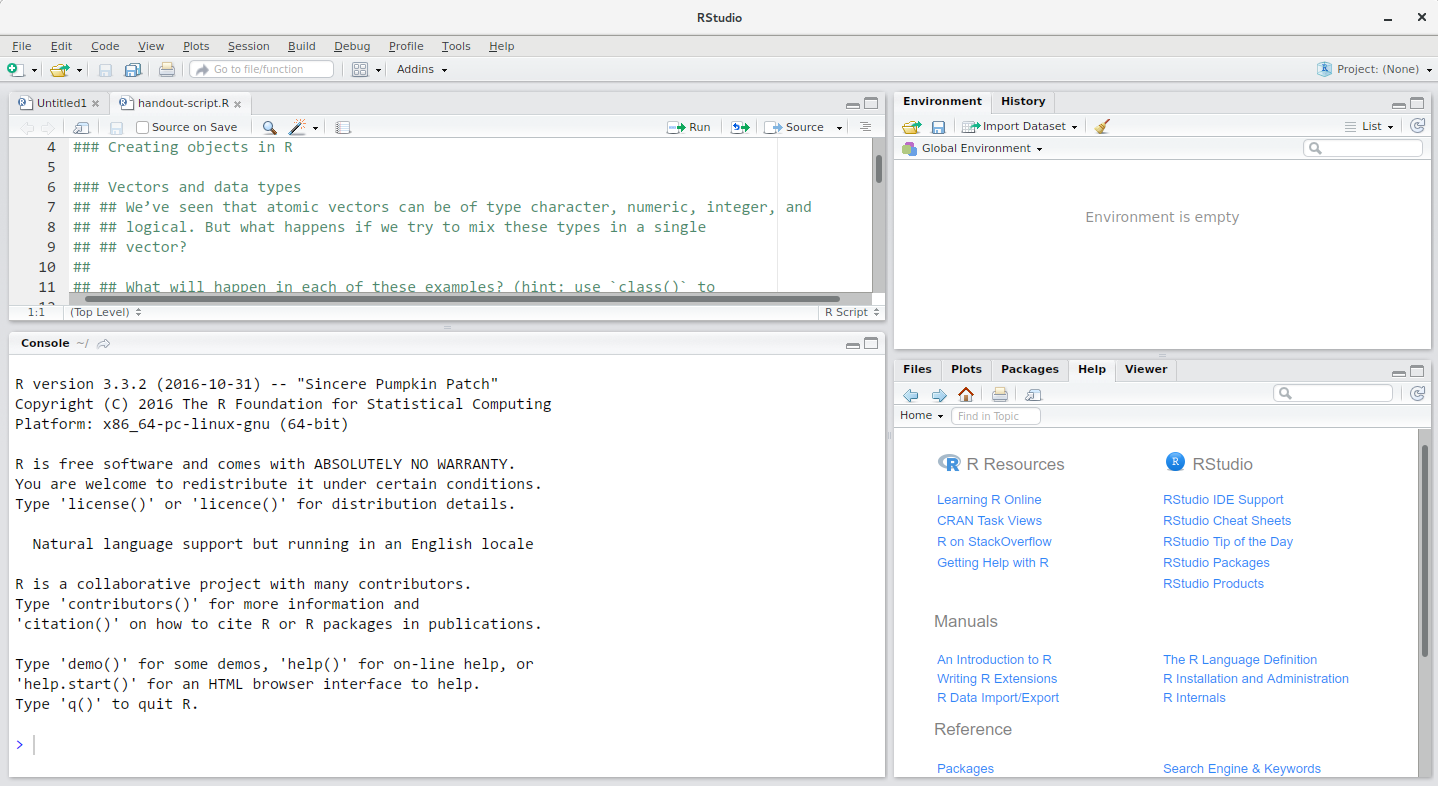
RStudio ウィンドウは 4 つの「ペイン」に分かれています:
- スクリプトとドキュメントの ソース ( のデフォルトレイアウトでは左上)
- あなたの環境/歴史(右上)、
- あなたのFiles/Plots/Packages/Help/Viewer(右下)、そして
- R コンソール(左下)。
これらのペインの配置とその内容はカスタマイズすることができます(
メニューの Tools -> Global Options -> Pane Layout
を参照してください)。
RStudioを使う利点の1つは、コードを書くために必要なすべての情報( )が1つのウィンドウで利用できることです。 さらに、 多くのショートカット、オートコンプリート、およびRでの開発中に使用する主な ファイルタイプのハイライトにより、RStudioは の入力を容易にし、エラーを少なくします。
セットアップ
関連するデータ、分析、テキスト( )のセットは、working directoryと呼ばれる1つのフォルダに自己完結させておくのがよい習慣である。 このフォルダー内のすべてのスクリプトは、 相対パス を使用して、 ファイルがプロジェクト内のどこにあるかを示すことができます( ファイルが特定のコンピューター上のどこにあるかを示す絶対パスとは異なります)。 この方法で作業することで、 、自分のコンピュータ上でプロジェクトを移動したり、 他の人と共有したりすることが、基盤となるスクリプト がまだ動くかどうかを心配することなく、とても簡単になる。
RStudioは、“Projects” インターフェイスを通じて、このような作業を行うための便利なツールセットを提供しています。このツールは、作業ディレクトリを作成するだけでなく、 その場所を記憶し(すぐに移動できるようになります)、 カスタム設定や開いているファイルを保存して、 休憩後に作業を再開しやすくすることもできます。 この チュートリアルのための “Rプロジェクト”の作成手順を以下に示す。
- RStudioを起動します。
- File
メニューの下にあるNew projectをクリックする。 新規ディレクトリを選択し、新規プロジェクトを選択する。 - この新しいフォルダ(または「ディレクトリ」)の名前を入力し、
便利な場所を選択します。 これはこのセッション (またはコース全体) の
作業ディレクトリ になります (例
bioc-intro)。 - Create project`をクリックする。
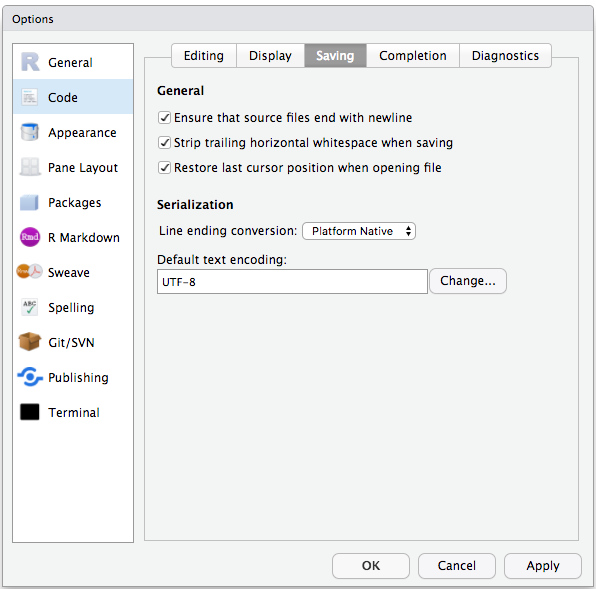
- (オプション)RStudio でワークスペースを保存しない設定にします。
RStudioのデフォルトの環境設定は一般的にうまく機能しますが、ワークスペースを .RDataに保存するのは、特に大きなデータセットを扱う場合は面倒です。 これをオフにするには、「ツール」→「グローバル・オプション」で、終了時に「ワークスペースを.RDataに保存する」 「決してしない」オプションを選択します。

ウィンドウズと他のオペレーティング・システム間の文字エンコーディングの問題](https://yihui.name/en/2018/11/biggest-regret-knitr/)を避けるため、 、デフォルトでUTF-8を設定します:
作業ディレクトリの整理
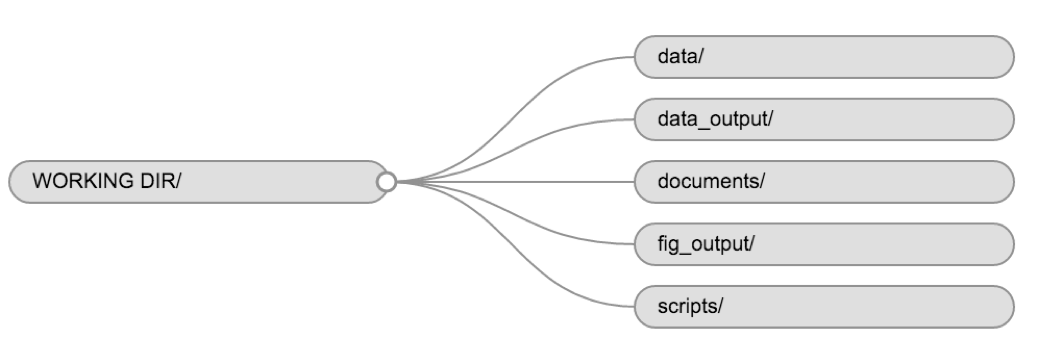
プロジェクト全体で一貫性のあるフォルダ構造を使うことで、 、整理整頓がしやすくなり、将来的に探し出したりファイルしたりするのも簡単になります。 この 、複数のプロジェクトを抱えているときには特に役立つ。 一般的に、 、*スクリプト、*データ、*ドキュメント用のディレクトリ(フォルダ)を作成することができます。
-
data/*
このフォルダは、生データと、特定の分析に必要な中間データセット(
)を保存するために使用します。 透明性と 出所のために、
常に 生データのコピーにアクセスできるようにしておき、
データのクリーンアップと前処理をできるだけプログラム的に(つまり、手作業ではなく
スクリプトで)行うべきである。 生データ
、加工データから切り離すのも良いアイデアだ。 例えば、
data/raw/tree_survey.plot1.txtと...plot2.txtのファイルを、scripts/01.preprocess.tree_survey.Rスクリプトによって生成されたdata/processed/tree.survey.csvファイルとは別に 。 -
documents/ここは、アウトライン、下書き、 、その他のテキストを保管する場所になります。 - **scripts/
** (またはsrc`) この場所には、さまざまな分析やプロット用の R スクリプトを保存し、 関数用の別フォルダを作成することもできます(詳しくは後述します)。
あなたのプロジェクトの必要性に応じて、追加のディレクトリやサブディレクトリが必要になるかもしれないが、これらはあなたの作業用 ディレクトリのバックボーンを形成するはずである。
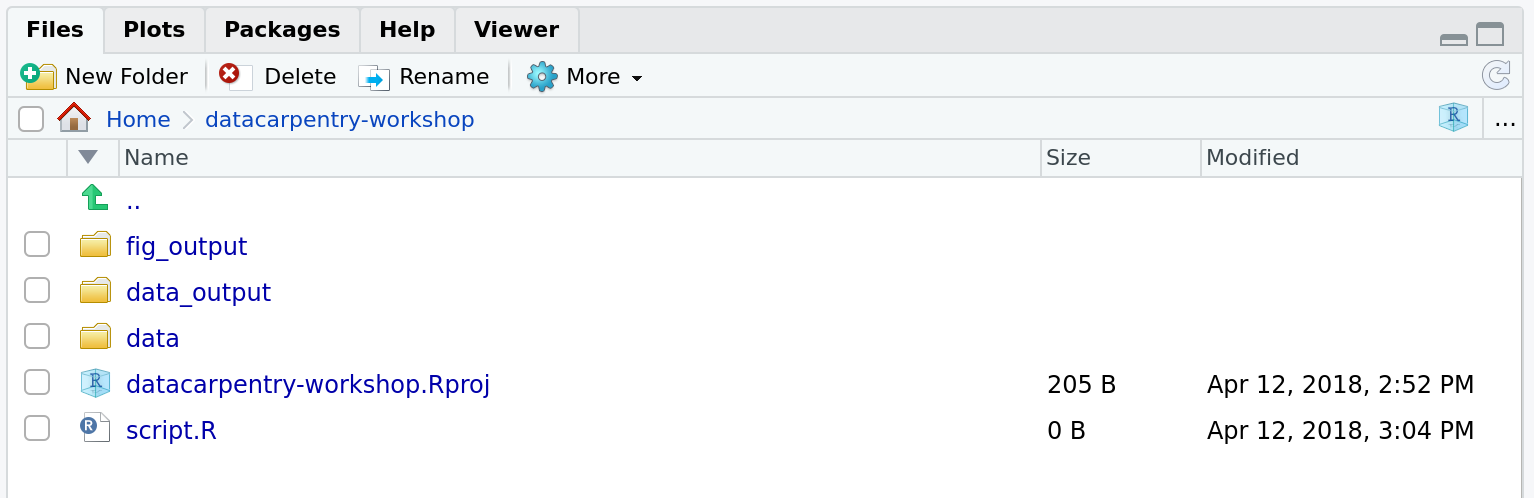
このコースでは、生データを保存するために data/
フォルダが必要です。 、データを CSV
ファイルとしてエクスポートする方法を学ぶために data_output/
フォルダを使用し、図を保存するために fig_output/
フォルダを使用します。
スクリプトは作業ディレクトリのルート に置くことにする。使用するのは1つのファイルだけだし、 事が簡単になるからだ。
作業ディレクトリはこのようになっているはずだ:
プロジェクト管理は、バイオインフォマティクス・プロジェクトにも適用できる。 3。 William Noble (@Noble:2009)は、 以下のディレクトリ構造を提案している:
ディレクトリ名は大きな書体で、ファイル名は小さな 。 ここに掲載したのは、その一部である。 なお、 の日付は、
- - というフォーマットになっているので、 時系列順に並べ替えることができる。 ソースコードsrc/ms-analysis.cがコンパイルされてbin/ms-analysisが作成され、doc/ms-analysis.htmlに文書化されている。 データ・ディレクトリ にあるREADMEファイルには、誰がどの URL から の日付にデータ・ファイルをダウンロードしたかが明記されている。 ドライバスクリプトresults/2009-01-15/runallは自動的に 3つのサブディレクトリ split1、split2、split3 を生成する。 は3つのクロスバリデーション分割に対応する。bin/parse-sqt.pyスクリプトはrunall` ドライバ スクリプトの両方から呼び出される。
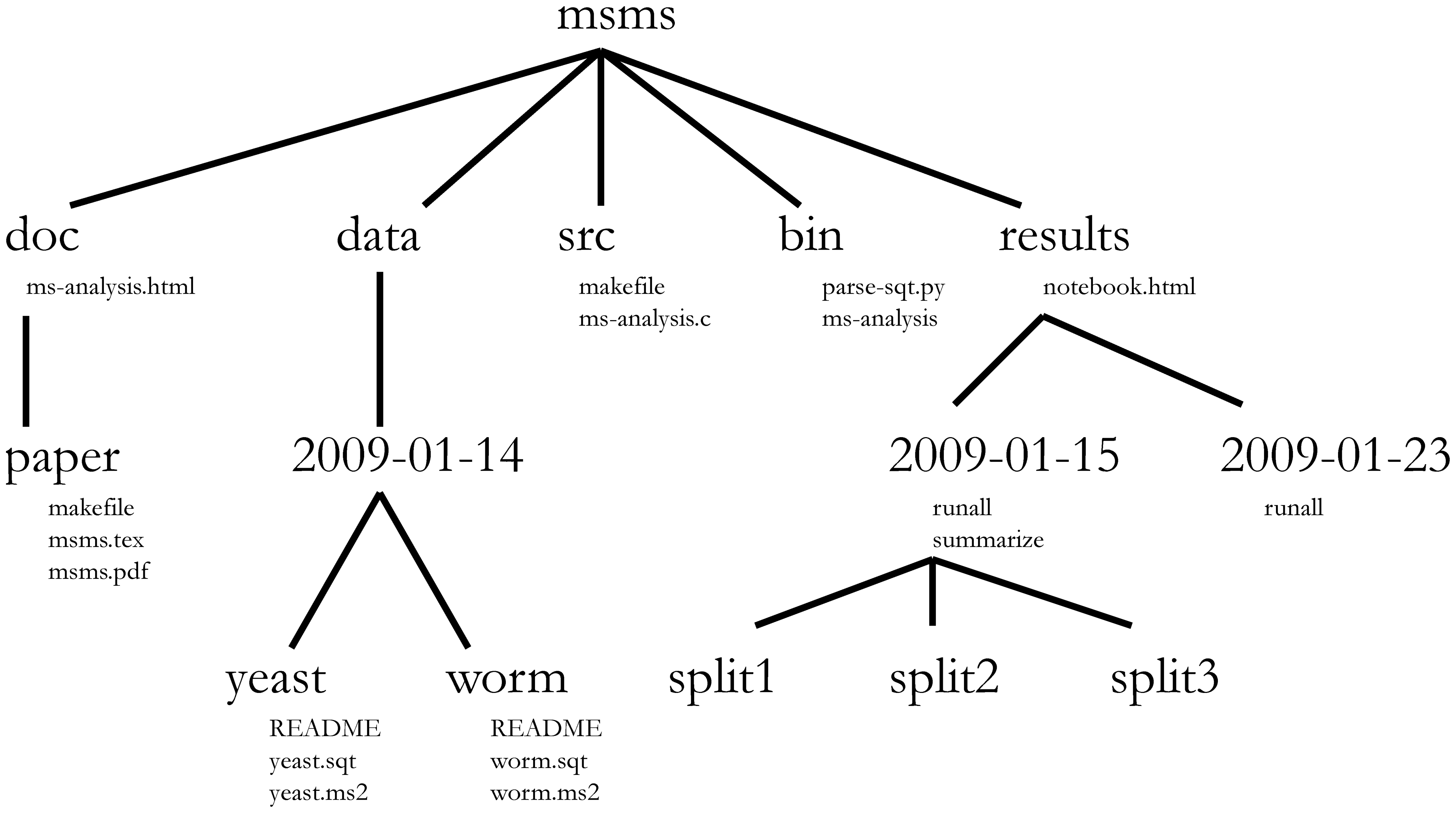
よく定義され、よく文書化された プロジェクトのディレクトリの最も重要な側面は、 プロジェクト4をよく知らない人が、次のことをできるようにすることである。
どのようなプロジェクトなのか、どのようなデータが入手可能なのか、どのような 分析が行われ、どのような結果が出たのかを理解すること、そして最も重要なことは、 を理解することである。
新しいデータで、あるいは 分析パラメーターの一部を変更して、分析を繰り返す。
作業ディレクトリ
作業ディレクトリは理解すべき重要な概念である。 Rがファイルを探して保存する 。 プロジェクトのコードを書くときは、作業ディレクトリのルート に関連するファイルを参照し、この 構造内のファイルだけが必要です。
RStudioプロジェクトを使用すると、この作業が簡単になり、
ディレクトリが適切に設定されます。 もし確認する必要があれば、
getwd() を使うことができる。
何らかの理由で作業ディレクトリが のようになっていない場合は、RStudio
のインターフェイスで
ファイルブラウザーで作業ディレクトリがあるべき場所に移動し、
青い歯車のアイコン More
をクリックし、Set As Working Directory
を選択して変更することができます。
あるいは、setwd("/path/to/working/directory")を使って、
作業ディレクトリをリセットすることもできる。
しかし、あなたのスクリプトには、
、この行を含めるべきではありません。なぜなら、他の誰かのコンピューターで失敗してしまうからです。
**例
以下のスキーマは作業ディレクトリ bioc-intro と
data と fig_output
のサブディレクトリ、そして後者にある2つのファイルを表しています:
bioc-intro/data/
/fig_output/fig1.pdf
/fig_output/fig2.pngもし作業ディレクトリにいれば、相対パス
bioc-intro/fig_output/fig1.pdf か、 絶対パス
/home/user/bioc-intro/fig_output/fig1.pdf を使って
fig1.pdf ファイルを参照することができます。
もし data ディレクトリにいたとしたら、相対パス
../fig_output/fig1.pdf か、同じ絶対パス
/home/user/bioc-intro/fig_output/fig1.pdf
を使うことになる。
Rとの対話
プログラミングの基本は、私たちが 、コンピュータが従うべき命令を書き記し、その 命令に従うようコンピュータに指示することである。 私たちがRで命令を書く、つまり_コード_を書くのは、それが 、コンピューターも私たちも理解できる共通言語だからだ。 私たちは を_コマンド_と呼び、それらのコマンドを_実行_(_実行_とも呼ぶ)することによって、 の指示に従うようにコンピュータに指示する。
Rと対話する主な方法は2つある:
コンソールを使う方法と、スクリプト(
あなたのコードを含むプレーンテキストファイル)を使う方法である。
コンソールペイン(RStudioでは左下のパネル)は、
、R言語で書かれたコマンドを入力し、
、コンピュータによって即座に実行される場所です。 また、
、実行されたコマンドの結果が表示される場所でもある。 コンソールに直接
コマンドを入力し、Enterを押すことで、それらの
コマンドを実行することができますが、セッションを閉じると忘れてしまいます。
コードとワークフローを再現できるようにしたいので、 、スクリプトエディターで必要なコマンドを入力し、 スクリプトを保存する方がよい。 こうすることで、私たちがしたことの完全な記録が残る。 、誰にでも(未来の自分も含めて!)。 は、 の結果を自分のコンピューターで簡単に再現できる。 ただし、スクリプトに 、単にコマンドを入力しただけでは自動的に_実行_されないことに注意してほしい。 、コンソールに送信して実行させる必要がある。
RStudio ではスクリプトエディター から Ctrl +
Enter ショートカット(Mac では Cmd +
Return で も可)で直接コマンドを実行できます。
Ctrl+Enter`を押すと、スクリプトの現在の行のコマンド(カーソルで
を示す)、または現在選択されているテキスト
のすべてのコマンドがコンソールに送られ、実行される。
その他のキーボードショートカットはRStudio cheatsheet about RStudio
IDEを参照してください。
分析のある時点で、
変数の内容やオブジェクトの構造をチェックしたくなるかもしれない。必ずしもスクリプトに
の記録を残しておく必要はない。
これらのコマンドを入力し、コンソールで直接 。 RStudio には
Ctrl + 1 と Ctrl + 2
のショートカットがあり、スクリプトと
のコンソールペイン間をジャンプすることができます。
Rがコマンドを受け付ける準備ができたら、Rコンソールに
> プロンプトが表示される。
コマンドを受信すると(タイプ、コピーペースト、またはスクリプト
エディターから Ctrl + Enter を使って送信)、R
はそれを実行しようとします。 準備ができると、結果を表示し、
新しいコマンドを待つために新しい >
プロンプトで戻ってきます。
Rがまだ
、データの入力を待っている場合は、コンソールに+プロンプトが表示されます。
これは、 、完全なコマンドの入力が終わっていないことを意味する。 これは、
、括弧や引用符を「閉じて」いないからです。つまり、
、左括弧と右括弧の数や、 、開閉引用符の数が同じではないからです。
このようなことが起こり、
、コマンドを入力し終わったと思った場合、コンソール
ウィンドウ内をクリックし、Escを押してください。これにより、不完全なコマンドがキャンセルされ、
>プロンプトに戻ります。
コース中やコース終了後にさらに学ぶには?
このコースで扱う内容は、あなた自身の 研究のためにデータを分析するために R をどのように使うことができるかを、 初めに体験していただくものです。 しかし、データセットのクリーニング、統計的手法の使用、 、美しいグラフィックスの作成5など、 の高度な操作を行うには、さらに学ぶ必要がある。 Rに習熟し、効率的に使えるようになるための最良の方法は、他のツールと同様、Rを使って 実際の研究課題に取り組むことである。 初心者の場合、ゼロからスクリプトを書かなければならないのは、 困難に感じるかもしれない。 多くの人が自分のコードをオンラインで公開していることを考えると、 自分の目的に合うように既存のコードを修正することで、簡単に始めることができるかもしれない。

助けを求める
RStudio 組み込みのヘルプインターフェイスを使用して、R 関数の詳細情報を検索します。
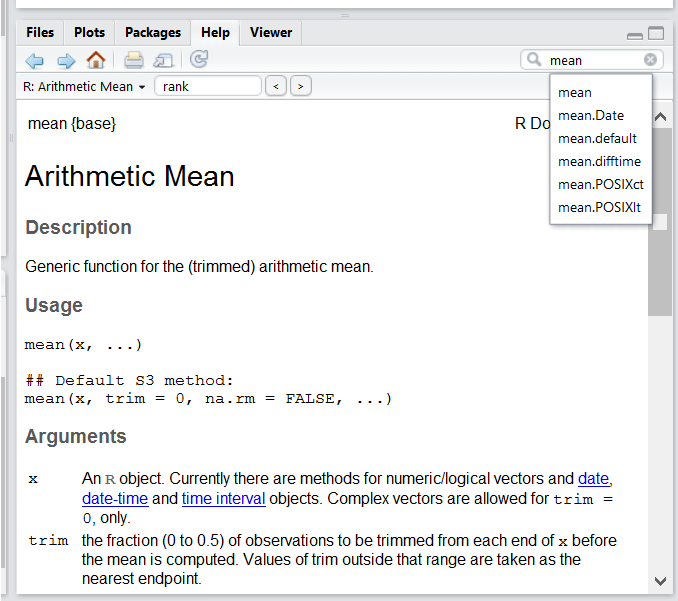
RStudio ヘルプ インターフェイスを使用するのが、ヘルプを得る最も早い方法の1つです。 このパネルはデフォルトで RStudio の右下 パネルにあります。 スクリーンショットに見られるように、 “Mean”という単語を入力すると、RStudioは 、あなたが興味を持ちそうな候補もいくつか出そうとする。 説明文は ウィンドウに表示される。
使いたい関数の名前はわかっているが、その使い方がわからない。
特定の関数、例えばbarplot()のヘルプが必要な場合は、
:
R
バープロット
引数の名前を思い出す必要がある場合は、次のようにすればよい:
R
引数(lm)
Xを行う関数を使いたい。そのための関数があるはずだが、どれがあるのかわからない…。
特定のタスクを実行する関数を探している場合は、
help.search()関数を使用することができます。この関数は二重の疑問符
`?
しかし、これはインストールされているパッケージの中から、検索リクエストと
一致するヘルプページを探すだけです。
R
クルスカル
探しているものが見つからない場合は、 rdocumentation.orgのウェブサイトを使うことができます。このウェブサイトは、利用可能なすべてのパッケージのヘルプファイルから を検索します。
最後に、一般的なグーグルやインターネット検索で “R <task>” を検索すると、多くの場合、 適切なパッケージ・ドキュメントにたどり着くか、 他の誰かがすでに質問している有益なフォーラムにたどり着く。
動けないんだ…。 理解できないエラーメッセージが表示されます。
エラーメッセージをググることから始めよう。 というのも、多くの場合、パッケージ開発者はRが提供するエラー・キャッチに依存しているからである。 、一般的なエラー・メッセージが表示されることになるが、これは の問題を診断するのにあまり役に立たないかもしれない(例えば、“subscript out of bounds”)。 メッセージが非常に一般的なものであれば、 、使用している関数やパッケージの名前を クエリに含めることもできる。
しかし、Stack Overflowをチェックする必要がある。 r]`タグを使って検索する。 ほとんどの の質問にはすでに答えが出されているが、 の答えを見つけるために、検索で適切な の言葉を使うことが課題である:
http://stackoverflow.com/questions/tagged/r
R言語入門](https://cran.r-project.org/doc/manuals/R-intro.pdf) も、プログラミング経験の少ない人にとっては内容が濃いかもしれないが、R言語の基礎を理解するには良い 場所である。
R FAQ](https://cran.r-project.org/doc/FAQ/R-FAQ.html)は密度が濃く、技術的である。 、しかし有用な情報が満載である。
助けを求める
誰かに助けてもらうために重要なのは、相手があなたの問題( )を素早く把握することだ。 、どこに問題がありそうかをできるだけ簡単に特定できるようにすべきだ。
あなたの問題を説明するために、正しい言葉を使うようにしてください。 例えば、 パッケージはライブラリとは違う。 ほとんどの人は、 、あなたが言いたかったことを理解するだろうが、意味の違いについて本当に強い感情を持つ人もいる 。 重要なのは、 、あなたを助けようとする人々を混乱させる可能性があるということだ。 できるだけ正確に問題を説明してください。
可能であれば、うまくいかないことを単純な例(再現可能な )にまで落とし込むようにする。 もし、あなたが50000行10000列のデータではなく、非常に小さなデータ フレームを使って問題を再現できるのであれば、 その小さなデータを問題の説明とともに提供してください。 適切な場合には、 、あなたのやっていることを一般化して、 分野に関係のない人でも質問を理解できるようにする。 例えば、 実際のデータセットのサブセットを使う代わりに、 小さな(3列、5行)一般的なものを作成する。 再現可能な 例の書き方については、Hadley Wickhamによるこの記事を参照のこと。
オブジェクトを他の人と共有するには、それが比較的小さければ、 、関数
dput() を使うことができる。
、メモリ上のオブジェクトとまったく同じオブジェクトを再作成するために使用できるRコードが出力される:
R
## irisはRに付属するデータフレームの例であり、 head()はデータフレームの最初の部分を返す
## 関数である
dput(head(iris))
出力
structure(list(Sepal.Length = c(5.1, 4.9, 4.7, 4.6, 5, 5.4),
Sepal.Width = c(3.5, 3, 3.2, 3.1, 3.6, 3.9), Petal.Length = c(1.4,
1.4, 1.3, 1.5, 1.4, 1.7), Petal.Width = c(0.2, 0.2, 0.2,
0.2, 0.2, 0.4), Species = structure(c(1L, 1L, 1L, 1L, 1L,
1L), levels = c("setosa", "versicolor", "virginica"), class = "factor")), row.names = c(NA,
6L), class = "data.frame")オブジェクトのサイズが大きい場合は、生ファイル(つまり、CSV ファイル)と、エラーが発生した時点までのスクリプト(およびの問題に関係ないものをすべて削除した後のファイル)を提供してください。 あるいは、特にあなたの質問が データフレームに関連していない場合は、任意のRオブジェクトをファイルに保存することができます[^export]:
R
saveRDS(iris, file="/tmp/iris.rds")
しかし、このファイルの内容は人間が読めるものではないので、 Stack
Overflowに直接投稿することはできません。 その代わりに、
。その人はreadRDS()コマンドでそのファイルを読むことができます(ここでは、
、ダウンロードされたファイルは
、そのユーザーのホームディレクトリのDownloadsフォルダにあると仮定しています):
R
some_data <- readRDS(file="~/Downloads/iris.rds")
最後になりますが、必ずsessionInfo()
の出力を含めるようにしてください。プラットフォーム、使用しているRと
パッケージのバージョン、その他問題を理解するのに非常に役立つ情報を提供してくれるからです。
R
sessionInfo()
出力
R version 4.4.1 (2024-06-14)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.4 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.10.0
LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.10.0
locale:
[1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
[4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
[7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
[10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
time zone: Asia/Tokyo
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
loaded via a namespace (and not attached):
[1] compiler_4.4.1 tools_4.4.1 highr_0.11 knitr_1.48
[5] xfun_0.47 evaluate_0.24.0どこに助けを求めればいいのか?
- コース中、あなたの隣に座っている人。 、ワークショップ中に隣の人と話し、自分の答えを比較し、 、助けを求めることをためらわないでください。
- 友好的な同僚:もしあなたより経験豊富な人を知っていれば、 、あなたを助けてくれるかもしれない。
- Stack Overflow: あなたの質問が過去に回答されたことがなく、よく練られたものであれば、 5分以内に回答が得られる可能性があります。 How to ask good questionのガイドラインに従うことを忘れずに。
- R-helpメーリングリスト ](https://stat.ethz.ch/mailman/listinfo/r-help): 多くの人に読まれていて(Rコアチームのほとんどを含む)、多くの人が 投稿していますが、口調はかなり辛口で、 新しいユーザーを必ずしも歓迎しているとは限りません。 あなたの質問が正当なものであれば、 、すぐに回答が返ってくる可能性が高いが、 、スマイルマーク付きで返ってくるとは思わないこと。 また、ここでは他のどこよりも、 正しい語彙を使うようにしましょう(そうしないと、 の質問に答えるのではなく、 あなたの言葉の誤用を指摘する答えが返ってくるかもしれません)。 また、質問内容が特定のパッケージではなく、 ベースとなる関数に関するものであれば、より成功しやすいでしょう。
- 質問が特定のパッケージに関するものであれば、そのパッケージのメーリングリスト(
)があるかどうか確認してください。 通常は
packageDescription("name-of-package")を使ってアクセスできるパッケージのDESCRIPTIONファイル に含まれています。 また、 そのパッケージの作者に直接メールを送ってみたり、 コードリポジトリ(例:GitHub)にissueを開いてみるのもよいだろう。 - また、トピックに特化したメーリングリスト(GIS、 系統遺伝学など)もある。全リストは こちら。
その他のリソース
Rメーリングリストの投稿ガイド。
How to ask for R help 役立つガイドライン。
Jon Skeetによるこのブログ記事 プログラミングの質問の仕方について、かなり包括的なアドバイスがある。
reprex](https://cran.rstudio.com/web/packages/reprex/)パッケージ は、 ヘルプを求めるときに、再現可能な例を作成するのに非常に役立ちます。 rOpenSciコミュニティコール “How to ask questions so they get answered” (Github link and video recording) には、 reprexパッケージとその哲学のプレゼンテーションが含まれています。
Rパッケージ
荷物の積み込み
上で見てきたように、RパッケージはRの基本的な役割を担っている。
、パッケージがインストールされていることを前提に、パッケージの機能を利用する。
、それを利用できるようにするには、まずパッケージをロードする必要がある。
これは library()関数で行う。 以下に ggplot2
をロードする。
R
library("ggplot2")
パッケージのインストール
デフォルトのパッケージリポジトリは The Comprehensive R Archive
Network (CRAN) で、CRAN で利用可能なパッケージは
install.packages() 関数で インストールできます。 例えば、
、後で説明する dplyr パッケージをインストールする。
R
install.packages("dplyr")
このコマンドは、dplyr パッケージと、その
依存パッケージ、つまり、そのパッケージが機能するために依存しているすべてのパッケージをインストールします。
もう一つの主要なRパッケージのリポジトリは、Bioconductorによって管理されている。
Bioconductorパッケージ
は、専用のパッケージ、 すなわち BiocManager
を使用して管理およびインストールされます。
R
install.packages("BiocManager")
SummarizedExperiment(後で )、DESeq2(RNA-Seq解析用)、その他BioconductorやCRANにあるパッケージは、BiocManager::install`で
。
R
BiocManager::install("SummarizedExperiment")
BiocManager::install("DESeq2")
デフォルトでは、BiocManager::install()
はインストールされているすべてのパッケージをチェックし、新しいバージョンがあるかどうかも確認します。
もしあれば、それが表示され、「すべて/いくつか/なしを更新しますか?
[a/s/n]:`、そしてあなたの答えを待つ。
パッケージのバージョンは最新のものを用意するよう努力すべきですが、実際には、パッケージがロードされる前の新鮮なRセッションでのみパッケージを更新することをお勧めします。
コマンドライン コンソールから直接Rを使うのとは対照的だ。 、Rとインターフェイスし統合するソフトウェアは他にもあるが、RStudioは非常に高度な機能を数多く備えながら、特に初心者向け 。↩︎
すなわち、バイオインフォマティクスのデータ解析など、Rに新しい機能を付与するアドオンである。 。↩︎
このコースでは、バイオインフォマティクスを、 、生物学的または生物医学的データに適用されるデータサイエンスと考える。↩︎
その誰かとは、 、分析が実行された数カ月後、あるいは数年後に、 、未来のあなた自身である可能性が高い。↩︎
ここでは、これらのほとんど(統計学を除く) を紹介するが、Rで可能なこと の富の表面をかすめることしかできない。↩︎
Content from R の紹介
最終更新日:2024-09-08 | ページの編集
所要時間: 120分
概要
質問
- R の最初のコマンド
目的
- R に関連する次の用語を定義します: オブジェクト、代入、呼び出し、関数、引数、オプション。
- R のオブジェクトに値を割り当てます。
- オブジェクトに _名前を付ける_方法を学ぶ
- コメントを使用してスクリプトに情報を与えます。
- R で単純な算術演算を解きます。
- 関数を呼び出し、引数を使用してデフォルトのオプションを変更します。
- ベクトルの内容を検査し、その内容を操作します。
- ベクトルから値をサブセット化して抽出します。
- データが欠落しているベクトルを解析します。
This episode is based on the Data Carpentries’s Data Analysis and Visualisation in R for Ecologists lesson.
R でオブジェクトを作成する
コンソールに math と入力するだけで、R から出力を取得できます。
R
3 + 5
出力
[1] 8R
12 / 7
出力
[1] 1.714286ただし、便利で興味深いことを行うには、_値_を オブジェクト
に割り当てる必要があります。
オブジェクトを作成するには、オブジェクトに名前を付け、その後に
代入演算子 <-
と、それに付けたい値を付ける必要があります。
R
weight_kg <- 55
<- は代入演算子です。 右側の値を左側の
個のオブジェクトに割り当てます。 したがって、「x <-
3」を実行すると、「x」の値は 3 になります。 矢印は 3
が x に入る と読むことができます。 歴史的
理由により、代入に = を使用することもできますが、
のコンテキストで使用できるわけではありません。 構文に わずかな違い](https://blog.revolutionanalytics.com/2008/12/use-equals-or-arrow-for-assignment.html)
があるため、常に < を使用することをお勧めします。 -
割り当て用。
RStudio では、 オプション を入力しながら、 Alt
+ - を入力すると ( - キーと同時に Alt
を押すと)、PC で 1 回のキーストロークで <-
が書き込まれます。 + - ( オプション -
キーと同時に押す) は、Mac でも と同じことを行います。
変数に名前を付ける
オブジェクトには、「x」、「current_temperature」、または「subject_id」などの任意
名前を付けることができます。 オブジェクト名は明示的で、長
ないようにしたいと考えています。 数字で始めることはできません
(「2x」は無効ですが、「x2」 は有効です)。 R
では大文字と小文字が区別されます (たとえば、weight_kg は の
Weight_kg とは異なります)。 R
の基本的な関数の名前であるため、使用でき 名前が かあります (例:
if、else、 for。2 こちらを参照)完全なリストについては、/R-manual/R-devel/library/base/html/Reserved.html)
)。 一般に、たとえ許可されていても、他の関数名 (例:
c、T、mean、data、df、
weights) は使用しないことが です。
疑問がある場合は、ヘルプを参照して、その名前がすでに
で使用されているかどうかを確認してください。 また
my.dataset のように、オブジェクト名内にドット
(.) を使用しないことも最善です。 R
には歴史的な理由から名前にドットが含まれる関数が多数あります が、R
(メソッド)
や他のプログラミング言語ではドットが特別な意味を持っているため、ドットは避けるのが最善です
。 オブジェクト名には名詞を使用し、関数名には動詞
を使用することもお勧めします。 のスタイル
(スペースを入れる場所、オブジェクトの名前など)
に一貫性を持たせることが重要です。 コーディング スタイルを使用すると、
の自分や共同作業者にとって、コードがより明確に読みやすくなります。 R
では、人気のあるスタイル ガイド には、Google の、 tidyverse の
スタイル、およびBioconductor スタイル ガイド。 Tidyverse
は非常に包括的であり、最初は では圧倒されるように思えるかもしれません。
lintr
パッケージを
にインストールすると、コードのスタイルの問題が自動的にチェックされます。
オブジェクトと変数: 「R」で「オブジェクト」として知られているものは、他の多くのプログラミング言語では「変数」として知られて ます。 に応じて、「オブジェクト」と「変数」は に異なる意味を持つ可能性があります。 ただし、このレッスンでは、2 つの単語は 的に使用されます。 詳細については、 ここを参照してください。
オブジェクトに値を割り当てるとき、R は何も出力しません。 かっこを使用するか 名を入力することで、 に値を強制的に出力させることができます。
R
weight_kg <- 55 # 何も出力しません
(weight_kg <- 55) # しかし、呼び出しを括弧で囲むと `weight_kg` の値が出力され、
出力
[1] 55R
weight_kg # オブジェクトの名前を入力しても同様に出力されます
出力
[1] 55R のメモリに「weight_kg」があるので、それを使って算術演算を行うことができます。 、この重量をポンドに変換したい場合があります (ポンドでの重量は kg での重量の 2.2 倍です)。
R
2.2 * weight_kg
出力
[1] 121オブジェクトに新しい値を割り当てることで、オブジェクトの値を変更することもできます。
R
weight_kg <- 57.5
2.2 * weight_kg
出力
[1] 126.5これは、
つのオブジェクトに値を割り当てても、他のオブジェクトの値は変更されないことを意味します。たとえば、動物の体重をポンド単位で新しい
オブジェクト weight_lb に保存してみましょう。
R
weight_lb <- 2.2 * weight_kg
次に「weight_kg」を 100 に変更します。
R
weight_kg <- 100
コメント
のコメント文字は # です。0 スクリプトの #
の右側にあるものはすべて R によって無視されます。スクリプトにメモ
説明を残すと便利です。
RStudio では、段落のコメントまたはコメント解除が簡単に行えます。 コメントしたい行を選択した後、 キーボード Ctrl + Shift + Cを同時に押します。 の場合、1 行だけをコメントアウトしたい場合は、その行の任意 位置にカーソルを置きます (つまり、行全体を選択する必要はありません)。その後 Ctrl + Shift + C押します。
関数とその引数
関数は、操作の割り当て を含む、より複雑なコマンド
セットを自動化する「定型スクリプト」です。
多くの関数は事前定義されているか、R パッケージ
をインポートすることで 可能になります (詳細は後ほど)。 関数
は通常、arguments と呼ばれる 1 つ以上の入力を取得します。
関数は多くの場合 (常に ではありませんが) 値 を返します。
典型的な例は関数 sqrt() です。 入力 (引数)
は数値でなければならず、戻り値 (実際には 出力) はその数値の平方根です。
関数の実行 (「実行中」) は関数の 呼び出し と呼ばれます。
関数呼び出しの例は次のとおりです。
R
b <- sqrt(a)
ここでは、a の値が sqrt()
関数に与えられ、sqrt() 関数は
平方根を計算し、その値をオブジェクト ` に代入して返します。
この関数は引数を 1 つだけ取るため、非常に単純です。
関数の戻り値「値」は数値 (sqrt() のような)
である必要はなく、
である必要もありません。また、単一の項目である必要もありません。一連のものや
、さらにはデータセットでも構いません。 データ ファイルを R
に読み込むと、それがわかります。
引数には、数値やファイル名だけでなく、他の も含めることができます。 各引数の正確な意味は関数ごとに異なるため、ドキュメントで調べて にする必要があります (下記を参照)。 一部の関数は引数を取ります はユーザーによって指定されるか、指定されなかった場合は デフォルト 値を取ります: これらは オプション と呼ばれます。 オプションは通常、「不正な値」を無視するかどうか、プロットでどのような記号を使用 かなど、 関数の動作方法を変更するために使用されます。 ただし、特定の値が必要な場合は、デフォルトの代わりに使用される値 を選択して指定できます。
複数の引数を取ることができる関数 round()
を試してみましょう。
R
round(3.14159)
出力
[1] 3ここでは、1 つの引数 3.14159 を指定して
round() を呼び出しましたが、 が値 3
を返しました。 これは、デフォルトでは最も近い 整数に丸められるためです。
さらに多くの桁が必要な場合は、「round」関数に関する
情報を取得することでその方法がわかります。 args(round)
を使用するか、?round を使用して
関数のヘルプを参照することができます。
R
args(round)
出力
function (x, digits = 0, ...)
NULLR
?round
別の桁数が必要な場合は、digits=2 または必要な桁数を入力
ことがわかります。
R
round(3.14159, digits = 2)
出力
[1] 3.14定義されているのとまったく同じ順序で引数を指定する場合は に名前を付ける必要はありません。
R
round(3.14159, 2)
出力
[1] 3.14引数に名前を付けた場合は、その順序を入れ替えることができます。
R
round(digits = 2, x = 3.14159)
出力
[1] 3.14関数呼び出しの最初にオプションではない引数 ( 四捨五入する数値など) を置き、すべてのオプションの 引数の名前を指定することをお勧めします。 そうしないと、コードを読む人が、 をしているのかを理解するために、なじみのない引数を持つ関数の定義を調べなければなら 可能性があります。 引数の名前を指定することで、関数インターフェースの将来の変更 (既存の引数の間に 引数が追加される可能性) から することもできます。
ベクトルとデータ型
ベクトルは R で最も一般的かつ基本的なデータ型であり、ほぼ R
の主力である です。ベクトルは、 数字や文字などの一連の値で構成されます。
の c()
関数を使用して、一連の値をベクトルに割り当てることができます。
たとえば、動物の体重のベクトルを作成し、それを新しいオブジェクト
weight_g に に割り当てることができます。
R
weight_g <- c(50, 60, 65, 82)
weight_g
出力
[1] 50 60 65 82ベクトルには文字も含めることができます。
R
molecules <- c("dna", "rna", "protein")
molecules
出力
[1] "dna" "rna" "protein"ここでは「dna」や「rna」などの引用符が重要です。 引用符 がないと、R
は dna、rna、および protein
と呼ばれるオブジェクトがあると想定します。 これらのオブジェクトは R
のメモリに存在しないため、エラー メッセージが されます。
ベクトルの内容を検査できる関数が多数あります。 length()
は、特定のベクトルに含まれる要素の数を示します。
R
length(weight_g)
出力
[1] 4R
length(molecules)
出力
[1] 3ベクトルの重要な特徴は、すべての要素が タイプのデータであることです。
関数 class() は、オブジェクトのクラス ( 型の要素)
を示します。
R
class(weight_g)
出力
[1] "numeric"R
class(molecules)
出力
[1] "character"関数 str() は、
オブジェクトとその要素の構造の概要を提供します。 これは、
て複雑なオブジェクトを扱う場合に便利な関数です。
R
str(weight_g)
出力
num [1:4] 50 60 65 82R
str(molecules)
出力
chr [1:3] "dna" "rna" "protein"c()
関数を使用して、ベクトルに他の要素を追加できます。
R
weight_g <- c(weight_g, 90) # ベクトルの最後に追加
weight_g <- c(30, weight_g) # ベクトルの先頭に追加
weight_g
出力
[1] 30 50 60 65 82 90最初の行では、元のベクトル weight_g を取得し、その末尾に
値 90 を追加し、結果を weight_g に保存します。
次に、値 30 を先頭に追加し、結果を再び として
weight_g に保存します。
これを何度も繰り返してベクトルを成長させたり、 データセットを組み立てたりすることができます。 これは、プログラムするときに、 または計算している結果を追加するのに役立つ場合があります。
アトミック ベクトルは最も単純な R
データ型であり、単一型の線形 ベクトルです。 上では、R
が使用する 6 つの主な アトミック ベクトル タイプのうち
2 つ、つまり "character" と "numeric" (または
"double") を見てきました。 これらは、すべての R
オブジェクト が構築される基本的な構成要素です。 他の 4 つの
原子ベクトル タイプは次のとおりです。
-
TRUEおよびFALSEの場合は"logical"(ブール データ型) - 整数の場合は
"integer"(たとえば、2L、Lは R にそれが整数であることを示します) -
"complex"は、実数と虚数の 部分を持つ複素数を表します (例: 1 + 4i)。これについて説明するのはこれですべてです。 - ビットストリームの「raw」` (これ以上は説明しません)
typeof() 関数
を使用し、ベクトルを引数として入力することで、ベクトルの型をチェックできます。
ベクトルは、R が使用する多くの データ構造 の 1
つです。 その他 重要なものは、リスト (list)、行列
(matrix)、データ フレーム (data.frame)、因子
(factor)、および配列 (array) です。
R はそれらをすべて同じ型に暗黙的に変換します。
R
class(num_char)
出力
[1] "character"R
num_char
出力
[1] "1" "2" "3" "a"R
class(num_logical)
出力
[1] "numeric"R
num_logical
出力
[1] 1 2 3 1 0R
class(char_logical)
出力
[1] "character"R
char_logical
出力
[1] "a" "b" "c" "TRUE"R
class(tricky)
出力
[1] "character"R
tricky
出力
[1] "1" "2" "3" "4"ベクトルのデータ型は 1 つだけです。 R は、 が情報を失わないという 共通分母 を見つけるために、このベクトルの内容を に変換 (強制) しようとします。
唯一。 過去のデータ型の記憶はなく、ベクトルが初めて評価されるときに強制 が発生します。 したがって、「num_logical」の「TRUE」 「combined_logical」で 「1」に変換される前に、「1」に変換されます。
R
combined_logical
出力
[1] "1" "2" "3" "1" "a" "b" "c" "TRUE"論理 → 数値 → 文字 ← 論理
ベクトルのサブセット化
ベクトルから 1 つまたは複数の値を抽出したい場合は、角括弧内に 1 つまたは複数のインデックスを指定する必要が ます。 例えば:
R
molecules <- c("dna", "rna", "peptide", "protein")
molecules[2]
出力
[1] "rna"R
molecules[c(3, 2)]
出力
[1] "peptide" "rna" インデックスを繰り返して、元のオブジェクトよりも要素 が多いオブジェクトを作成することもできます。
R
more_molecules <- molecules[c(1, 2, 3, 2, 1, 4)]
more_molecules
出力
[1] "dna" "rna" "peptide" "rna" "dna" "protein"R インデックスは 1 から始まります。 Fortran、MATLAB、 Julia、R などのプログラミング言語は から数え始めます。これは人間が通常行うことだからです。 C ファミリの言語 (C++、Java、Perl、 、Python を含む) は 0 からカウントします。これは、コンピュータにとってその方が簡単なためです。
最後に、負のインデックスを使用して、指定された一部の要素を除くベクトル のすべての要素を取得することもできます。
R
分子 ## すべての分子
エラー
Error in eval(expr, envir, enclos): object '分子' not foundR
分子[-1] ## 最初の分子を除くすべての分子
エラー
Error in eval(expr, envir, enclos): object '分子' not foundR
分子[-c(1, 3)] ## 1 番目/3 番目の分子を除くすべての分子
エラー
Error in eval(expr, envir, enclos): object '分子' not foundR
分子[c(-1, -3)] # # 1番目/3番目を除くすべて
エラー
Error in eval(expr, envir, enclos): object '分子' not found条件付きサブセット化
サブセット化のもう 1
つの一般的な方法は、論理ベクトルを使用することです。 TRUE
は同じインデックスを持つ要素を選択し が、FALSE
は選択しません。
R
weight_g <- c(21, 34, 39, 54, 55)
weight_g[c(TRUE, FALSE, TRUE, TRUE, FALSE)]
出力
[1] 21 39 54通常、これらの論理ベクトルは手動で入力されるのではなく、他の関数または論理テストの 出力です。 たとえば、50 を超える値のみを選択したい場合は、 のようにします。
R
## will return logicals with TRUE for the indices that meet
## the condition
weight_g > 50
出力
[1] FALSE FALSE FALSE TRUE TRUER
## so we can use this to select only the values above 50
weight_g[weight_g > 50]
出力
[1] 54 55& (両方の条件が true、 AND) または |
(少なくとも 1 つの条件が true、OR)
を使用して複数のテストを結合できます。
R
weight_g[weight_g < 30 | weight_g > 50]
出力
[1] 21 54 55R
weight_g[weight_g >= 30 & weight_g == 21]
出力
numeric(0)ここで、「<」は「より小さい」、「>」は「より大きい」、「>=」は
「以上」、「==」は「等しい」を表します。 2 つの等号 記号「==」は、左側と
の数値が等しいかどうかをテストするものであり、(「<-」と同様に)
変数の代入を する単一の = 記号と混同しないでください。
。
一般的なタスクは、ベクトル内の特定の文字列を検索することです。
「or」演算子 |
を使用して複数の値が等しいかどうかをテストすることもできますが、
これはすぐに面倒になります。 関数 %in%
を使用すると、検索ベクトルの要素が見つかったかどうかを できます。
R
分子 <- c("dna", "rna", "タンパク質", "ペプチド")
分子[分子 == "rna" |分子 == "dna"] # rna と dna の両方を返します
分子 %in% c("rna", "dna", "代謝物", "ペプチド", "グリセロール")
分子[分子 %in% c("rna", " 「DNA」、「代謝物」、「ペプチド」、「グリセロール」)]エラー
Error: <text>:4:21: unexpected INCOMPLETE_STRING
3: 分子 %in% c("rna", "dna", "代謝物", "ペプチド", "グリセロール")
4: 分子[分子 %in% c("rna", " 「DNA」、「代謝物」、「ペプチド」、「グリセロール」)]
^R
"four" > "five"
出力
[1] TRUE文字列で > または < を使用すると、R
はそれらのアルファベット順を比較します。 ここで、"four" は
"five" の後に来るので、それは * より大きい* です。
名前
ベクトルの各要素に名前を付けることができます。 より下のコード チャンクは、名前のない初期ベクトル、名前の設定方法、および 取得される様子を示しています。
R
x <- c(1, 5, 3, 5, 10)
names(x) ## 名前なし
出力
NULLR
names(x) <- c("A", "B", "C", "D", " E")
名前(x) ## これで名前が決まりました
エラー
Error in 名前(x): could not find function "名前"ベクトルに名前がある場合、 に加えて名前によって要素にアクセスすることができます。
R
x[c(1, 3)]
出力
A C
1 3 R
x[c("A", "C")]
出力
A C
1 3 データが欠落しています
R はデータセットを分析するように設計されているため、欠損データが であるという概念が含まれています (これは のプログラミング言語では一般的ではありません)。 欠損データはベクトルで「NA」として表されます。
数値の演算を行う場合、扱っているデータに欠損値が含まれている場合
ほとんどの関数は「NA」を返します。 この機能により、 データを処理し
いるケースを見逃しにくくなります。 引数 na.rm = TRUE
を追加すると、欠損値を無視して結果を として計算できます。
R
heights <- c(2, 4, 4, NA, 6)
mean(heights)
出力
[1] NAR
max(heights)
出力
[1] NAR
mean(heights, na.rm = TRUE)
出力
[1] 4R
max(heights, na.rm = TRUE)
出力
[1] 6データに欠損値が含まれている場合は、関数
is.na()、na.omit()、および
complete.cases() に ておくとよいでしょう。
例については、以下の を参照してください。
R
## 欠損値のない要素を抽出します。
heights[!is.na(heights)]
出力
[1] 2 4 4 6R
## 不完全なケースを削除したオブジェクトを返します。
## 返されるオブジェクトは、タイプ `"numeric"` の原子ベクトルです。
## (または `"double"`)。
na.omit(heights)
出力
[1] 2 4 4 6
attr(,"na.action")
[1] 4
attr(,"class")
[1] "omit"R
## 完全なケースである要素を抽出します。
## 返されるオブジェクトは、タイプ `"numeric"` の原子ベクトルです。
## (または `"double"`)。
の高さ[完全なケース(高さ)]
エラー
Error in eval(expr, envir, enclos): object 'の高さ' not foundR
heights_no_na <- heights[!is.na(heights)]
## または
heights_no_na <- na.omit(heights)
R
median(heights, na.rm = TRUE)
出力
[1] 4R
height_above_67 <- height_no_na[heights_no_na > 67]
エラー
Error in eval(expr, envir, enclos): object 'height_no_na' not foundR
長さ(heights_above_67)
エラー
Error in 長さ(heights_above_67): could not find function "長さ"ベクトル {#sec:genvec}の生成
コンストラクター
異なるタイプのベクトルを生成する関数がいくつか存在します。
数値のベクトルを生成 には、numeric()
コンストラクターを使用し、出力ベクトルの長さを
パラメーターとして指定します。 値は 0 で初期化されます。
R
数値(3)
エラー
Error in 数値(3): could not find function "数値"R
数値(10)
エラー
Error in 数値(10): could not find function "数値"長さ 0 の数値ベクトルを要求すると、次のように が得られることに注意してください。
R
数値(0)
エラー
Error in 数値(0): could not find function "数値"文字と論理に対しても同様のコンストラクターがあり、
character() と logical()
という名前が付けられます。
R
文字(2) ## 空の文字
エラー
Error in 文字(2): could not find function "文字"R
論理的(2) ## FALSE
エラー
Error in 論理的(2): could not find function "論理的"要素を複製する
rep 関数を使用すると、値を特定の回数 ( 回)
繰り返すことができます。 たとえば、長さ 5 の数値ベクトルを から値 -1
で開始したい場合は、次のようにすることができます。
R
担当者(-1, 5)
エラー
Error in 担当者(-1, 5): could not find function "担当者"同様に、収集されるデータ に仮定を設定せずに、欠損値が入力されたベクトルを生成するには (多くの場合、 から始めるのが良い方法です):
R
担当者(NA, 5)
エラー
Error in 担当者(NA, 5): could not find function "担当者"rep は、入力として任意の長さのベクトル (上記では長さ 1
のベクトル を使用しました) および任意のタイプを受け取ることができます。
たとえば、 値 1、2、3 を 5 回繰り返す場合は、次のようにします。
R
rep(c(1, 2, 3), 5)
出力
[1] 1 2 3 1 2 3 1 2 3 1 2 3 1 2 3R
rep(c(1, 2, 3), each = 5)
出力
[1] 1 1 1 1 1 2 2 2 2 2 3 3 3 3 3R
sort(rep(c(1, 2, 3), 5))
出力
[1] 1 1 1 1 1 2 2 2 2 2 3 3 3 3 3シーケンスの生成
もう 1 つの非常に便利な関数は、 の数値シーケンスを生成する
seq です。 たとえば、1 から 20 までの整数のシーケンスを 2
ずつ生成するには、次のコマンドを使用します。
R
seq(from = 1, to = 20, by = 2)
出力
[1] 1 3 5 7 9 11 13 15 17 19by のデフォルト値は 1 で、1 のステップで 1
つの値から別の値への シーケンスの生成が頻繁に使用されることを考えると、
というショートカットがあります。
R
seq(1, 5, 1)
出力
[1] 1 2 3 4 5R
seq(1, 5) ## default by
出力
[1] 1 2 3 4 5R
1:5
出力
[1] 1 2 3 4 5最終長さが の 1 から 20 までの一連の数値を生成するには、次のコマンドを使用します。
R
seq(from = 1, to = 20, length.out = 3)
出力
[1] 1.0 10.5 20.0ランダムなサンプルと順列
有用な関数の最後のグループは、ランダムな データを生成する関数です。
最初の sample は、 のベクトルのランダムな置換を生成します。
たとえば、口頭試験を行わない 人の生徒にランダムな順序を付けるには
まず各生徒に 1 から 10 までの番号を割り当てます
(たとえば、名前のアルファベット順に基づきます)。次に次のようにします。
R
sample(1:10)
出力
[1] 9 4 7 1 2 5 3 10 6 8さらなる引数がなければ、sample はベクトルのすべての
要素の順列を返します。 特定のサイズのランダムなサンプルが必要な場合、I
はこの値を 2 番目の引数として設定します。 以下では、事前定義された
letters ベクトルに含まれるアルファベットから 5
つのランダムな 文字をサンプリングします。
R
sample(letters, 5)
出力
[1] "s" "a" "u" "x" "j"入力ベクトルよりも大きな出力が必要な場合、または一部の要素を複数回
できるようにしたい場合は、引数 replace を TRUE
に設定する必要があります。
R
sample(1:5, 10, replace = TRUE)
出力
[1] 2 1 5 5 1 1 5 5 2 2チャレンジ:
上記の関数を試してみると、 サンプルは実際にランダムであり、同じ
順列が 2 回発生することはないことがわかるでしょう。
これらのランダムな描画を再現できるようにするには、ランダム
サンプルを描画する前に set.seed() を使用
て乱数生成シードを手動で設定します。
近所の人と一緒にこの機能をテストしてください。 まず、「1:10」のランダムな 順列を 2 つ個別に描画し、 の異なる結果が得られることを観察します。
次に、たとえば set.seed(123) でシードを設定し、
ランダムな描画を繰り返します。
同じランダムな抽選が行われることに注目してください。
別のシードを設定して繰り返します。
さまざまな順列
R
sample(1:10)
出力
[1] 9 1 4 3 6 2 5 8 10 7R
sample(1:10)
出力
[1] 4 9 7 6 1 10 8 3 2 5シード 123 と同じ順列
R
set.seed(123)
sample(1:10)
出力
[1] 3 10 2 8 6 9 1 7 5 4R
set.seed(123)
sample(1:10)
出力
[1] 3 10 2 8 6 9 1 7 5 4違う種
R
set.seed(1)
sample(1:10)
出力
[1] 9 4 7 1 2 5 3 10 6 8R
set.seed(1)
sample(1:10)
出力
[1] 9 4 7 1 2 5 3 10 6 8正規分布からサンプルを抽出する
最後に説明する関数は rnorm で、正規分布からランダムな
サンプルを抽出します。 平均 および 100、標準偏差 1 および 5 の 2
つの正規分布 (N(0, 1) および N(100, 5) と表記)
を以下に示します。
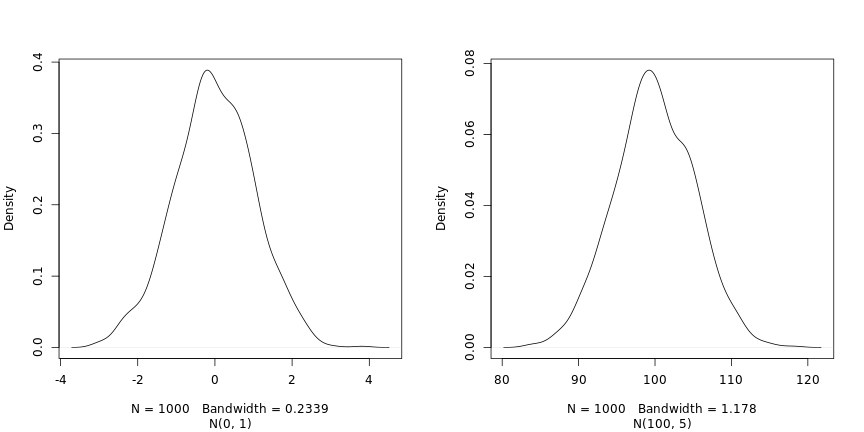
3 つの引数「n」、「mean」、「sd」は、サンプル のサイズと、正規分布のパラメーター、つまり平均 とその標準偏差を定義します。 後者のデフォルトは 0 と 1 です。
R
rnorm(5)
出力
[1] 0.69641761 0.05351568 -1.31028350 -2.12306606 -0.20807859R
rnorm(5, 2, 2)
出力
[1] 1.3744268 -0.1164714 2.8344472 1.3690969 3.6510983R
rnorm(5, 100, 5)
出力
[1] 106.45636 96.87448 95.62427 100.71678 107.12595スクリプトの書き方と のデータ構造の基本を学習したので、より大きなデータの操作を開始する準備が整い、データ フレームについて します。
Content from データから始める
最終更新日:2024-09-08 | ページの編集
所要時間: 60分
概要
質問
- Rによる最初のデータ分析
目的
-
data.frameが何なのか説明してみましょう。 - .csv ファイルからデータ フレームに外部データを読み込みましょう。
- データフレームの内容を要約してみましょう。
- ファクターとは何か?
- string と factor を変換してみましょう。
- factor の並び替えとリネームを行ってみましょう。
- 日付をフォーマットしてみましょう。
- データをエクスポートして保存してみましょう。
このエピソードは、Data Carpentriesの_Data Analysis and Visualisation in R for Ecologists_レッスンに基づいています。
遺伝子発現データのプレゼンテーション
Blackmore et al. (2017)](https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5544260/), The effect of upper-respiratory infection on transcriptomic changes in CNS によって発表されたデータの一部を使用する予定である。 研究の目的は、 上部呼吸器感染症が、感染後の小脳と脊髄で 起こるRNA転写の変化に及ぼす影響を調べることであった。 性別を一致させた8匹の 週齢のC57BL/6マウスに、生理食塩水または 鼻腔内経路でインフルエンザAを接種し、0日目 (非感染)、4日目、8日目に小脳と 脊髄組織におけるトランスクリプトーム変化をRNA-seqで評価した。
データセットは、カンマ区切りの値(CSV)ファイルとして保存される。 各行 は1つのRNA発現測定の情報を持ち、最初の11列 はそれを表している:
| コラム | 説明 |
|---|---|
| 遺伝子 | 測定された遺伝子名 |
| サンプル | 遺伝子発現を測定したサンプル名 |
| 表現 | 遺伝子発現の値 |
| 有機体 | 生物/種 - ここではすべてのデータはマウスに由来する |
| 年齢 | マウスの年齢(ここではすべてのマウスが8週齢であった) |
| セックス | マウスの性別 |
| 感染症 | マウスの感染状態、すなわちA型インフルエンザに感染しているか、感染していないか。 |
| 緊張 | インフルエンザA型。 |
| 時間 | 感染期間(日単位)。 |
| 組織 | 遺伝子発現実験に使用した組織、すなわち小脳または脊髄。 |
| マウス | マウス固有の識別子。 |
R関数のdownload.file()を使って遺伝子発現データを含む
CSVファイルをダウンロードし、 read.csv()
を使ってCSVファイルの内容を
data.frameクラスのオブジェクトとしてメモリにロードする。
download.fileコマンドの内部では、 の最初のエントリーは、ソースURLの文字列である。 このソースURL はGitHubリポジトリからCSVファイルをダウンロードします。 、カンマ(“data/rnaseq.csv”)の後のテキストは、 、ローカルマシン上のファイルの保存先です。 あなたのマシンに“data”というフォルダを用意し、そこにファイルをダウンロードする必要があります。 そこで、このコマンドは リモートファイルをダウンロードし、“rnaseq.csv”という名前を付けて、“data”`
という名前の フォルダに追加する。
R
download.file(url = "https://github.com/carpentries-incubator/bioc-intro/raw/main/episodes/data/rnaseq.csv",
destfile = "data/rnaseq.csv")
これでデータをロードする準備ができた:
R
rna <- read.csv("data/rnaseq.csv")
、代入は何も表示しないからだ。 、データがロードされたことを確認したい場合は、 、その名前をタイプすることでデータフレームの中身を見ることができる:
R
RNA
うわぁ…。 多くのアウトプットがあった。 少なくとも、 。 関数
head() を使って、このデータ・フレーム
の先頭(最初の6行)をチェックしてみよう:
R
head(rna)
出力
gene sample expression organism age sex infection strain time
1 Asl GSM2545336 1170 Mus musculus 8 Female InfluenzaA C57BL/6 8
2 Apod GSM2545336 36194 Mus musculus 8 Female InfluenzaA C57BL/6 8
3 Cyp2d22 GSM2545336 4060 Mus musculus 8 Female InfluenzaA C57BL/6 8
4 Klk6 GSM2545336 287 Mus musculus 8 Female InfluenzaA C57BL/6 8
5 Fcrls GSM2545336 85 Mus musculus 8 Female InfluenzaA C57BL/6 8
6 Slc2a4 GSM2545336 782 Mus musculus 8 Female InfluenzaA C57BL/6 8
tissue mouse ENTREZID
1 Cerebellum 14 109900
2 Cerebellum 14 11815
3 Cerebellum 14 56448
4 Cerebellum 14 19144
5 Cerebellum 14 80891
6 Cerebellum 14 20528
product
1 argininosuccinate lyase, transcript variant X1
2 apolipoprotein D, transcript variant 3
3 cytochrome P450, family 2, subfamily d, polypeptide 22, transcript variant 2
4 kallikrein related-peptidase 6, transcript variant 2
5 Fc receptor-like S, scavenger receptor, transcript variant X1
6 solute carrier family 2 (facilitated glucose transporter), member 4
ensembl_gene_id external_synonym chromosome_name gene_biotype
1 ENSMUSG00000025533 2510006M18Rik 5 protein_coding
2 ENSMUSG00000022548 <NA> 16 protein_coding
3 ENSMUSG00000061740 2D22 15 protein_coding
4 ENSMUSG00000050063 Bssp 7 protein_coding
5 ENSMUSG00000015852 2810439C17Rik 3 protein_coding
6 ENSMUSG00000018566 Glut-4 11 protein_coding
phenotype_description
1 abnormal circulating amino acid level
2 abnormal lipid homeostasis
3 abnormal skin morphology
4 abnormal cytokine level
5 decreased CD8-positive alpha-beta T cell number
6 abnormal circulating glucose level
hsapiens_homolog_associated_gene_name
1 ASL
2 APOD
3 CYP2D6
4 KLK6
5 FCRL2
6 SLC2A4R
##
## View(rna)も試してみる。
注*。
read.csv()は、フィールドがカンマで区切られていると仮定しているが、 いくつかの国では、カンマは小数の区切り文字として使用され、 セミコロン(;)はフィールドの区切り文字として使用される。 Rでこの種のファイルを 、read.csv2()関数を使うことができる。read.csv()と全く同じ動作をするが、 小数とフィールドのセパレーターに異なるパラメーターを使用する。 別の フォーマットを使用している場合は、ユーザーが両方指定することができます。 詳しくは、read.csv()のヘルプを?read.csvと入力して確認してください。 また、read.delim()関数があり、タブ区切りのデータファイルを読み込むことができる。 重要なことは、これらの関数はすべて、 メインのread.table()関数に異なる引数を指定するためのラッパー関数であるということです。 そのため、 上のデータは、read.table()、区切りの引数を,`
にしてロードすることもできた。 コードは以下の通り:
R
rna <- read.table(file = "data/rnaseq.csv",
sep = ",",
header = TRUE)
デフォルトでは read.table() の header 引数は FALSE
に設定されているので、 のヘッダーを読むためには header 引数を TRUE
に設定しなければならない。
データフレームとは?
データ・フレームは、ほとんどの表データ、 、統計やプロットに使われる_事実上の_データ構造である。
データフレームは手作業で作成することもできますが、最も一般的なのは、関数
read.csv() や read.table() によって生成される
データフレームです。
データフレームとは、 、列がすべて同じ長さのベクトルである表の形式でデータを表現したものである。 、列はベクトルであるため、各列は1種類のデータ (文字、整数、因子など)を含まなければならない。 例えば、 、数値、文字、 論理ベクトルからなるデータフレームを示す図である。
str()`という関数で :
R
str(rna)
出力
'data.frame': 32428 obs. of 19 variables:
$ gene : chr "Asl" "Apod" "Cyp2d22" "Klk6" ...
$ sample : chr "GSM2545336" "GSM2545336" "GSM2545336" "GSM2545336" ...
$ expression : int 1170 36194 4060 287 85 782 1619 288 43217 1071 ...
$ organism : chr "Mus musculus" "Mus musculus" "Mus musculus" "Mus musculus" ...
$ age : int 8 8 8 8 8 8 8 8 8 8 ...
$ sex : chr "Female" "Female" "Female" "Female" ...
$ infection : chr "InfluenzaA" "InfluenzaA" "InfluenzaA" "InfluenzaA" ...
$ strain : chr "C57BL/6" "C57BL/6" "C57BL/6" "C57BL/6" ...
$ time : int 8 8 8 8 8 8 8 8 8 8 ...
$ tissue : chr "Cerebellum" "Cerebellum" "Cerebellum" "Cerebellum" ...
$ mouse : int 14 14 14 14 14 14 14 14 14 14 ...
$ ENTREZID : int 109900 11815 56448 19144 80891 20528 97827 118454 18823 14696 ...
$ product : chr "argininosuccinate lyase, transcript variant X1" "apolipoprotein D, transcript variant 3" "cytochrome P450, family 2, subfamily d, polypeptide 22, transcript variant 2" "kallikrein related-peptidase 6, transcript variant 2" ...
$ ensembl_gene_id : chr "ENSMUSG00000025533" "ENSMUSG00000022548" "ENSMUSG00000061740" "ENSMUSG00000050063" ...
$ external_synonym : chr "2510006M18Rik" NA "2D22" "Bssp" ...
$ chromosome_name : chr "5" "16" "15" "7" ...
$ gene_biotype : chr "protein_coding" "protein_coding" "protein_coding" "protein_coding" ...
$ phenotype_description : chr "abnormal circulating amino acid level" "abnormal lipid homeostasis" "abnormal skin morphology" "abnormal cytokine level" ...
$ hsapiens_homolog_associated_gene_name: chr "ASL" "APOD" "CYP2D6" "KLK6" ...data.frame` オブジェクトの検査
関数 head() と str() が、
データフレームの内容と構造をチェックするのに便利であることは、すでに説明した。
以下は、 データの内容/構造を知るための、 非網羅的な機能のリストである。
試してみよう!
**サイズ
- dim(rna)` - 行数を最初の 要素とし、列数を2番目の要素(オブジェクトの dimensions )とするベクトルを返す。
- nrow(rna)` - 行の数を返す。
- ncol(rna)` - 列数を返す。
**内容
- head(rna)` - 最初の6行を表示する。
- tail(rna)` - 最後の6行を表示する。
名前:
- names(rna)
- 列名を返す(data.frameオブジェクトのcolnames()` と同義)。 - rownames(rna)` - 行の名前を返す。
要約:
- str(rna)` - オブジェクトの構造と、 クラス、各カラムの長さと内容に関する情報。
-
summary(rna)- 各カラムの要約統計量。
注:これらの関数のほとんどは
“ジェネリック”であり、data.frame以外の
オブジェクトにも使用できます。
- クラス: データ・フレーム
- 行数:66465、列数:11:11
データフレームのインデックス化とサブセット化
rna`データフレームには行と列がある(2次元ある)。 、そこから特定のデータを抽出したい場合は、 「座標」を指定する必要がある。 行番号が最初に来て、 列番号がそれに続く。 しかし、これらの 座標を指定する方法が異なれば、異なるクラスの結果が得られることに注意されたい。
R
# データフレームの1列目の最初の要素(ベクトルとして)
rna[1, 1]
# 6列目の最初の要素(ベクトルとして)
rna[1, 6]
# データフレームの1列目の要素(ベクトルとして)
rna[, 1]
# データフレームの1列目の要素(data.フレームとして)
rna[1]
# 7列目の最初の3要素(ベクトルとして)
rna[1:3, 7]
# データフレームの3行目(data.frameとして)
rna[3, ]
# head_rna <- head(rna)
head_rna <- rna[1:6, ]
head_rna
1:10と10:1`は の例で、
の増加または減少の順序で整数の数値ベクトルを作成する特別な関数である。
詳しくは@ref(sec:genvec)を参照のこと。
また、「-」記号を使ってデータフレームの特定のインデックスを除外することもできる:
R
rna[, -1] ## 最初の列を除いたデータフレーム全体
rna[-c(7:66465), ] ## head(rna)と等価
データフレームは、インデックス(前に示したように)や 、列名を直接呼び出してサブセットすることもできる:
R
rna["gene"] # Result is a data.frame
rna[, "gene"] # Result is a vector
rna[["gene"]]. # 結果はベクトル
rna$gene # 結果はベクトルRStudio では、オートコンプリート機能を使用して、列の完全で 正しい名前を取得できます。
チャレンジ
データセット
rnaの 行 200 番目のデータのみを含むdata.frame(rna_200) を作成する。nrow()
がdata.frame`の行数を示していることに気づいただろうか?
この数字を使って、最初の
rnaデータフレームの最後の行だけを取り出す。tail()`を使った最後の行と比較し、 、期待に応えていることを確認する。
行番号の代わりに
nrow()を使って最後の行を取り出す。最後の行から新しいデータフレーム(
rna_last)を作成する。
rnaデータフレームの中央にある行を抽出するにはnrow()を使用する。 この行の内容をオブジェクトrna_middleに格納する。nrow()
と上記の-表記を組み合わせると、rnaデータセットの1行目から6行目までの 行だけを保持し、head(rna)`の 挙動を再現することができる。
R
## 1.
rna_200 <- rna[200, ]
## 2.
## Saving `n_rows` to improve readability and reduce duplication
n_rows <- nrow(rna)
rna_last <- rna[n_rows, ]
## 3.
rna_middle <- rna[n_rows / 2, ]
## 4.
rna_head <- rna[-(7:n_rows), ]
要因
要因はカテゴリーデータを表す。 これらは、 ラベルに関連付けられた整数として格納され、順序付けされたものであっても、順序付けされていないものであってもよい。 因子は文字ベクトルのように見える(そしてしばしば振舞う)が、 、実際にはRでは整数ベクトルとして扱われる。そのため、文字列として扱う場合は非常に 注意する必要がある。
いったん作成されたファクターは、あらかじめ定義された値のセット( 、_レベル_として知られている)しか含むことができない。 デフォルトでは、Rは常にレベルをアルファベット順( )でソートする。 例えば、2つのレベルを持つ因子があるとする:
R
セックス <- factor(c("male", "female", "female", "male", "female"))
Rは1をレベル"female"に、2をレベル
"male"に割り当てる(このベクトルの最初の要素
が"male"であるにもかかわらず、fがmの前に来るため)。
これは、 levels()
という関数を使うことで見ることができ、nlevels()
を使えばレベル数を知ることができる:
R
levels(sex)
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundR
nlevels(sex)
エラー
Error in eval(expr, envir, enclos): object 'sex' not found要因の順番が重要でない場合もあるが、 、意味がある(例えば、“low”、
“medium”、“high”)、視覚化が向上する、または特定のタイプの分析で必要である(
)ため、順番を指定したい場合もある。 ここで、
sexベクトルでレベルを並べ替える一つの方法は次のようになる:
R
sex ## current order
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundR
sex <- factor(sex, levels = c("male", "female"))
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundR
sex ## after re-ordering
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundRの記憶では、これらの因子は整数(1, 2, 3)、 で表現されるが、因子は自己
を記述するため、整数よりも情報量が多い。"女性"、"男性"は1、
2よりも説明的である。 どちらが “男性”ですか?
の整数データだけではわからないだろう。
一方、ファクターはこの情報を内蔵している。
特に、レベルが多い場合(例のデータセットの
遺伝子バイオタイプのような)に便利である。
データが因子として格納されているとき、各因子レベルによって表現されるオブザベーションの数
を素早く見るために、 plot() 関数を使うことができます。
データ中の男性 、女性の数を見てみよう。
R
プロット(性)
エラー
Error in プロット(性): could not find function "プロット"文字への変換
因数を文字ベクトルに変換する必要がある場合は、
as.character(x).
R
as.character(性)
エラー
Error in eval(expr, envir, enclos): object '性' not found要因の名称変更
これらのファクターの名前を変えたい場合は、 :
R
levels(sex)
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundR
levels(sex) <- c("M", "F")
エラー
Error: object 'sex' not foundR
sex
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundR
plot(sex)
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundR
levels(sex)
エラー
Error in eval(expr, envir, enclos): object 'sex' not foundR
levels(sex) <- c("Male", "Female")
エラー
Error: object 'sex' not found- 動物の名前の周りに引用符がない
- “feel”欄に1つ記入がない(おそらく毛皮の動物の1つ)。
- 体重欄のコンマが1つ足りない
R
country_climate <- data.frame(
country = c("Canada", "Panama", "South Africa", "Australia"),
climate = c("cold", "hot", "temperate", "hot/temperate"),
temperature = c(10, 30, 18, "15"),
northern_hemisphere = c(TRUE, TRUE, FALSE, "FALSE"),
has_kangaroo = c(FALSE, FALSE, FALSE, 1)
)
str(country_climate)
出力
'data.frame': 4 obs. of 5 variables:
$ country : chr "Canada" "Panama" "South Africa" "Australia"
$ climate : chr "cold" "hot" "temperate" "hot/temperate"
$ temperature : chr "10" "30" "18" "15"
$ northern_hemisphere: chr "TRUE" "TRUE" "FALSE" "FALSE"
$ has_kangaroo : num 0 0 0 1データ型の自動変換は、時に恵みであり、時に 迷惑である。 その存在を認識し、ルールを学び、Rでインポートするデータ がデータフレーム内で正しい型であることを再確認すること。 そうでない場合は、 データ入力中に生じたかもしれないミス(例えば、数字しか入っていないはずの列に文字が入っている)を検出するために、 を活用する。
詳しくはRStudio チュートリアルをご覧ください。
マトリックス
先に進む前に、データ・フレームについて学んだので、
パッケージのインストールを復習し、新しいデータ型、すなわち
matrix について学んでみよう。
data.frameのように、行列は行と 列の2つの次元を持つ。 しかし大きな違いは、行列のすべてのセルは 同じ型でなければならないということである:numeric、character、logical、…
その点で、行列は data.frame よりも vector
に近い。
行列のデフォルトコンストラクタは matrix である。
行列を構成するための の値のベクトルと、行および/または の列数1を取る。
下の図( )のように、値は列に沿ってソートされる。
R
m <- matrix(1:9, ncol = 3, nrow = 3)
m
出力
[,1] [,2] [,3]
[1,] 1 4 7
[2,] 2 5 8
[3,] 3 6 9R
##
ip <- installed.packages()
head(ip)
## View(ip)
## パッケージの数
nrow(ip)
## インストールされている全てのパッケージの名前
rownames(ip)
## 各パッケージに関する情報の種類
colnames(ip)
テストデータとして、大規模なランダムデータ行列を作成することはしばしば有用である。
以下の練習問題は、平均0、標準偏差 1の正規分布から無作為に
データを抽出して、そのような行列を作成するものです。これは
rnorm() 関数で行うことができます。
R
set.seed(123)
m <- matrix(rnorm(3000), ncol = 3)
dim(m)
出力
[1] 1000 3R
head(m)
出力
[,1] [,2] [,3]
[1,] -0.56047565 -0.99579872 -0.5116037
[2,] -0.23017749 -1.03995504 0.2369379
[3,] 1.55870831 -0.01798024 -0.5415892
[4,] 0.07050839 -0.13217513 1.2192276
[5,] 0.12928774 -2.54934277 0.1741359
[6,] 1.71506499 1.04057346 -0.6152683日付の書式設定
新人(そしてベテラン!)が抱える最も一般的な問題の1つである。 Rユーザーは、 、日付と時刻の情報を、 適切で分析中に使用可能な変数に変換している。
表計算ソフトの日付に関する注意
スプレッドシートの日付は通常、1つの列に格納される。 これが日付を記録する最も自然な方法のように思えるが、実際には ベストプラクティスではない。 スプレッドシート・アプリケーションは、 一見正しい方法で日付を表示する(人間の観察者には)。しかし、実際に どのように日付を処理し、保存するかには問題があるかもしれない。 YEAR、MONTH、DAYを別々のカラムに、または 、YEARとDAY-OF-YEARを別々のカラムに保存した方が、 より安全な場合が多い。
LibreOffice、Microsoft Excel、OpenOffice、 Gnumericなどの表計算プログラム。 は、 日付のエンコード方法が異なる(そしてしばしば互換性がない)(同じプログラムであっても、バージョンやオペレーティング システム間で)。 さらに、エクセルは日付でないものを 日付に変える (@Zeeberg:2004)ことができる。例えば、MAR1、DEC1、 OCT4のような名前や識別子である。 そのため、全体的に日付フォーマットを避けているのであれば、 、こうした問題を特定しやすくなる。
Data CarpentryレッスンのDates as data セクションでは、スプレッドシートを使った日付の落とし穴について、さらなる洞察 を提供しています。
lubridate** パッケージのymd()関数を使用します (**tidyverse
に属します。詳しくは こちら)。
. **lubridate**は**tidyverse**のインストールの一部として 。
tidyverse
(library(tidyverse)) をロードすると、コアパッケージ
(ほとんどのデータ分析で使用される パッケージ) がロードされます。
lubridate
しかし、コアTidyverseには属さないので、
library(lubridate)で明示的にロードする必要があります。
必要なパッケージをロードすることから始める:
R
library("lubridate")
ymd()は年、月、日を表すベクトルを受け取り、Dateベクトルに変換する。 DateはRが
、日付であると認識するデータのクラスであり、そのように操作することができる。
関数が必要とする引数は柔軟であるが、ベストプラクティスとしては、“YYYY-MM-DD”としてフォーマットされた文字
ベクトルである。
日付オブジェクトを作成し、構造を調べてみよう:
R
my_date <- ymd("2015-01-01")
str(my_date)
出力
Date[1:1], format: "2015-01-01"では、年、月、日を別々に貼り付けてみよう:
R
# sep は各コンポーネントを区切るために使う文字を示す
my_date <- ymd(paste("2015", "1", "1", sep = "-"))
str(my_date)
出力
Date[1:1], format: "2015-01-01"それでは、典型的な日付操作 のパイプラインに慣れておこう。
以下の小さなデータには、異なる year、
month、day 列に日付が格納されている。
R
x <- data.frame(year = c(1996, 1992, 1987, 1986, 2000, 1990, 2002, 1994, 1997, 1985),
month = c(2, 3, 10, 1, 8, 3, 4, 5, 5),
day = c(24, 8, 1, 5, 8, 17, 13, 10, 11, 24),
value = c(4, 5, 1, 9, 3, 8, 10, 2, 6, 7))
エラー
Error in data.frame(year = c(1996, 1992, 1987, 1986, 2000, 1990, 2002, : arguments imply differing number of rows: 10, 9R
x
エラー
Error in eval(expr, envir, enclos): object 'x' not found次に、この関数を x データセットに適用する。
まず、paste() を使って、x の
year、month、day 列から
の文字ベクトルを作る:
R
paste(x$year, x$month, x$day, sep = "-")
エラー
Error in eval(expr, envir, enclos): object 'x' not foundこの文字ベクトルは ymd()
の引数として使うことができる:
R
ymd(paste(x$year, x$month, x$day, sep = "-"))
エラー
Error in eval(expr, envir, enclos): object 'x' not found出来上がった Date ベクトルは x に
date という新しいカラムとして追加することができる:
R
x$date <- ymd(paste(x$year, x$month, x$day, sep = "-"))
エラー
Error in eval(expr, envir, enclos): object 'x' not foundR
str(x) # '日付'をクラスとする新しいカラムに注目。
エラー
Error in eval(expr, envir, enclos): object 'x' not foundすべてが正しく機能していることを確認しよう。
新しいカラムを検査する一つの方法は、summary()を使うことである:
R
summary(x$date)
エラー
Error in eval(expr, envir, enclos): object 'x' not foundymd()は、年、月、日を の順番で持つことを期待している。 例えば、日、月、年があれば、dmy()`
が必要になる。
R
dmy(paste(x$day, x$month, x$year, sep = "-"))
エラー
Error in eval(expr, envir, enclos): object 'x' not foundlubdridate`は、あらゆる日付のバリエーションに対応する多くの関数を持っている。
Rオブジェクトの概要
これまで、次元数( )、格納できるデータの種類( )が単一か複数かによって異なる、いくつかのタイプのRオブジェクトを見てきた:
- vector`:1次元(長さがある)、1種類のデータ。
- マトリックス`:2次元、単一データ型。
- data.frame`:2次元、1列1型。
リスト
まだ見ていないが、知っておくと便利なデータ型がリストだ。 、先ほどのまとめから続く:
-
list: 1つの次元で、各項目は異なるデータ 型にすることができる。
以下では、数値、文字、 行列、データフレーム、別のリストのベクトルを含むリストを作ってみよう:
R
l <- list(1:10, ## numeric
letters, ## character
installed.packages(), ## a matrix
cars, ## a data.frame
list(1, 2, 3)) ## a list
length(l)
出力
[1] 5R
str(l)
出力
List of 5
$ : int [1:10] 1 2 3 4 5 6 7 8 9 10
$ : chr [1:26] "a" "b" "c" "d" ...
$ : chr [1:187, 1:16] "askpass" "assertthat" "backports" "base64enc" ...
..- attr(*, "dimnames")=List of 2
.. ..$ : chr [1:187] "askpass" "assertthat" "backports" "base64enc" ...
.. ..$ : chr [1:16] "Package" "LibPath" "Version" "Priority" ...
$ :'data.frame': 50 obs. of 2 variables:
..$ speed: num [1:50] 4 4 7 7 8 9 10 10 10 11 ...
..$ dist : num [1:50] 2 10 4 22 16 10 18 26 34 17 ...
$ :List of 3
..$ : num 1
..$ : num 2
..$ : num 3リストのサブセットは []
を使って新しいサブリストをサブセットするか、[]]
を使ってそのリストの単一要素を取り出す(
リストに名前がついている場合は、インデックスか名前を使う)。
R
l[[1]]##
出力
[1] 1 2 3 4 5 6 7 8 9 10R
l[1:2] ## 長さ 2 のリスト
出力
[[1]]
[1] 1 2 3 4 5 6 7 8 9 10
[[2]]
[1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s"
[20] "t" "u" "v" "w" "x" "y" "z"R
l[1] ## 長さ 1 のリスト
出力
[[1]]
[1] 1 2 3 4 5 6 7 8 9 10表形式データのエクスポートと保存
read.table
ファミリーの関数を使って、テキストベースのスプレッドシートをRに読み込む方法を見てきた。
data.frameを テキストベースのスプレッドシートにエクスポートするには、 関数のwrite.tableセット(write.csv,write.delim, ...)を使用します。 これらはすべて、 エクスポートする変数と、エクスポートするファイルを指定する。 例えば、rnaのデータをdata_outputディレクトリのmy_rna.csv`
ファイルにエクスポートするには、次のように実行する:
R
write.csv(rna, file = "data_output/my_rna.csv")
この新しいcsvファイルは、
、Rに精通していない他の共同研究者と共有することができます。data.frameのフィールドの一部(例えば、“product”列を参照)にカンマがあるにもかかわらず、Rはデフォルトで
、各フィールドを引用符で囲みます。したがって、
、列の区切り文字としてカンマを使用しているにもかかわらず、
、Rに正しく読み込むことができます。
行数か列数のどちらかだけで十分で、もう一方は値の長さから推測できる。 値と行/列の数が合わない場合に何が起こるか試してみてください。↩︎
Content from dplyrによるデータの操作と分析
最終更新日:2024-09-08 | ページの編集
所要時間: 150分
概要
質問
- tidyverseメタパッケージを用いたRでのデータ分析
目的
- dplyr
** と **tidyr** パッケージの目的を説明する。 - データを操作するのに非常に便利な関数をいくつか説明する。
- ワイド表形式とロング表形式の概念を説明し、 、データ・フレームを一方の形式から他方の形式に変更する方法を見る。
- テーブルの結合方法を示す。
このエピソードは、Data Carpentriesの_Data Analysis and Visualisation in R for Ecologists_レッスンに基づいています。
dplyr**と**tidyr**を使ったデータ操作
ブラケット・サブセットは便利だが、煩雑で 、特に複雑な操作では読みにくい。
いくつかのパッケージは、データを操作する際に私たちの作業を大いに助けてくれる。
Rのパッケージは基本的に、
、より多くのことができるようにする追加関数のセットである。
いくつかのパッケージは、データを操作する際に私たちの作業を大いに助けてくれる。
Rのパッケージは基本的に、
、より多くのことができるようにする追加関数のセットである。
これまで使ってきた str() や data.frame()
などの関数は、Rに組み込まれています。パッケージをロードすることで、その他の
固有の関数にアクセスできるようになります。
初めてパッケージを使用する前に、
をマシンにインストールする必要がある。その後、 R
セッションでパッケージが必要になったら、毎回インポートする必要がある。
初めてパッケージを使用する前に、
をマシンにインストールする必要がある。その後、 R
セッションでパッケージが必要になったら、毎回インポートする必要がある。
dplyr\`** パッケージは、データ操作タスクのための強力なツールを提供します。 データフレームを直接操作できるように構築されており、多くの操作タスクが に最適化されている。 データフレームを直接操作できるように構築されており、多くの操作タスクが に最適化されている。
後述するように、 、特定の分析や視覚化を行うために、データフレームの形を変えたいことがある。 tidyr`**パッケージは、 、データの形を変えるというこの一般的な問題に対処し、 データを整然と操作するためのツールを提供する。
ワークショップの後、**dplyr**と**tidyr**についてもっと知りたい方は、
、こちらのhandy data transformation with をご覧ください。
- tidyverse
**パッケージは "umbrella-package "であり、 、データ解析のためのいくつかの便利なパッケージがインストールされます。 には、**tidyr**, dplyr**, **ggplot2, **tibble\`**などがあります。 これらのパッケージは、データを操作したり対話したりするのに役立ちます。 サブセット化、変換、 ビジュアライズなど、データを使ってさまざまなことができる。 サブセット化、変換、 ビジュアライズなど、データを使ってさまざまなことができる。
セットアップを行ったのであれば、すでにtidyverseパッケージがインストールされているはずです。 ライブラリから読み込んでみて、それがあるかどうか確認してください: ライブラリから読み込んでみて、それがあるかどうか確認してください:
R
## dplyr を含む tidyverse パッケージをロード
library("tidyverse")
tidyverse\`** パッケージをインストールするには、以下のようにタイプしてください:
R
BiocManager::install("tidyverse")
もし、**tidyverse**パッケージをインストールしなければならなかったなら、上記のlibrary()\`コマンドを使って、このRセッションでロードすることを忘れないでください!
tidyverseでデータをロードする
read.csv()の代わりに、tidyverseパッケージ **readr**の
read_csv()
関数(.の代わりに_があることに注意)を使ってデータを読み込みます。
R
rna <- read_csv("data/rnaseq.csv")
## データを見る
rna
出力
# A tibble: 32,428 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 1170 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 Apod GSM254… 36194 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
3 Cyp2d22 GSM254… 4060 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
4 Klk6 GSM254… 287 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
5 Fcrls GSM254… 85 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
6 Slc2a4 GSM254… 782 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
7 Exd2 GSM254… 1619 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 Gjc2 GSM254… 288 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
9 Plp1 GSM254… 43217 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
10 Gnb4 GSM254… 1071 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>データのクラスが “tibble”と呼ばれていることに注目してほしい。
Tibblesは、以前 で紹介したデータ・フレーム・オブジェクトの動作の一部を微調整している。 データ構造はデータフレームによく似ている。 我々の目的にとって、唯一の違いはそれだ:
各列のデータ型が列名の下に表示される。 <
dbl> は の小数点を持つ数値を保持するために定義されたデータ型である。 データで作業するとき、我々はしばしば、各因子または因子の組み合わせについて 見つかったオブザベーションの数を知りたい。 このタスクのために、**dplyr** はcount()\` を提供している。 例えば、感染したサンプルと感染していないサンプルそれぞれについて、 、データの行数をカウントしたい場合、次のようにする:これは、データの最初の数行と、 1画面に収まるだけの列数だけを印刷する。
これから、最も一般的な dplyr\` 関数のいくつかを学びます:
- select()\`: カラムのサブセット
-
filter(): 条件で行をサブセットする。 - mutate()\`: 他のカラムの情報を使って新しいカラムを作成する。
- group_by()
とsummarise()\`: グループ化されたデータの要約統計量を作成する。 - arrange()\`:結果の並べ替え
- count()\`: 離散値を数える
列の選択と行のフィルタリング
データフレームの列を選択するには select() を使う。
この関数の最初の引数 はデータフレーム (rna) で、続く
の引数は保持する列です。 Tibblesは、以前
で紹介したデータ・フレーム・オブジェクトの動作の一部を微調整している。
データ構造はデータフレームによく似ている。
我々の目的にとって、唯一の違いはそれだ:
R
select(rna, gene, sample, tissue, expression)
出力
# A tibble: 32,428 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545336 Cerebellum 1170
2 Apod GSM2545336 Cerebellum 36194
3 Cyp2d22 GSM2545336 Cerebellum 4060
4 Klk6 GSM2545336 Cerebellum 287
5 Fcrls GSM2545336 Cerebellum 85
6 Slc2a4 GSM2545336 Cerebellum 782
7 Exd2 GSM2545336 Cerebellum 1619
8 Gjc2 GSM2545336 Cerebellum 288
9 Plp1 GSM2545336 Cerebellum 43217
10 Gnb4 GSM2545336 Cerebellum 1071
# ℹ 32,418 more rows特定の列を除く*すべての列を選択するには、 その変数の前に”-“を付けて除外する。
R
select(rna, -tissue, -organism)
出力
# A tibble: 32,428 × 17
gene sample expression age sex infection strain time mouse ENTREZID
<chr> <chr> <dbl> <dbl> <chr> <chr> <chr> <dbl> <dbl> <dbl>
1 Asl GSM2545… 1170 8 Fema… Influenz… C57BL… 8 14 109900
2 Apod GSM2545… 36194 8 Fema… Influenz… C57BL… 8 14 11815
3 Cyp2d22 GSM2545… 4060 8 Fema… Influenz… C57BL… 8 14 56448
4 Klk6 GSM2545… 287 8 Fema… Influenz… C57BL… 8 14 19144
5 Fcrls GSM2545… 85 8 Fema… Influenz… C57BL… 8 14 80891
6 Slc2a4 GSM2545… 782 8 Fema… Influenz… C57BL… 8 14 20528
7 Exd2 GSM2545… 1619 8 Fema… Influenz… C57BL… 8 14 97827
8 Gjc2 GSM2545… 288 8 Fema… Influenz… C57BL… 8 14 118454
9 Plp1 GSM2545… 43217 8 Fema… Influenz… C57BL… 8 14 18823
10 Gnb4 GSM2545… 1071 8 Fema… Influenz… C57BL… 8 14 14696
# ℹ 32,418 more rows
# ℹ 7 more variables: product <chr>, ensembl_gene_id <chr>,
# external_synonym <chr>, chromosome_name <chr>, gene_biotype <chr>,
# phenotype_description <chr>, hsapiens_homolog_associated_gene_name <chr>これは rna の中の、 tissue と
organism 以外のすべての変数を選択する。
特定の条件に基づいて行を選択するには、filter()
を使用する:
R
filter(rna, sex == "Male")
出力
# A tibble: 14,740 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 626 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
2 Apod GSM254… 13021 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
3 Cyp2d22 GSM254… 2171 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
4 Klk6 GSM254… 448 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
5 Fcrls GSM254… 180 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
6 Slc2a4 GSM254… 313 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
7 Exd2 GSM254… 2366 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
8 Gjc2 GSM254… 310 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
9 Plp1 GSM254… 53126 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
10 Gnb4 GSM254… 1355 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
# ℹ 14,730 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>R
filter(rna, sex == "Male" & infection == "NonInfected")
出力
# A tibble: 4,422 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 535 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
2 Apod GSM254… 13668 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
3 Cyp2d22 GSM254… 2008 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
4 Klk6 GSM254… 1101 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
5 Fcrls GSM254… 375 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
6 Slc2a4 GSM254… 249 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
7 Exd2 GSM254… 3126 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
8 Gjc2 GSM254… 791 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
9 Plp1 GSM254… 98658 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
10 Gnb4 GSM254… 2437 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
# ℹ 4,412 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>ここで、このデータセットで解析されたマウス
遺伝子のヒトホモログに興味があるとしよう。 この情報は、
hsapiens_homolog_associated_gene_name という名前の
rna tibbleの 最後のカラムにある。
ここで、このデータセットで解析されたマウス
遺伝子のヒトホモログに興味があるとしよう。 この情報は、
hsapiens_homolog_associated_gene_name という名前の
rna tibbleの 最後のカラムにある。 簡単に視覚化するために、
、2つの列geneと
hsapiens_homolog_associated_gene_nameだけを含む新しいテーブルを作成する。
R
genes <- select(rna, gene, hsapiens_homolog_associated_gene_name)
genes
出力
# A tibble: 32,428 × 2
gene hsapiens_homolog_associated_gene_name
<chr> <chr>
1 Asl ASL
2 Apod APOD
3 Cyp2d22 CYP2D6
4 Klk6 KLK6
5 Fcrls FCRL2
6 Slc2a4 SLC2A4
7 Exd2 EXD2
8 Gjc2 GJC2
9 Plp1 PLP1
10 Gnb4 GNB4
# ℹ 32,418 more rowsマウス遺伝子の中にはヒトにホモログがないものもある。 これらは、
filter() と、 何かが NA かどうかを判定する
is.na() 関数を使って取得することができる。
R
filter(genes, is.na(hsapiens_homolog_associated_gene_name))
出力
# A tibble: 4,290 × 2
gene hsapiens_homolog_associated_gene_name
<chr> <chr>
1 Prodh <NA>
2 Tssk5 <NA>
3 Vmn2r1 <NA>
4 Gm10654 <NA>
5 Hexa <NA>
6 Sult1a1 <NA>
7 Gm6277 <NA>
8 Tmem198b <NA>
9 Adam1a <NA>
10 Ebp <NA>
# ℹ 4,280 more rowsヒトのホモログを持つマウス遺伝子だけを保持したい場合、
、結果を否定する”!“記号を挿入することができる。したがって、
、hsapiens_homolog_associated_gene_name is not an
NA となるすべての行を求めることになる。
R
filter(genes, !is.na(hsapiens_homolog_associated_gene_name))
出力
# A tibble: 28,138 × 2
gene hsapiens_homolog_associated_gene_name
<chr> <chr>
1 Asl ASL
2 Apod APOD
3 Cyp2d22 CYP2D6
4 Klk6 KLK6
5 Fcrls FCRL2
6 Slc2a4 SLC2A4
7 Exd2 EXD2
8 Gjc2 GJC2
9 Plp1 PLP1
10 Gnb4 GNB4
# ℹ 28,128 more rowsパイプ
選択とフィルタを同時に行いたい場合は? これを行うには、 、中間ステップ、ネストされた関数、パイプの3つの方法がある。 これを行うには、 、中間ステップ、ネストされた関数、パイプの3つの方法がある。
中間ステップでは、一時的なデータフレームを作成し、 、次の関数の入力として使用する:
R
rna2 <- filter(rna, sex == "Male")
rna3 <- select(rna2, gene, sample, tissue, expression)
rna3
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsこれは読みやすいが、 、個別に名前を付けなければならない中間オブジェクトがたくさんあるため、ワークスペースが散らかる可能性がある。 複数の 、それを把握するのは難しいかもしれない。 複数の 、それを把握するのは難しいかもしれない。
、関数を入れ子にすることもできる:
R
rna3 <- select(filter(rna, sex == "Male", gene, sample, tissue, expression))
エラー
Error in `filter()`:
ℹ In argument: `gene`.
Caused by error:
! `..2` must be a logical vector, not a character vector.R
rna3
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsこれは便利だが、 Rは式を内側から外側へと評価する(この場合、フィルタリングしてから選択する)ため、関数が入れ子になりすぎると読みにくくなることがある。
最後のオプションである_パイプ_は、Rに最近追加されたものである。パイプを使うと、ある関数の出力を 、次の関数に直接送ることができる。これは、同じデータセットに対して多くの処理を行う必要がある場合に便利である 。
ミューテート R のパイプは %>%
(magrittr パッケージで利用可能) または
|> (ベース R で利用可能) のように見えます。
RStudioを使用する場合は、
PCをお持ちの場合はCtrl+Shift+M、
Macをお持ちの場合はCmd+Shift+Mでパイプを
。
上記のコードでは、パイプを使って rna データセットをまず
filter() を通して sex が Male
である行を残し、次に select() を通して gene,
sample, tissue, expressioncolumns
だけを残すように送っている。
パイプ %>% はその左側にあるオブジェクトを受け取り、
その右側にある関数の最初の引数として直接渡します。 filter()
と select()
関数の引数として明示的にデータフレームを含める必要はもうありません。
R
rna %>%
filter(sex == "Male") %>%
select(gene, sample, tissue, expression)
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsパイプを “then”のように読むことが役に立つと思う人もいるだろう。
パイプを “then”のように読むことが役に立つと思う人もいるだろう。 例えば、
上の例では、データフレーム rna
を取得し、sex=="Male" の行を で
フィルターし、gene, sample,
tissue, expression の列を 選択
した。
dplyr\`**関数はそれ自体ではやや単純だが、 、パイプを使った線形ワークフローに組み合わせることで、 、データフレームのより複雑な操作を行うことができる。
この小さいバージョンのデータで新しいオブジェクトを作りたい場合、 、新しい名前を割り当てることができる:
R
rna3<- rna %>%
filter(sex == "Male") %>%
select(gene, sample, tissue, expression)
rna3
出力
# A tibble: 14,740 × 4
gene sample tissue expression
<chr> <chr> <chr> <dbl>
1 Asl GSM2545340 Cerebellum 626
2 Apod GSM2545340 Cerebellum 13021
3 Cyp2d22 GSM2545340 Cerebellum 2171
4 Klk6 GSM2545340 Cerebellum 448
5 Fcrls GSM2545340 Cerebellum 180
6 Slc2a4 GSM2545340 Cerebellum 313
7 Exd2 GSM2545340 Cerebellum 2366
8 Gjc2 GSM2545340 Cerebellum 310
9 Plp1 GSM2545340 Cerebellum 53126
10 Gnb4 GSM2545340 Cerebellum 1355
# ℹ 14,730 more rowsR
rna %>%
filter(expression > 50000,
sex == "Female",
time == 0 ) %>%
select(gene, sample, time, expression, age)
出力
# A tibble: 9 × 5
gene sample time expression age
<chr> <chr> <dbl> <dbl> <dbl>
1 Plp1 GSM2545337 0 101241 8
2 Atp1b1 GSM2545337 0 53260 8
3 Plp1 GSM2545338 0 96534 8
4 Atp1b1 GSM2545338 0 50614 8
5 Plp1 GSM2545348 0 102790 8
6 Atp1b1 GSM2545348 0 59544 8
7 Plp1 GSM2545353 0 71237 8
8 Glul GSM2545353 0 52451 8
9 Atp1b1 GSM2545353 0 61451 8ミューテート
例えば、単位変換をしたり、2つの
列の値の比率を求めたりするために、既存の
列の値に基づいて新しい列を作成したいことがよくあります。 これには
mutate() を使う。 これには mutate()
を使う。
時間単位の新しい列を作成する:
R
rna %>%
mutate(time_hours = time * 24) %>%
select(time, time_hours)
出力
# A tibble: 32,428 × 2
time time_hours
<dbl> <dbl>
1 8 192
2 8 192
3 8 192
4 8 192
5 8 192
6 8 192
7 8 192
8 8 192
9 8 192
10 8 192
# ℹ 32,418 more rowsまた、mutate()の同じ呼び出しの中で、最初の新しいカラムに基づいて2番目の新しいカラムを作成することもできる:
R
rna %>%
mutate(time_hours = time * 24,
time_mn = time_hours * 60) %>%
select(time, time_hours, time_mn)
出力
# A tibble: 32,428 × 3
time time_hours time_mn
<dbl> <dbl> <dbl>
1 8 192 11520
2 8 192 11520
3 8 192 11520
4 8 192 11520
5 8 192 11520
6 8 192 11520
7 8 192 11520
8 8 192 11520
9 8 192 11520
10 8 192 11520
# ℹ 32,418 more rowsチャレンジ
後述するように、
、特定の分析や視覚化を行うために、データフレームの形を変えたいことがある。
tidyr`**パッケージは、
、データの形を変えるというこの一般的な問題に対処し、
データを整然と操作するためのツールを提供する。 の値は対数変換する。
以下の 条件を満たす
rnaデータから新しいデータフレームを作成する:gene、chromosome_name、
phenotype_description、sample、expression\
列のみを含む。 の値は対数変換する。 このデータフレームは、
、性染色体に位置し、 phenotype_descriptionに関連し、log
expressionが5より高い遺伝子のみを含んでいなければならない。
ヒント:このデータフレームを 、どのようにコマンドを並べるべきか考えてみよう!
R
rna %>%
mutate(expression = log(expression)) %>%
select(gene, chromosome_name, phenotype_description, sample, expression) %>%
filter(chromosome_name == "X" | chromosome_name == "Y") %>%
filter(!is.na(phenotype_description)) %>%
filter(expression > 5)
出力
# A tibble: 649 × 5
gene chromosome_name phenotype_description sample expression
<chr> <chr> <chr> <chr> <dbl>
1 Plp1 X abnormal CNS glial cell morphology GSM25… 10.7
2 Slc7a3 X decreased body length GSM25… 5.46
3 Plxnb3 X abnormal coat appearance GSM25… 6.58
4 Rbm3 X abnormal liver morphology GSM25… 9.32
5 Cfp X abnormal cardiovascular system phys… GSM25… 6.18
6 Ebp X abnormal embryonic erythrocyte morp… GSM25… 6.68
7 Cd99l2 X abnormal cellular extravasation GSM25… 8.04
8 Piga X abnormal brain development GSM25… 6.06
9 Pim2 X decreased T cell proliferation GSM25… 7.11
10 Itm2a X no abnormal phenotype detected GSM25… 7.48
# ℹ 639 more rows分割-適用-結合データ分析
多くのデータ分析タスクは、
_split-apply-combine_パラダイムを使ってアプローチすることができる:データをグループに分割し、各グループにいくつかの
分析を適用し、その結果を組み合わせる。
**dplyr** はgroup_by()`
関数を使って、これを非常に簡単にしている。
**dplyr\*\* はgroup_by()`
関数を使って、これを非常に簡単にしている。
R
rna %>%
group_by(gene)
出力
# A tibble: 32,428 × 19
# Groups: gene [1,474]
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 1170 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 Apod GSM254… 36194 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
3 Cyp2d22 GSM254… 4060 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
4 Klk6 GSM254… 287 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
5 Fcrls GSM254… 85 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
6 Slc2a4 GSM254… 782 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
7 Exd2 GSM254… 1619 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 Gjc2 GSM254… 288 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
9 Plp1 GSM254… 43217 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
10 Gnb4 GSM254… 1071 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>group_by()関数はデータ処理を行わず、 データをサブセットにグループ化する。上の例では、 32428オブザベーションの最初のtibbleは、1474グループにgene\`
変数に基づいて分割される。
同様に、ティブルをサンプルごとにグループ分けすることもできる:
R
rna %>%
group_by(sample)
出力
# A tibble: 32,428 × 19
# Groups: sample [22]
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Asl GSM254… 1170 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 Apod GSM254… 36194 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
3 Cyp2d22 GSM254… 4060 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
4 Klk6 GSM254… 287 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
5 Fcrls GSM254… 85 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
6 Slc2a4 GSM254… 782 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
7 Exd2 GSM254… 1619 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 Gjc2 GSM254… 288 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
9 Plp1 GSM254… 43217 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
10 Gnb4 GSM254… 1071 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>ここで、最初の 32428 オブザベーションの tibble
は、sample 変数に基づいて、 22 グループに分割される。
いったんデータがグループ化されると、その後の操作は各グループに独立して 。
summarise()`関数
group_by()はsummarise()\` と一緒に使われることが多く、
は各グループを1行の要約に折りたたむ。
group_by()は、 **カテゴリー** 変数を含むカラム名を引数として取り、 統計のサマリーを計算します。 group_by()\
は、 カテゴリー 変数を含むカラム名を引数として取り、
統計のサマリーを計算します。
そこで、遺伝子ごとの平均「発現」を計算する:
エラー
Error: <text>:4:0: unexpected end of input
2: group_by(gene %>%
3: summarise(mean_expression = mean(expression))
^また、各サンプルの全遺伝子の平均発現量を計算することもできる:
エラー
Error: <text>:4:0: unexpected end of input
2: group_by(sample %>%
3: summarise(mean_expression = mean(expression))
^しかし、複数の列でグループ化することもできる:
R
rna %>%
group_by(gene, infection, time) %>%
summarise(mean_expression = mean(expression))
出力
`summarise()` has grouped output by 'gene', 'infection'. You can override using
the `.groups` argument.出力
# A tibble: 4,422 × 4
# Groups: gene, infection [2,948]
gene infection time mean_expression
<chr> <chr> <dbl> <dbl>
1 AI504432 InfluenzaA 4 1104.
2 AI504432 InfluenzaA 8 1014
3 AI504432 NonInfected 0 1034.
4 AW046200 InfluenzaA 4 152.
5 AW046200 InfluenzaA 8 81
6 AW046200 NonInfected 0 155.
7 AW551984 InfluenzaA 4 302.
8 AW551984 InfluenzaA 8 342.
9 AW551984 NonInfected 0 238
10 Aamp InfluenzaA 4 4870
# ℹ 4,412 more rowsいったんデータがグループ化されると、同じ (必ずしも同じ変数でなくてもよい)時間に複数の変数を要約することもできる。 例えば、遺伝子別、条件別の「発現」の中央値を示す 列を追加することができる: 例えば、遺伝子別、条件別の「発現」の中央値を示す 列を追加することができる:
R
rna %>%
group_by(gene, infection, time) %>%
summarise(mean_expression = mean(expression),
median_expression = median(expression))
出力
`summarise()` has grouped output by 'gene', 'infection'. You can override using
the `.groups` argument.出力
# A tibble: 4,422 × 5
# Groups: gene, infection [2,948]
gene infection time mean_expression median_expression
<chr> <chr> <dbl> <dbl> <dbl>
1 AI504432 InfluenzaA 4 1104. 1094.
2 AI504432 InfluenzaA 8 1014 985
3 AI504432 NonInfected 0 1034. 1016
4 AW046200 InfluenzaA 4 152. 144.
5 AW046200 InfluenzaA 8 81 82
6 AW046200 NonInfected 0 155. 163
7 AW551984 InfluenzaA 4 302. 245
8 AW551984 InfluenzaA 8 342. 287
9 AW551984 NonInfected 0 238 265
10 Aamp InfluenzaA 4 4870 4708
# ℹ 4,412 more rowsR
rna %>%
filter(gene == "Dok3") %>%
group_by(time) %>%
summarise(mean = mean(expression))
出力
# A tibble: 3 × 2
time mean
<dbl> <dbl>
1 0 169
2 4 156.
3 8 61 カウント
データで作業するとき、我々はしばしば、各因子または因子の組み合わせについて
見つかったオブザベーションの数を知りたい。
このタスクのために、**dplyr** はcount()` を提供している。
例えば、感染したサンプルと感染していないサンプルそれぞれについて、
、データの行数をカウントしたい場合、次のようにする:
R
rna %>%
count(infection)
出力
# A tibble: 2 × 2
infection n
<chr> <int>
1 InfluenzaA 22110
2 NonInfected 10318count()関数は、すでに見たことのある、変数でグループ化し、そのグループ内のオブザベーションの数をカウントして要約する、ということの省略記法です。 言い換えれば、rna
%>% count(infection)`は次のものと等価である:
言い換えれば、rna %>% count(infection)\は次のものと等価である:
R
rna %>%
group_by(infection) %>%
summarise(n = n())
出力
# A tibble: 2 × 2
infection n
<chr> <int>
1 InfluenzaA 22110
2 NonInfected 10318先ほどの例では、count()
を使って、_1つの_要因(つまり感染)について
、行数/観察数を数えている。 先ほどの例では、count()
を使って、_1つの_要因(つまり感染)について
、行数/観察数を数えている。
もし、感染と時間のような_要因の組み合わせ_をカウントしたいのであれば、
、count()の引数として1つ目と2つ目の要因を指定することになる:
R
rna %>%
count(infection, time)
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 InfluenzaA 4 11792
2 InfluenzaA 8 10318
3 NonInfected 0 10318これと等価である:
R
rna %>%
group_by(infection, time) %>%
summarise(n = n())
出力
`summarise()` has grouped output by 'infection'. You can override using the
`.groups` argument.出力
# A tibble: 3 × 3
# Groups: infection [2]
infection time n
<chr> <dbl> <int>
1 InfluenzaA 4 11792
2 InfluenzaA 8 10318
3 NonInfected 0 10318比較を容易にするために、結果を並べ替えると便利なことがある。 arrange()`を使って表を並べ替えることができる。 例えば、上の表を時間順に並べたいとする:
R
rna %>%
count(infection, time) %>%
arrange(time)
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 NonInfected 0 10318
2 InfluenzaA 4 11792
3 InfluenzaA 8 10318あるいは回数で:
R
rna %>%
count(infection, time) %>%
arrange(n)
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 InfluenzaA 8 10318
2 NonInfected 0 10318
3 InfluenzaA 4 11792降順にソートするには、desc()関数を追加する必要がある:
R
rna %>%
count(infection, time) %>%
arrange(desc(n))
出力
# A tibble: 3 × 3
infection time n
<chr> <dbl> <int>
1 InfluenzaA 4 11792
2 InfluenzaA 8 10318
3 NonInfected 0 10318R
##
rna %>%
count(sample)
## 2.
rna %>%
group_by(sample) %>%
summary(seq_depth = sum(expression)) %>%
arrange(desc(seq_depth))
## 3.
rna %>%
filter(sample == "GSM2545336") %>%
count(gene_biotype) %>%
arrange(desc(n))
## 4.
rna %>%
filter(phenotype_description == "abnormal DNA methylation") %>%
group_by(gene, time) %>%
summary(mean_expression = mean(log(expression)) %>%
arrange()エラー
Error: <text>:20:0: unexpected end of input
18: summary(mean_expression = mean(log(expression)) %>%
19: arrange()
^データの再構築
rnatibble の行には、geneとsample\`
という2つの変数の組み合わせに関連付けられた発現値(単位)が格納されている。
比較を容易にするために、結果を並べ替えると便利なことがある。 arrange()\`を使って表を並べ替えることができる。 例えば、上の表を時間順に並べたいとする: または遺伝子(gene_biotype, ENTREZ_ID, product, …)。 その他の列はすべて、 (生物、年齢、性別、…)のいずれかを記述する変数に対応している。 または遺伝子(gene_biotype, ENTREZ_ID, product, …)。 遺伝子やサンプルによって変化しない変数は、すべての行で同じ値を持つ。
R
rna %>%
arrange(gene)
出力
# A tibble: 32,428 × 19
gene sample expression organism age sex infection strain time tissue
<chr> <chr> <dbl> <chr> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 AI504432 GSM25… 1230 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
2 AI504432 GSM25… 1085 Mus mus… 8 Fema… NonInfec… C57BL… 0 Cereb…
3 AI504432 GSM25… 969 Mus mus… 8 Fema… NonInfec… C57BL… 0 Cereb…
4 AI504432 GSM25… 1284 Mus mus… 8 Fema… Influenz… C57BL… 4 Cereb…
5 AI504432 GSM25… 966 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
6 AI504432 GSM25… 918 Mus mus… 8 Male Influenz… C57BL… 8 Cereb…
7 AI504432 GSM25… 985 Mus mus… 8 Fema… Influenz… C57BL… 8 Cereb…
8 AI504432 GSM25… 972 Mus mus… 8 Male NonInfec… C57BL… 0 Cereb…
9 AI504432 GSM25… 1000 Mus mus… 8 Fema… Influenz… C57BL… 4 Cereb…
10 AI504432 GSM25… 816 Mus mus… 8 Male Influenz… C57BL… 4 Cereb…
# ℹ 32,418 more rows
# ℹ 9 more variables: mouse <dbl>, ENTREZID <dbl>, product <chr>,
# ensembl_gene_id <chr>, external_synonym <chr>, chromosome_name <chr>,
# gene_biotype <chr>, phenotype_description <chr>,
# hsapiens_homolog_associated_gene_name <chr>この構造はlong-formatと呼ばれ、1つのカラムにはすべての値、
、もう1つのカラムには値のコンテキストが列挙されている。
場合によっては、long-formatは実際には
“human-readable”ではなく、別のフォーマット、
wide-formatがよりコンパクトにデータを表現する方法として好まれる。
これは通常、科学者が
、行が遺伝子、列がサンプルを表す行列として見るのに慣れている遺伝子発現値の場合である。
これは通常、科学者が
、行が遺伝子、列がサンプルを表す行列として見るのに慣れている遺伝子発現値の場合である。
このフォーマットでは、 、サンプル内の遺伝子発現レベルとサンプル間の遺伝子発現レベル の関係を調べることができる。
出力
# A tibble: 1,474 × 23
gene GSM2545336 GSM2545337 GSM2545338 GSM2545339 GSM2545340 GSM2545341
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Asl 1170 361 400 586 626 988
2 Apod 36194 10347 9173 10620 13021 29594
3 Cyp2d22 4060 1616 1603 1901 2171 3349
4 Klk6 287 629 641 578 448 195
5 Fcrls 85 233 244 237 180 38
6 Slc2a4 782 231 248 265 313 786
7 Exd2 1619 2288 2235 2513 2366 1359
8 Gjc2 288 595 568 551 310 146
9 Plp1 43217 101241 96534 58354 53126 27173
10 Gnb4 1071 1791 1867 1430 1355 798
# ℹ 1,464 more rows
# ℹ 16 more variables: GSM2545342 <dbl>, GSM2545343 <dbl>, GSM2545344 <dbl>,
# GSM2545345 <dbl>, GSM2545346 <dbl>, GSM2545347 <dbl>, GSM2545348 <dbl>,
# GSM2545349 <dbl>, GSM2545350 <dbl>, GSM2545351 <dbl>, GSM2545352 <dbl>,
# GSM2545353 <dbl>, GSM2545354 <dbl>, GSM2545362 <dbl>, GSM2545363 <dbl>,
# GSM2545380 <dbl>rnaの遺伝子発現値をワイドフォーマットに変換するには、 、sample\`カラムの値が
、カラム変数の名前になる新しいテーブルを作成する必要がある。
ここでの重要なポイントは、我々はまだ 、整然としたデータ構造に従っているが、 、興味のある観察に従ってデータを整形したということである:遺伝子ごと、サンプルごとに記録する代わりに、遺伝子ごとの発現レベル 。
逆の変換は、列名を新しい変数の値( )に変換することである。
pivot_longer() と pivot_wider() の2つの
tidyr 関数を使って、これらの変換を行うことができます( こちら
を参照してください)( )。
より広いフォーマットへのデータのピボット
rnaの最初の3列を選択し、pivot_wider()\`
を使ってデータをワイドフォーマットに変換してみよう。
R
rna_exp <- rna %>%
select(gene, sample, expression)
rna_exp
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Cyp2d22 GSM2545336 4060
4 Klk6 GSM2545336 287
5 Fcrls GSM2545336 85
6 Slc2a4 GSM2545336 782
7 Exd2 GSM2545336 1619
8 Gjc2 GSM2545336 288
9 Plp1 GSM2545336 43217
10 Gnb4 GSM2545336 1071
# ℹ 32,418 more rowspivot_wider\`は主に3つの引数を取る:
- 変換されるデータ;
- the
names_from: その値が新しいカラム の名前になるカラム; - value_from\`: 新しいカラム を埋める値。
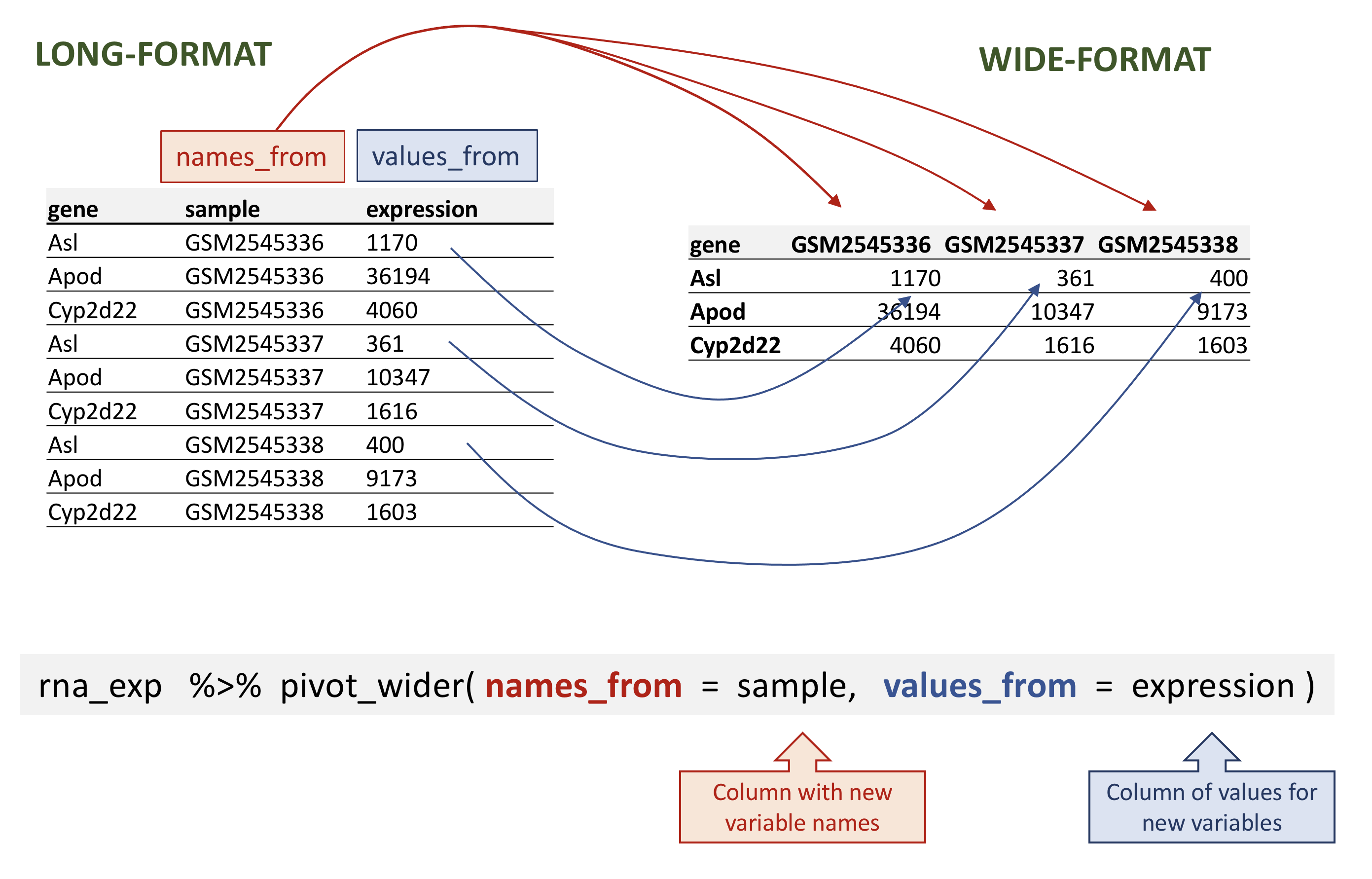
rna data.
R
rna_wide <- rna_exp %>%
pivot_wider(names_from = sample,
values_from = expression)
rna_wide
出力
# A tibble: 1,474 × 23
gene GSM2545336 GSM2545337 GSM2545338 GSM2545339 GSM2545340 GSM2545341
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Asl 1170 361 400 586 626 988
2 Apod 36194 10347 9173 10620 13021 29594
3 Cyp2d22 4060 1616 1603 1901 2171 3349
4 Klk6 287 629 641 578 448 195
5 Fcrls 85 233 244 237 180 38
6 Slc2a4 782 231 248 265 313 786
7 Exd2 1619 2288 2235 2513 2366 1359
8 Gjc2 288 595 568 551 310 146
9 Plp1 43217 101241 96534 58354 53126 27173
10 Gnb4 1071 1791 1867 1430 1355 798
# ℹ 1,464 more rows
# ℹ 16 more variables: GSM2545342 <dbl>, GSM2545343 <dbl>, GSM2545344 <dbl>,
# GSM2545345 <dbl>, GSM2545346 <dbl>, GSM2545347 <dbl>, GSM2545348 <dbl>,
# GSM2545349 <dbl>, GSM2545350 <dbl>, GSM2545351 <dbl>, GSM2545352 <dbl>,
# GSM2545353 <dbl>, GSM2545354 <dbl>, GSM2545362 <dbl>, GSM2545363 <dbl>,
# GSM2545380 <dbl>デフォルトでは、pivot_wider() 関数は欠損値に対して
NA を追加することに注意してください。
何らかの理由で、あるサンプルで 、いくつかの遺伝子の発現値が欠落していたとしよう。 何らかの理由で、あるサンプルで 、いくつかの遺伝子の発現値が欠落していたとしよう。 以下の架空の例では、遺伝子Cyp2d22の発現値はGSM2545338サンプルの 。
R
rna_with_missing_values <- rna %>%
select(gene, sample, expression) %>%
filter(gene %in% c("Asl", "Apod", "Cyp2d22")) %>%
filter(sample %in% c("GSM2545336", "GSM2545337", "GSM2545338")) %>%
arrange(sample) %>%
filter(!(gene == "Cyp2d22" & sample != "GSM2545338"))
rna_with_missing_values
出力
# A tibble: 7 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Asl GSM2545337 361
4 Apod GSM2545337 10347
5 Asl GSM2545338 400
6 Apod GSM2545338 9173
7 Cyp2d22 GSM2545338 1603デフォルトでは、pivot_wider()関数は、
の値が見つからない場合に NA を追加する。 これは、
pivot_wider() 関数の values_fill
引数でパラメータ化できる。 summarise()\`関数
R
rna_with_missing_values %>%
pivot_wider(names_from = sample,
values_from = expression)
出力
# A tibble: 3 × 4
gene GSM2545336 GSM2545337 GSM2545338
<chr> <dbl> <dbl> <dbl>
1 Asl 1170 361 400
2 Apod 36194 10347 9173
3 Cyp2d22 NA NA 1603R
rna_with_missing_values %>%
pivot_wider(names_from = sample,
values_from = expression,
values_fill = 0)
出力
# A tibble: 3 × 4
gene GSM2545336 GSM2545337 GSM2545338
<chr> <dbl> <dbl> <dbl>
1 Asl 1170 361 400
2 Apod 36194 10347 9173
3 Cyp2d22 0 0 1603データを長いフォーマットにピボットする
逆の状況では、カラム名を使い、 、新しい変数のペアに変えている。 一方の変数はカラム名を の値で表し、もう一方の変数にはカラム名に関連付けられている以前の値 が格納されている。 一方の変数はカラム名を の値で表し、もう一方の変数にはカラム名に関連付けられている以前の値 が格納されている。
pivot_longer()\`は主に4つの引数を取る:
- 変換されるデータ;
- names_to\`: の現在のカラム名で作成したい新しいカラム名;
- value_to\`: 作成したい新しいカラム名で、 の現在の値を格納する;
- 変数
names_toとvalues_toに格納する(または削除する)列の名前。

rnaデータのロングピボット。
rna_wideからrna_longを再作成するには、sampleというキーとexpressionという値を作成し、gene以外のすべてのカラム 。ここでは、gene`カラム
をマイナス記号で削除する。
ここで、新しい変数名がどのように引用されるかに注目してください。
R
rna_long<- rna_wide %>%
pivot_longer(names_to = "sample",
values_to = "expression",
-gene)
rna_long
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Asl GSM2545339 586
5 Asl GSM2545340 626
6 Asl GSM2545341 988
7 Asl GSM2545342 836
8 Asl GSM2545343 535
9 Asl GSM2545344 586
10 Asl GSM2545345 597
# ℹ 32,418 more rowsまた、 、どのようなカラムを含めるかという指定も使えたはずだ。
これは、 のカラムが多数あり、
のままにしておくよりも、何を集めるかを指定する方が簡単な場合に便利である。
また、 、どのようなカラムを含めるかという指定も使えたはずだ。 これは、
のカラムが多数あり、
のままにしておくよりも、何を集めるかを指定する方が簡単な場合に便利である。
ここで、starts_with()関数を使えば、
のサンプル名をすべてリストアップすることなく取得することができる!
もう一つの可能性は : 演算子を使うことである!
もう一つの可能性は : 演算子を使うことである!
R
rna_wide %>%
pivot_longer(names_to = "sample",
values_to = "expression",
cols = starts_with("GSM"))
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Asl GSM2545339 586
5 Asl GSM2545340 626
6 Asl GSM2545341 988
7 Asl GSM2545342 836
8 Asl GSM2545343 535
9 Asl GSM2545344 586
10 Asl GSM2545345 597
# ℹ 32,418 more rowsR
rna_wide %>%
pivot_longer(names_to = "sample",
values_to = "expression",
GSM2545336:GSM2545380)
出力
# A tibble: 32,428 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Asl GSM2545339 586
5 Asl GSM2545340 626
6 Asl GSM2545341 988
7 Asl GSM2545342 836
8 Asl GSM2545343 535
9 Asl GSM2545344 586
10 Asl GSM2545345 597
# ℹ 32,418 more rowsワイドフォーマットで欠損値があった場合、新しいロングフォーマットではNAが
。
前回の欠損値を含む架空のティブルを思い出してほしい:
R
rna_with_missing_values
出力
# A tibble: 7 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Asl GSM2545337 361
4 Apod GSM2545337 10347
5 Asl GSM2545338 400
6 Apod GSM2545338 9173
7 Cyp2d22 GSM2545338 1603R
wide_with_NA<- rna_with_missing_values %>%
pivot_wider(names_from = sample,
values_from = expression)
wide_with_NA
出力
# A tibble: 3 × 4
gene GSM2545336 GSM2545337 GSM2545338
<chr> <dbl> <dbl> <dbl>
1 Asl 1170 361 400
2 Apod 36194 10347 9173
3 Cyp2d22 NA NA 1603R
wide_with_NA %>%
pivot_longer(names_to = "sample",
values_to = "expression",
-gene)
出力
# A tibble: 9 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Asl GSM2545337 361
3 Asl GSM2545338 400
4 Apod GSM2545336 36194
5 Apod GSM2545337 10347
6 Apod GSM2545338 9173
7 Cyp2d22 GSM2545336 NA
8 Cyp2d22 GSM2545337 NA
9 Cyp2d22 GSM2545338 1603より幅の広い、より長いフォーマットへの移行は、データセットのバランスをとるのに有効な方法である。 、どの複製も同じ構成になる。
R
rna1<- rna %>%
select(gene, mouse, expression) %>%
pivot_wider(names_from = mouse, values_from = expression)
rna1
出力
# A tibble: 1,474 × 23
gene `14` `9` `10` `15` `18` `6` `5` `11` `22` `13` `23`
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 Asl 1170 361 400 586 626 988 836 535 586 597 938
2 Apod 36194 10347 9173 10620 13021 29594 24959 13668 13230 15868 27769
3 Cyp2d22 4060 1616 1603 1901 2171 3349 3122 2008 2254 2277 2985
4 Klk6 287 629 641 578 448 195 186 1101 537 567 327
5 Fcrls 85 233 244 237 180 38 68 375 199 177 89
6 Slc2a4 782 231 248 265 313 786 528 249 266 357 654
7 Exd2 1619 2288 2235 2513 2366 1359 1474 3126 2379 2173 1531
8 Gjc2 288 595 568 551 310 146 186 791 454 370 240
9 Plp1 43217 101241 96534 58354 53126 27173 28728 98658 61356 61647 38019
10 Gnb4 1071 1791 1867 1430 1355 798 806 2437 1394 1554 960
# ℹ 1,464 more rows
# ℹ 11 more variables: `24` <dbl>, `8` <dbl>, `7` <dbl>, `1` <dbl>, `16` <dbl>,
# `21` <dbl>, `4` <dbl>, `2` <dbl>, `20` <dbl>, `12` <dbl>, `19` <dbl>R
rna1 %>%
pivot_longer(names_to = "mouse_id", values_to = "counts", -gene)
出力
# A tibble: 32,428 × 3
gene mouse_id counts
<chr> <chr> <dbl>
1 Asl 14 1170
2 Asl 9 361
3 Asl 10 400
4 Asl 15 586
5 Asl 18 626
6 Asl 6 988
7 Asl 5 836
8 Asl 11 535
9 Asl 22 586
10 Asl 13 597
# ℹ 32,418 more rowsまず、 男性と女性のサンプルから、XとYの連鎖遺伝子の平均発現量を計算してみよう…
R
rna %>%
filter(chromosome_name == "Y" | chromosome_name == "X") %>%
group_by(sex, chromosome_name) %>%
summise(mean = mean(expression))
エラー
Error in summise(., mean = mean(expression)): could not find function "summise"そして、表をワイドフォーマットにピボットする
R
rna_1<- rna %>%
filter(chromosome_name == "Y" | chromosome_name == "X") %>%
group_by(sex, chromosome_name) %>%
summise(mean = mean(expression)) %>%
pivot_wider(names_from = sex,
values_from = mean)
エラー
Error in summise(., mean = mean(expression)): could not find function "summise"R
rna_1
エラー
Error in eval(expr, envir, enclos): object 'rna_1' not found各行が一意な chromosome_name と gender
の組み合わせになるように、このデータフレームを
pivot_longer() で変換する。
R
rna_1 %>%
pivot_longer(names_to = "gender",
values_to = "mean",
-chromosome_name)
エラー
Error in eval(expr, envir, enclos): object 'rna_1' not foundまず、遺伝子別、時間別の平均発現量を計算してみよう。
R
rna %>%
group_by(gene, time) %>%
summarise(mean_exp = mean(expression))
出力
`summarise()` has grouped output by 'gene'. You can override using the
`.groups` argument.出力
# A tibble: 4,422 × 3
# Groups: gene [1,474]
gene time mean_exp
<chr> <dbl> <dbl>
1 AI504432 0 1034.
2 AI504432 4 1104.
3 AI504432 8 1014
4 AW046200 0 155.
5 AW046200 4 152.
6 AW046200 8 81
7 AW551984 0 238
8 AW551984 4 302.
9 AW551984 8 342.
10 Aamp 0 4603.
# ℹ 4,412 more rowspivot_wider()関数を使用する前に
R
rna_time<- rna %>%
group_by(gene, time) %>%
summarise(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp)
出力
`summarise()` has grouped output by 'gene'. You can override using the
`.groups` argument.R
rna_time
出力
# A tibble: 1,474 × 4
# Groups: gene [1,474]
gene `0` `4` `8`
<chr> <dbl> <dbl> <dbl>
1 AI504432 1034. 1104. 1014
2 AW046200 155. 152. 81
3 AW551984 238 302. 342.
4 Aamp 4603. 4870 4763.
5 Abca12 5.29 4.25 4.14
6 Abcc8 2576. 2609. 2292.
7 Abhd14a 591. 547. 432.
8 Abi2 4881. 4903. 4945.
9 Abi3bp 1175. 1061. 762.
10 Abl2 2170. 2078. 2131.
# ℹ 1,464 more rowsこれにより、数字で始まるカラム名を持つティブルが生成されることに注意。 これにより、数字で始まるカラム名を持つティブルが生成されることに注意。 タイムポイントに対応するカラムを選択したい場合、 、カラム名を直接使うことはできない。 列4を選択するとどうなるか? 列4を選択するとどうなるか?
R
rna %>%
group_by(gene, time) %>%
summary(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp) %>%
select(gene, 4)
エラー
Error in UseMethod("pivot_wider"): no applicable method for 'pivot_wider' applied to an object of class "table"タイムポイント4を選択するには、“˶\`” というバックスティックを付けたカラム名を引用しなければならない。
R
rna %>%
group_by(gene, time) %>%
summarise(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp) %>%
select(gene, `4`)
出力
`summarise()` has grouped output by 'gene'. You can override using the
`.groups` argument.出力
# A tibble: 1,474 × 2
# Groups: gene [1,474]
gene `4`
<chr> <dbl>
1 AI504432 1104.
2 AW046200 152.
3 AW551984 302.
4 Aamp 4870
5 Abca12 4.25
6 Abcc8 2609.
7 Abhd14a 547.
8 Abi2 4903.
9 Abi3bp 1061.
10 Abl2 2078.
# ℹ 1,464 more rows、数字で始まらない名前を選択する:
R
rna %>%
group_by(gene, time) %>%
summary(mean_exp = mean(expression)) %>%
pivot_wider(names_from = time,
values_from = mean_exp) %>%
rename("time0" = `0`, "time4" = `4`, "time8" = `8`) %>%
select(gene, time4)
エラー
Error in UseMethod("pivot_wider"): no applicable method for 'pivot_wider' applied to an object of class "table"rna_time tibbleから開始する:
R
rna_time
出力
# A tibble: 1,474 × 4
# Groups: gene [1,474]
gene `0` `4` `8`
<chr> <dbl> <dbl> <dbl>
1 AI504432 1034. 1104. 1014
2 AW046200 155. 152. 81
3 AW551984 238 302. 342.
4 Aamp 4603. 4870 4763.
5 Abca12 5.29 4.25 4.14
6 Abcc8 2576. 2609. 2292.
7 Abhd14a 591. 547. 432.
8 Abi2 4881. 4903. 4945.
9 Abi3bp 1175. 1061. 762.
10 Abl2 2170. 2078. 2131.
# ℹ 1,464 more rowsフォールドチェンジを計算する:
R
rna_time %>%
mutate(time_8_vs_0 = `8` / `0`, time_8_vs_4 = `8` / `4`)
出力
# A tibble: 1,474 × 6
# Groups: gene [1,474]
gene `0` `4` `8` time_8_vs_0 time_8_vs_4
<chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 AI504432 1034. 1104. 1014 0.981 0.918
2 AW046200 155. 152. 81 0.522 0.532
3 AW551984 238 302. 342. 1.44 1.13
4 Aamp 4603. 4870 4763. 1.03 0.978
5 Abca12 5.29 4.25 4.14 0.784 0.975
6 Abcc8 2576. 2609. 2292. 0.889 0.878
7 Abhd14a 591. 547. 432. 0.731 0.791
8 Abi2 4881. 4903. 4945. 1.01 1.01
9 Abi3bp 1175. 1061. 762. 0.649 0.719
10 Abl2 2170. 2078. 2131. 0.982 1.03
# ℹ 1,464 more rowsそして、pivot_longer()関数を使用する:
R
rna_time %>%
mutate(time_8_vs_0 = `8` / `0`, time_8_vs_4 = `8` / `4`) %>%
pivot_longer(names_to = "comparisons",
values_to = "Fold_changes",
time_8_vs_0:time_8_vs_4)
出力
# A tibble: 2,948 × 6
# Groups: gene [1,474]
gene `0` `4` `8` comparisons Fold_changes
<chr> <dbl> <dbl> <dbl> <chr> <dbl>
1 AI504432 1034. 1104. 1014 time_8_vs_0 0.981
2 AI504432 1034. 1104. 1014 time_8_vs_4 0.918
3 AW046200 155. 152. 81 time_8_vs_0 0.522
4 AW046200 155. 152. 81 time_8_vs_4 0.532
5 AW551984 238 302. 342. time_8_vs_0 1.44
6 AW551984 238 302. 342. time_8_vs_4 1.13
7 Aamp 4603. 4870 4763. time_8_vs_0 1.03
8 Aamp 4603. 4870 4763. time_8_vs_4 0.978
9 Abca12 5.29 4.25 4.14 time_8_vs_0 0.784
10 Abca12 5.29 4.25 4.14 time_8_vs_4 0.975
# ℹ 2,938 more rowsテーブルの結合
実生活の多くの場面で、データは複数のテーブルにまたがっている。 通常このようなことが起こるのは、異なる情報源から異なるタイプの情報が 収集されるからである。 通常このようなことが起こるのは、異なる情報源から異なるタイプの情報が 収集されるからである。
分析によっては、2つ以上のテーブル( )のデータを、すべてのテーブルに共通するカラム( )に基づいて1つのデータフレームにまとめることが望ましい場合がある。
dplyr\` パッケージは、指定されたカラム内のマッチに基づいて、2つの データフレームを結合するための結合関数のセットを提供する。 ここでは、 、結合について簡単に紹介する。 詳しくは、 テーブル ジョインの章を参照されたい。 データ変換チート シート 、テーブル結合に関する簡単な概要も提供している。 ここでは、 、結合について簡単に紹介する。 詳しくは、 テーブル ジョインの章を参照されたい。 データ変換チート シート 、テーブル結合に関する簡単な概要も提供している。
、元のrnaテーブルをサブセットして作成し、
、3つのカラムと10行だけを残す。
R
rna_mini<- rna %>%
select(gene, sample, expression) %>%
head(10)
rna_mini
出力
# A tibble: 10 × 3
gene sample expression
<chr> <chr> <dbl>
1 Asl GSM2545336 1170
2 Apod GSM2545336 36194
3 Cyp2d22 GSM2545336 4060
4 Klk6 GSM2545336 287
5 Fcrls GSM2545336 85
6 Slc2a4 GSM2545336 782
7 Exd2 GSM2545336 1619
8 Gjc2 GSM2545336 288
9 Plp1 GSM2545336 43217
10 Gnb4 GSM2545336 10712番目のテーブルannot1には、遺伝子と
gene_descriptionの2つのカラムがある。
2番目のテーブルannot1には、遺伝子と
gene_descriptionの2つのカラムがある。 download
annot1.csv
リンクをクリックしてdata/フォルダに移動するか、
以下のRコードを使って直接フォルダにダウンロードすることができる。
R
download.file(url = "https://carpentries-incubator.github.io/bioc-intro/data/annot1.csv",
destfile = "data/annot1.csv")
annot1 <- read_csv(file = "data/annot1.csv")
annot1
出力
# A tibble: 10 × 2
gene gene_description
<chr> <chr>
1 Cyp2d22 cytochrome P450, family 2, subfamily d, polypeptide 22 [Source:MGI S…
2 Klk6 kallikrein related-peptidase 6 [Source:MGI Symbol;Acc:MGI:1343166]
3 Fcrls Fc receptor-like S, scavenger receptor [Source:MGI Symbol;Acc:MGI:19…
4 Plp1 proteolipid protein (myelin) 1 [Source:MGI Symbol;Acc:MGI:97623]
5 Exd2 exonuclease 3'-5' domain containing 2 [Source:MGI Symbol;Acc:MGI:192…
6 Apod apolipoprotein D [Source:MGI Symbol;Acc:MGI:88056]
7 Gnb4 guanine nucleotide binding protein (G protein), beta 4 [Source:MGI S…
8 Slc2a4 solute carrier family 2 (facilitated glucose transporter), member 4 …
9 Asl argininosuccinate lyase [Source:MGI Symbol;Acc:MGI:88084]
10 Gjc2 gap junction protein, gamma 2 [Source:MGI Symbol;Acc:MGI:2153060] ここで、dplyr パッケージの full_join()
関数を使用して、これら2つのテーブルを、すべての
変数を含む1つのテーブルに結合したいと思います。
関数は、最初のテーブルと2番目のテーブルの列
に一致する共通変数を自動的に見つける。
この場合、geneは共通の 。 このような変数をキーと呼ぶ。
キーは、
オブザベーションを異なるテーブル間でマッチさせるために使用される。
関数は、最初のテーブルと2番目のテーブルの列
に一致する共通変数を自動的に見つける。
この場合、geneは共通の 。 このような変数をキーと呼ぶ。
キーは、
オブザベーションを異なるテーブル間でマッチさせるために使用される。
R
full_join(rna_mini, annot1)
出力
Joining with `by = join_by(gene)`出力
# A tibble: 10 × 4
gene sample expression gene_description
<chr> <chr> <dbl> <chr>
1 Asl GSM2545336 1170 argininosuccinate lyase [Source:MGI Symbol;Acc…
2 Apod GSM2545336 36194 apolipoprotein D [Source:MGI Symbol;Acc:MGI:88…
3 Cyp2d22 GSM2545336 4060 cytochrome P450, family 2, subfamily d, polype…
4 Klk6 GSM2545336 287 kallikrein related-peptidase 6 [Source:MGI Sym…
5 Fcrls GSM2545336 85 Fc receptor-like S, scavenger receptor [Source…
6 Slc2a4 GSM2545336 782 solute carrier family 2 (facilitated glucose t…
7 Exd2 GSM2545336 1619 exonuclease 3'-5' domain containing 2 [Source:…
8 Gjc2 GSM2545336 288 gap junction protein, gamma 2 [Source:MGI Symb…
9 Plp1 GSM2545336 43217 proteolipid protein (myelin) 1 [Source:MGI Sym…
10 Gnb4 GSM2545336 1071 guanine nucleotide binding protein (G protein)…実生活では、遺伝子アノテーションのラベルが異なることがある。
annot2テーブルは、遺伝子名を含む 変数のラベルが異なる以外は、annot1と全く同じである。 この場合も、 [download annot2.csv](https://carpentries-incubator.github.io/bioc-intro/data/annot2.csv) 、自分でdata/`に移動するか、以下のRコードを使う。
この場合も、 download
annot2.csv
、自分でdata/\に移動するか、以下のRコードを使う。
R
download.file(url = "https://carpentries-incubator.github.io/bioc-intro/data/annot2.csv",
destfile = "data/annot2.csv")
annot2 <- read_csv(file = "data/annot2.csv")
annot2
出力
# A tibble: 10 × 2
external_gene_name description
<chr> <chr>
1 Cyp2d22 cytochrome P450, family 2, subfamily d, polypeptide 22 [S…
2 Klk6 kallikrein related-peptidase 6 [Source:MGI Symbol;Acc:MGI…
3 Fcrls Fc receptor-like S, scavenger receptor [Source:MGI Symbol…
4 Plp1 proteolipid protein (myelin) 1 [Source:MGI Symbol;Acc:MGI…
5 Exd2 exonuclease 3'-5' domain containing 2 [Source:MGI Symbol;…
6 Apod apolipoprotein D [Source:MGI Symbol;Acc:MGI:88056]
7 Gnb4 guanine nucleotide binding protein (G protein), beta 4 [S…
8 Slc2a4 solute carrier family 2 (facilitated glucose transporter)…
9 Asl argininosuccinate lyase [Source:MGI Symbol;Acc:MGI:88084]
10 Gjc2 gap junction protein, gamma 2 [Source:MGI Symbol;Acc:MGI:…どの変数名も一致しない場合、マッチングに使用する
変数を手動で設定することができる。
どの変数名も一致しない場合、マッチングに使用する
変数を手動で設定することができる。 これらの変数は、rna_mini
と annot2 テーブルを使用して以下に示すように、
by 引数を使用して設定することができる。
R
full_join(rna_mini, annot2, by = c("gene" = "external_gene_name"))
出力
# A tibble: 10 × 4
gene sample expression description
<chr> <chr> <dbl> <chr>
1 Asl GSM2545336 1170 argininosuccinate lyase [Source:MGI Symbol;Acc…
2 Apod GSM2545336 36194 apolipoprotein D [Source:MGI Symbol;Acc:MGI:88…
3 Cyp2d22 GSM2545336 4060 cytochrome P450, family 2, subfamily d, polype…
4 Klk6 GSM2545336 287 kallikrein related-peptidase 6 [Source:MGI Sym…
5 Fcrls GSM2545336 85 Fc receptor-like S, scavenger receptor [Source…
6 Slc2a4 GSM2545336 782 solute carrier family 2 (facilitated glucose t…
7 Exd2 GSM2545336 1619 exonuclease 3'-5' domain containing 2 [Source:…
8 Gjc2 GSM2545336 288 gap junction protein, gamma 2 [Source:MGI Symb…
9 Plp1 GSM2545336 43217 proteolipid protein (myelin) 1 [Source:MGI Sym…
10 Gnb4 GSM2545336 1071 guanine nucleotide binding protein (G protein)…上で見たように、最初のテーブルの変数名は、結合されたテーブルでも 。
チャレンジだ:
こちら
をクリックして annot3
テーブルをダウンロードし、そのテーブルをあなたの data/
リポジトリに置いてください。
full_join()関数を使用して、テーブルrna_miniとannot3`
を結合する。
、遺伝子_Klk6_、mt-Tf、mt-Rnr1、mt-Tv、mt-Rnr2、mt-Tl1_はどうなったのか?
full_join()関数を使用して、テーブルrna_miniとannot3`
を結合する。
、遺伝子_Klk6、mt-Tf、mt-Rnr1、mt-Tv、mt-Rnr2、_mt-Tl1_はどうなったのか?
R
annot3 <- read_csv("data/annot3.csv")
full_join(rna_mini, annot3)
出力
# A tibble: 15 × 4
gene sample expression gene_description
<chr> <chr> <dbl> <chr>
1 Asl GSM2545336 1170 argininosuccinate lyase [Source:MGI Symbol;Acc…
2 Apod GSM2545336 36194 apolipoprotein D [Source:MGI Symbol;Acc:MGI:88…
3 Cyp2d22 GSM2545336 4060 cytochrome P450, family 2, subfamily d, polype…
4 Klk6 GSM2545336 287 <NA>
5 Fcrls GSM2545336 85 Fc receptor-like S, scavenger receptor [Source…
6 Slc2a4 GSM2545336 782 solute carrier family 2 (facilitated glucose t…
7 Exd2 GSM2545336 1619 exonuclease 3'-5' domain containing 2 [Source:…
8 Gjc2 GSM2545336 288 gap junction protein, gamma 2 [Source:MGI Symb…
9 Plp1 GSM2545336 43217 proteolipid protein (myelin) 1 [Source:MGI Sym…
10 Gnb4 GSM2545336 1071 guanine nucleotide binding protein (G protein)…
11 mt-Tf <NA> NA mitochondrially encoded tRNA phenylalanine [So…
12 mt-Rnr1 <NA> NA mitochondrially encoded 12S rRNA [Source:MGI S…
13 mt-Tv <NA> NA mitochondrially encoded tRNA valine [Source:MG…
14 mt-Rnr2 <NA> NA mitochondrially encoded 16S rRNA [Source:MGI S…
15 mt-Tl1 <NA> NA mitochondrially encoded tRNA leucine 1 [Source…遺伝子_Klk6_はrna_miniにのみ存在し、遺伝子_mt-Tf_、mt-Rnr1、mt-Tv、
mt-Rnr2、_mt-Tl1_はannot3テーブルにのみ存在する。
表の 変数のそれぞれの値は、欠損として符号化されている。 表の
変数のそれぞれの値は、欠損として符号化されている。
データのエクスポート
dplyr\`を使って、 から情報を抽出したり、生データを要約したりする方法を学んだので、これらの新しいデータセットをエクスポートして、 を共同研究者と共有したり、アーカイブしたりしたいと思うかもしれない。
RにCSVファイルを読み込むために使用される read_csv()
関数と同様に、 、データフレームからCSVファイルを生成する
write_csv() 関数があります。
write_csv()を使う前に、生成されたデータセットを格納する新しいフォルダdata_outputを作業ディレクトリに作成する。 、生成されたデータセットを生データと同じディレクトリに書き込みたくない。 別々にするのは良い習慣だ。 dataフォルダーには、
、変更されていない生のデータだけを入れておく。
、削除したり変更したりしないように、そのままにしておく。
対照的に、このスクリプトはdata_output
ディレクトリの内容を生成するので、そこに含まれるファイルが削除されても、
再生成することができる。
、生成されたデータセットを生データと同じディレクトリに書き込みたくない。
別々にするのは良い習慣だ。
dataフォルダーには、 、変更されていない生のデータだけを入れておく。 、削除したり変更したりしないように、そのままにしておく。 対照的に、このスクリプトはdata_output`
ディレクトリの内容を生成するので、そこに含まれるファイルが削除されても、
再生成することができる。
write_csv()\`を使用して、以前に作成したrna_wideテーブルを保存しよう。
R
write_csv(rna_wide, file = "data_output/rna_wide.csv")
Content from データの可視化
最終更新日:2024-09-08 | ページの編集
所要時間: 120分
概要
質問
- Rによる可視化
目的
- ggplotを使って散布図、箱ひげ図、折れ線グラフなどを作成する。
- ユニバーサルプロット設定を行う。
- ファセットとは何かを説明し、ggplotでファセットを適用する。
- 既存のggplotプロットの美学(軸ラベルや色を含む)を修正する。
- データフレーム内のデータから複雑でカスタマイズされたプロットを作成。
このエピソードは、Data Carpentriesの_Data Analysis and Visualisation in R for Ecologists_レッスンに基づいています。
データの可視化
必要なパッケージをロードすることから始める。
**ggplot2**は、 **tidyverse**パッケージに含まれています。
R
library("tidyverse")
ワークスペースにまだない場合は、前回の レッスンで保存したデータをロードします。
R
rna <- read.csv("data/rnaseq.csv")
Data Visualization Cheat Sheet](https://raw.githubusercontent.com/rstudio/cheatsheets/main/data-visualization.pdf)
は ggplot2 の基本からより高度な機能までをカバーし、
パッケージで利用可能な多くのデータ表現
の概要を理解するためのリマインダとしてだけでなく、手助けになるでしょう。
Thomas Lin Pedersen氏による以下のビデオ チュートリアル(パート1と 2)
も非常に参考になる。
ggplot2`によるプロット
ggplot2は、データフレーム内のデータから複雑な プロットを簡単に作成できるプロットパッケージである。 どの変数をプロットするか、どのように表示するか、 、一般的なビジュアル・プロパティを指定するための、よりプログラム的な 。ggplot2`を支える理論的基盤は、Grammar
of Graphics (@Wilkinson:2005)である。
この
アプローチを使用すると、基礎となるデータが変更された場合、または棒グラフから散布図に変更することを決めた場合に
、最小限の変更で済む。 これは、
、最小限の調整で出版品質のプロットを作成するのに役立ちます
、微調整。
ggplot2に関する本(@ggplot2book)があり、 。 第3版は現在準備中で、 [オンラインで自由に利用できる](https://ggplot2-book.org/)。 のggplot2`
ウェブページ(https://ggplot2.tidyverse.org)に十分なドキュメントがあります。
ggplot2の関数は、'long'形式のデータ、つまり、 すべての次元を表す列と、すべてのオブザベーションを表す行を持つ。 よく構造化されたデータ 、ggplot2`で図を作成する時間を大幅に節約できる。
ggplotグラフィックスは、新しい要素を追加することによって段階的に構築される。 この方法で レイヤーを追加すると、プロットの広範な柔軟性と カスタマイズが可能になる。
Grammar of Graphicsの背後にある考え方は、同じ3つのコンポーネントからすべての グラフを構築できるということである:(1)データセット、(2)座標系、 、(3)ジオム、つまりデータ点を表す視覚的マーク 1 である。
ggplotを構築するために、 、さまざまなタイプのプロットに使用できる以下の基本テンプレートを使用する:
ggplot(data =<DATA>, mapping = aes(<MAPPINGS>)) +<GEOM_FUNCTION>()- ggplot()
関数を使用し、data`引数を使用して特定のdata frameにプロットをバインドする。
R
ggplot(data = rna)
- (
aes)関数を使用して)マッピングを定義する。 、プロットする変数を選択し、 、x/yの位置や、 、サイズ、形、色などの特徴として、グラフでどのように表示するかを指定する。
R
ggplot(data = rna, mapping = aes(x = expression))
-
追加 ‘geoms’ - ジオメトリ、つまりプロット内の データのグラフ表現(点、線、棒)。 ggplot2`は多くの 、様々なジオムを提供している:
* 散布図やドットプロットなどには `geom_point()` を使用する。 * * `geom_boxplot()` ボックスプロット! * トレンドライン、時系列など。
プロットにgeom(etry)を追加するには + 演算子を使います。
geom_histogram() をまず使ってみよう:
R
ggplot(data = rna, mapping = aes(x = expression)) +
geom_histogram()
出力
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
ggplot2パッケージの+は特に便利で、 、既存のggplot`オブジェクトを修正することができる。
つまり、 、プロット・テンプレートを簡単に設定し、
、さまざまなタイプのプロットを便利に調べることができる:
R
# Assign plot to a variable
rna_plot <- ggplot(data = rna,
mapping = aes(x = expression))
# Draw the plot
rna_plot + geom_histogram()
R
# change bins
ggplot(rna, aes(x = expression)) +
geom_histogram(bins = 15)
R
# change binwidth
ggplot(rna, aes(x = expression)) +
geom_histogram(binwidth = 2000)
データが右に偏っていることがわかる。 より対称的な分布にするために、
log2変換を適用することができる。 ここでは、
、0に等しい式の値に対して返される-Inf値
を避けるために、小さな定数値(+1)を追加していることに注意してください。
R
rna<- rna %>%
mutate(expression_log = log2(expression + 1))
ここで対数変換した式のヒストグラムを描いてみると、 の分布は確かに正規分布に近くなっている。
R
ggplot(rna, aes(x = expression_log)) + geom_histogram()
出力
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
これからは対数変換した発現値を扱うことにする。
R
ggplot(data = rna,mapping = aes(x = expression))+
geom_histogram() +
scale_x_log10()
警告
Warning in scale_x_log10(): log-10 transformation introduced infinite values.出力
`stat_bin()` using `bins = 30`. Pick better value with `binwidth`.警告
Warning: Removed 507 rows containing non-finite outside the scale range
(`stat_bin()`).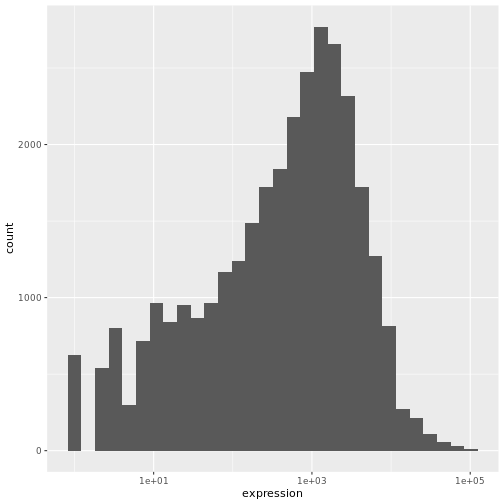
注*。
- ggplot()
関数で設定したものはすべて、あなたが追加したgeom レイヤーで見ることができます(つまり、これらはグローバルなプロット設定です)。 この には、aes()`で設定したx軸とy軸のマッピングが含まれる。 - また、
ggplot()関数でグローバルに定義された マッピングとは別に、与えられたジオムに対するマッピングを指定することもできます。 - 新しいレイヤーを追加するために使われる
+記号は、_前の_レイヤーを含む 行の最後に置かなければなりません。 その代わりに、+記号が 新しいレイヤーを含む行の先頭に追加された場合、ggplot2は新しいレイヤーを追加せず、エラー メッセージを返す。
R
# これはレイヤーを追加するための正しい構文です
rna_plot +
geom_histogram()
# これは新しいレイヤーを追加せず、エラーメッセージを返します
rna_plot
+ geom_histogram()
反復的にプロットを構築する
ここでは、2つの連続変数と
geom_point()関数を使って散布図を描きます。
このグラフは、時間8と時間0、時間4と時間0を比較した発現のlog2倍変化 。
この目的のために、まず対数変換した
発現値の平均値を遺伝子ごと、時間ごとに計算する必要がある。次に、
、時間8と時間0の間の平均対数発現と、
、時間4と時間0の間の平均対数発現を差し引くことにより、対数倍変化を計算する。
ここでは、後で遺伝子を表現するために使用する遺伝子
バイオタイプも含めていることに注意されたい。 フォールドの変化を
rna_fc. という新しいデータフレームに保存する。
R
rna_fc <- rna %>% select(gene, time,
gene_biotype, expression_log) %>%
group_by(gene, time, gene_biotype) %>%
summarize(mean_exp = mean(expression_log)) %>%
pivot_wider(names_from = time,
values_from = mean_exp) %>%
mutate(time_8_vs_0 = `8` - `0`, time_4_vs_0 = `4` - `0`)
出力
`summarise()` has grouped output by 'gene', 'time'. You can override using the
`.groups` argument.新しく作成されたデータセット rna_fc を使って ggplot
を作成することができる。
ggplot2`でプロットを作成するのは、通常、反復的なプロセスである。
まず、使用するデータセットを定義し、軸を配置し、 ジオムを選択する:
R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0)) +
geom_point()
次に、このプロットからより多くの情報を抽出するために、プロットを修正し始める。
例えば、オーバープロットを避けるために透明度(α)を加えることができる:
R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0)) +
geom_point(alpha = 0.3)
また、すべてのポイントに色を付けることもできる:
R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0)) +
geom_point(alpha = 0.3, color = "blue")
あるいは、プロット中の各遺伝子を異なる色にするために、
、引数colorの入力としてベクトルを使うこともできる。
ggplot2は、ベクトルの異なる値に対応する異なる 。 以下は、gene_biotype`を使った例である:
R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0)) +
geom_point(alpha = 0.3, aes(color = gene_biotype))

また、
ggplot()関数で提供されるマッピングの中で直接色を指定することもできる。
これはどのジオムレイヤーでも見ることができ、 のマッピングは
aes() で設定したx軸とy軸によって決定される。
R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0,
color = gene_biotype)) +
geom_point(alpha = 0.3)

最後に、geom_abline() :
R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0,
color = gene_biotype)) +
geom_point(alpha = 0.3) +
geom_abline(intercept = 0)
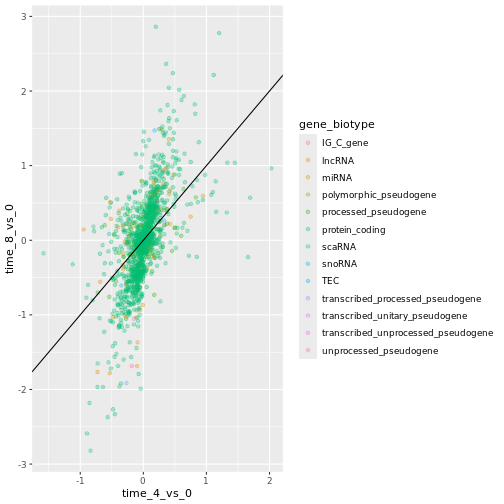
ジオムレイヤーを geom_point から
geom_jitter に変更しても、色は gene_biotype
によって決定されることに注意してください。
R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0,
color = gene_biotype)) +
geom_jitter(alpha = 0.3) +
geom_abline(intercept = 0)

エラー
Error in library("hexbin"): there is no package called 'hexbin'チャレンジ
散布図は、小規模なデータセットの探索に役立つツールである。 rna_fc` データセットのような多数のオブザベーションを持つ データセットの場合、点のオーバープロットは散布図の制限となりうる。 このような設定を扱うための1つの戦略は、 の観測値を六角形にビニングすることである。 プロット空間は六角形にテッセレーションされている。 それぞれの 六角形は、 その境界内に入るオブザベーションの数に基づいて色が割り当てられる。
ggplot2
で六角ビニングを使用するには、まずRパッケージhexbin`をCRANからインストールしてロードする。そして、
geom_hex()関数を使ってhexbin図を作成する。散布図と比較して、六角形のビン プロットの相対的な長所と短所は何か? 上記の散布図( )を調べ、作成した六角形のビンプロットと比較する。
R
install.packages("hexbin")
R
library("hexbin")
エラー
Error in library("hexbin"): there is no package called 'hexbin'R
ggplot(data = rna_fc, mapping = aes(x = time_4_vs_0, y = time_8_vs_0)) +
geom_hex() +
geom_abline(intercept = 0)
警告
Warning: Computation failed in `stat_binhex()`.
Caused by error in `compute_group()`:
! The package "hexbin" is required for `stat_bin_hex()`.
R
ggplot(data = rna, mapping = aes(y = expression_log, x = sample)) +
geom_point(aes(color = time))
ボックスプロット
ボックスプロットを使って、各サンプル内の遺伝子発現の分布( )を可視化することができる:
R
ggplot(data = rna,
mapping = aes(y = expression_log, x = sample)) +
geom_boxplot()

boxplotにポイントを追加することで、 の測定数とその分布をよりよく知ることができる:
R
ggplot(data = rna,
mapping = aes(y = expression_log, x = sample)) +
geom_jitter(alpha = 0.2, color = "tomato") +
geom_boxplot(alpha = 0)
この2つのジオムの順番を入れ替えるべきだ:
R
ggplot(data = rna,
mapping = aes(y = expression_log, x = sample))+
geom_boxplot(alpha = 0) +
geom_jitter(alpha = 0.2, color = "tomato")
X軸の値がまだ正しく 読めないことにお気づきかもしれない。 ラベルの向きを変え、 縦と横に重ならないように調整しよう。 90度の角度を使ってもいいし、 斜め向きのラベルに適切な角度を見つけるために実験してもいい:
R
ggplot(data = rna,
mapping = aes(y = expression_log, x = sample))+
geom_jitter(alpha = 0.2, color = "tomato") +
geom_boxplot(alpha = 0) +
theme(axis.text.x = element_text(angle = 90, hjust = 0.5, vjust = 0.5))
R
# time as integer
ggplot(data = rna,
mapping = aes(y = expression_log,
x = sample))+
geom_jitter(alpha = 0.2, aes(color = time))+
geom_boxplot(alpha = 0) +
theme(axis.text.x = element_text(angle = 90, hjust = 0.5, vjust = 0.5))
R
# time as factor
ggplot(data = rna,
mapping = aes(y = expression_log,
x = sample))+
geom_jitter(alpha = 0.2, aes(color = as.factor(time)))+
geom_boxplot(alpha = 0) +
theme(axis.text.x = element_text(angle = 90, hjust = 0.5, vjust = 0.5))
R
ggplot(data = rna,
mapping = aes(y = expression_log, x = sample))+
geom_violin(aes(fill = as.factor(time)))+
theme(axis.text.x = element_text(angle = 90, hjust = 0.5, vjust = 0.5))
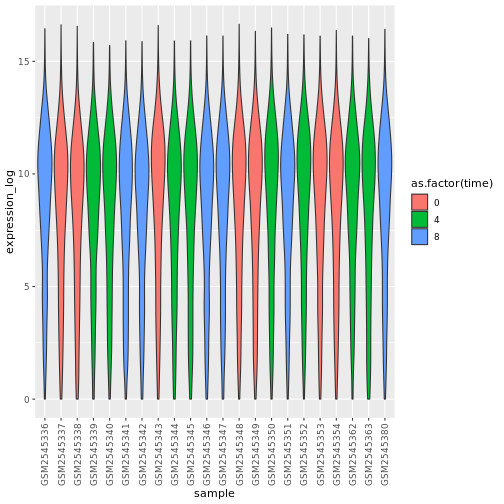
R
ggplot(data = rna,
mapping = aes(y = expression_log, x = sample))+
geom_violin(aes(fill = sex))+
theme(axis.text.x = element_text(angle = 90, hjust = 0.5, vjust = 0.5))
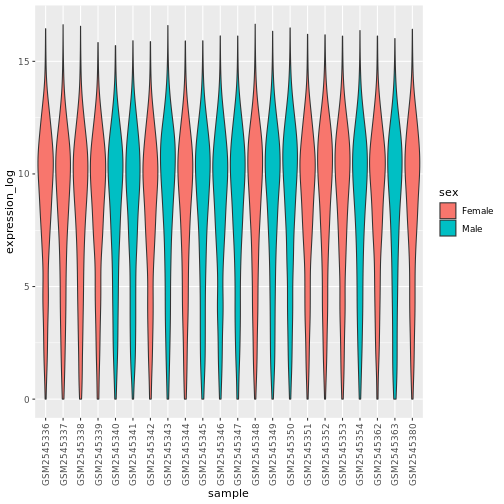
線プロット
時刻8と 時刻0を比較し、log
fold変化が最も大きかった10の遺伝子について、感染期間ごとの平均発現量を計算してみよう。
まず、遺伝子を選択し、選択した10遺伝子を含むsub_rnaと呼ばれるrna
のサブセットを作成する必要がある。次に、
データをグループ化し、各グループ内の平均遺伝子発現を計算する必要がある:
R
rna_fc<- rna_fc %>% arrange(desc(time_8_vs_0))
genes_selected <- rna_fc$gene[1:10]
sub_rna<- rna %>%
filter(gene %in% genes_selected)
mean_exp_by_time<- sub_rna %>%
group_by(gene,time) %>%
summarize(mean_exp = mean(expression_log))
出力
`summarise()` has grouped output by 'gene'. You can override using the
`.groups` argument.R
mean_exp_by_time
出力
# A tibble: 30 × 3
# Groups: gene [10]
gene time mean_exp
<chr> <int> <dbl>
1 Acr 0 5.07
2 Acr 4 5.54
3 Acr 8 7.31
4 Aipl1 0 3.70
5 Aipl1 4 3.89
6 Aipl1 8 6.56
7 Bst1 0 3.20
8 Bst1 4 3.77
9 Bst1 8 5.22
10 Chil3 0 4.00
# ℹ 20 more rowsX軸に感染期間( )、Y軸に平均発現をとって折れ線グラフを作成することができる:
R
ggplot(data = mean_exp_by_time, mapping = aes(x = time, y = mean_exp))+
geom_line()
残念なことに、これはうまくいかない。というのも、
の全遺伝子のデータをまとめてプロットしたからである。
各遺伝子に対して線を引くようにggplotに指示する必要がある。
、group = geneを含むようにesthetic関数を修正する:
R
ggplot(data = mean_exp_by_time,
mapping = aes(x = time, y = mean_exp, group = gene))+
geom_line()
色をつければ、プロットの中で遺伝子を区別できるようになる(
colorを使えば、自動的にデータをグループ化することもできる):
R
ggplot(data = mean_exp_by_time,
mapping = aes(x = time, y = mean_exp, color = gene))+
geom_line()
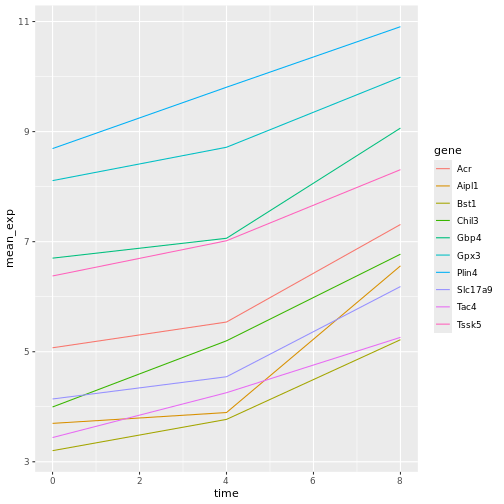
ファセット
ggplot2`には_faceting_と呼ばれる特別なテクニックがあり、 、データセットに含まれる 、1つのプロットを複数の(サブ)プロットに分割することができる。 こ れ ら の異な る サブプ ロ ッ ト は、 同 じ プ ロ パテ ィ を継承 し ます (軸の限界、目盛り、 …)。 直接比較しやすくするためだ。 、これを用いて各遺伝子について時間軸に沿った折れ線グラフを作成する:
R
ggplot(data = mean_exp_by_time,
mapping = aes(x = time, y = mean_exp))+ geom_line() +
facet_wrap(~ gene)
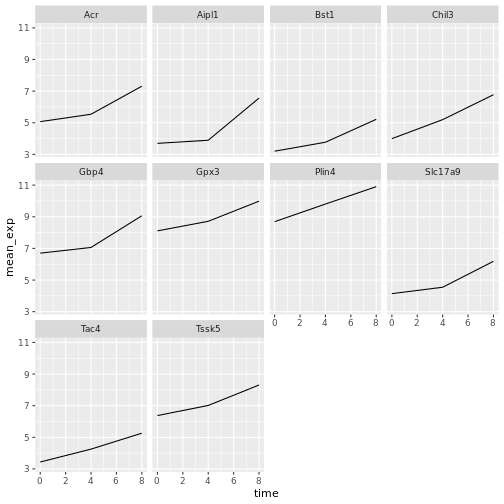
ここでは、X軸とY軸はすべてのサブプロットで同じスケールになっている。、Y軸のスケールを自由に設定できるようにscalesを修正することで、このデフォルトの動作を変更することができる:
R
ggplot(data = mean_exp_by_time,
mapping = aes(x = time, y = mean_exp))+
geom_line() +
facet_wrap(~ gene, scales = "free_y")
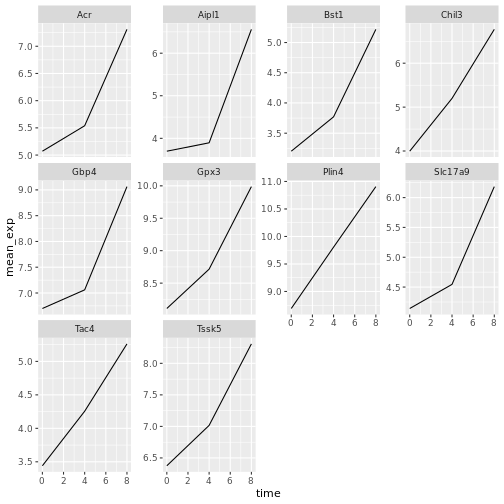
ここで、各プロットの線をマウスの性別で分けたい。 そのためには、 、遺伝子、時間、性別でグループ化したデータフレームの平均発現を計算する必要がある:
R
mean_exp_by_time_sex<- sub_rna %>%
group_by(gene, time, sex) %>%
summarize(mean_exp = mean(expression_log))
出力
`summarise()` has grouped output by 'gene', 'time'. You can override using the
`.groups` argument.R
mean_exp_by_time_sex
出力
# A tibble: 60 × 4
# Groups: gene, time [30]
gene time sex mean_exp
<chr> <int> <chr> <dbl>
1 Acr 0 Female 5.13
2 Acr 0 Male 5.00
3 Acr 4 Female 5.93
4 Acr 4 Male 5.15
5 Acr 8 Female 7.27
6 Acr 8 Male 7.36
7 Aipl1 0 Female 3.67
8 Aipl1 0 Male 3.73
9 Aipl1 4 Female 4.07
10 Aipl1 4 Male 3.72
# ℹ 50 more rowscolor
を使ってさらに分割することで、ファセット化されたプロットを作ることができる(1つのプロット内で):
R
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex))+
geom_line() +
facet_wrap(~ gene, scales = "free_y")
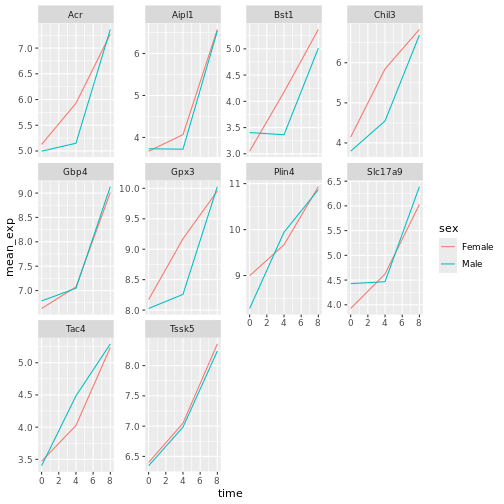
通常、背景が白いプロットは印刷したときに読みやすくなる。 、関数
theme_bw() を使って背景を白に設定することができる。
さらに、グリッドを削除することもできる:
R
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex))+
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank())
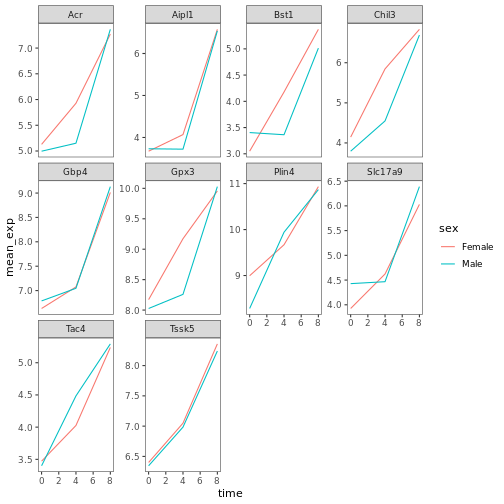
R
mean_exp_by_chromosome<- rna %>%
group_by(chromosome_name, time) %>%
summary(mean_exp = mean(expression_log))
ggplot(data = mean_exp_by_chromosome, mapping = aes(x = time,
y = mean_exp))+
geom_line() +
facet_wrap(~ chromosome_name, scales = "free_y")
エラー
Error in `fortify()`:
! `data` must be a <data.frame>, or an object coercible by `fortify()`,
or a valid <data.frame>-like object coercible by `as.data.frame()`.
Caused by error in `.postvalidate_data_frame_like_object()`:
! `as.data.frame(data)` must preserve dimensions.facet_wrapジオメトリは、1ページにきれいに収まるように、プロットを任意の数の 次元に抽出する。 一方、facet_gridジオメトリでは、 数式表記(rows
~ columns;.`
は、1つの行または列のみを示すプレースホルダとして使用できます)によって、プロットの配置方法を明示的に指定することができます。
先ほどのプロットを修正し、男性と女性の平均遺伝子発現 が経時的にどのように変化したかを比較してみよう:
R
# 1列、行ごとのファセット
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = gene))+
geom_line() +
facet_grid(sex ~ .)
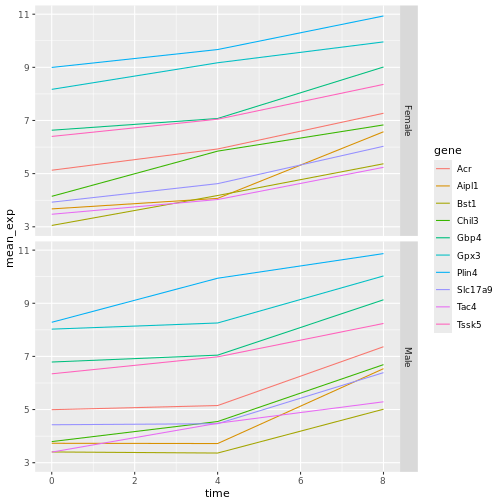
R
# 1行、列ごとのファセット
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = gene))+
geom_line() +
facet_grid(. ~ sex)
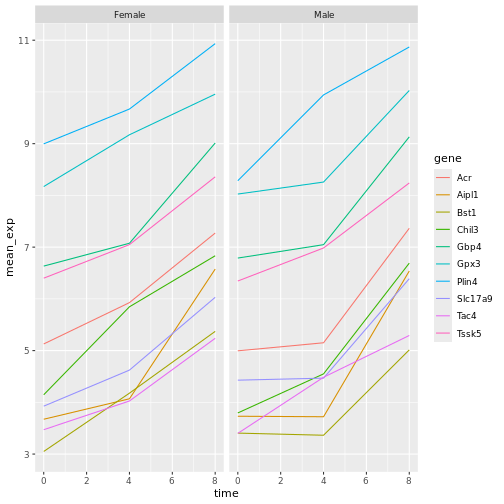
ggplot2`テーマ
ggplot2 には、プロットの背景を白に変更する
theme_bw() に加えて、
手早く視覚化の見た目を変更するのに便利なテーマがいくつか用意されている。
テーマの全リストはhttps://ggplot2.tidyverse.org/reference/ggtheme.htmlで入手できる。
theme_minimal()やtheme_light()は人気があり、theme_void()`
は新しい手作りのテーマを作る出発点として役に立つ。
ggthemes](https://jrnold.github.io/ggthemes/reference/index.html)
パッケージは様々なオプション(Excel 2003 テーマを含む)を提供します。
ggplot2 extensions website](https://exts.ggplot2.tidyverse.org/) は、追加の
テーマを含む ggplot2 の機能を拡張する
パッケージのリストを提供しています。
カスタマイズ
時間別、遺伝子別の平均発現のファセット・プロットに戻ろう。 性別に色分けされている。
ggplot2`のカンニング シート](https://raw.githubusercontent.com/rstudio/cheatsheets/main/data-visualization.pdf), を見て、プロットを改善する方法を考えてみてください。
ここで、軸の名前を ‘time’ や ‘mean_exp’ よりも情報量の多いものに変更し、図にタイトルを追加する:
R
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex))+
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank()) +
labs(title = "感染期間別の平均遺伝子発現",
x = "感染期間(日単位)",
y = "平均遺伝子発現")
軸にはより情報量の多い名前が付けられているが、フォントサイズを大きくすることで読みやすさは :
R
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex))+
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank()) +
labs(title = "感染期間別の平均遺伝子発現",
x = "感染期間(日単位)",
y = "平均遺伝子発現") +
theme(text = element_text(size = 16))
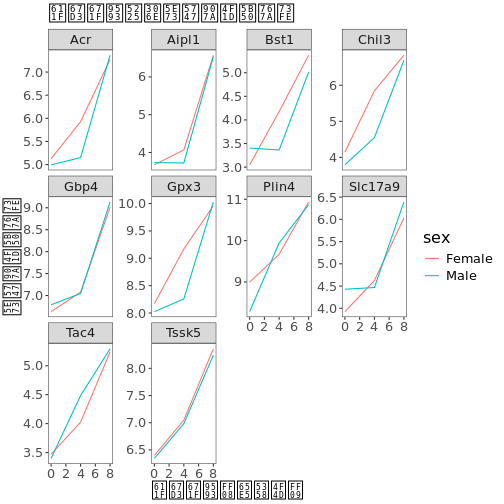
プロットのフォントを変更することも可能です。 Windowsをお使いの場合は、 をインストールする必要があるかもしれません。
さらに、X軸とY軸のテキストの色、
グリッドの色などをカスタマイズできる。 例えば、
legend.positionを"top"に設定することで、凡例を一番上に移動させることもできる。
R
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex))+
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank()) +
labs(title = "感染期間別の平均遺伝子発現",
x = "感染期間(日単位)",
y = "平均遺伝子発現") +
theme(text = element_text(size = 16),
axis.text.x = element_text(color = "royalblue4", size = 12),
axis.text.y = element_text(color = "royalblue4", size = 12),
panel.grid = element_line(color="lightsteelblue1"),
legend.position = "top")
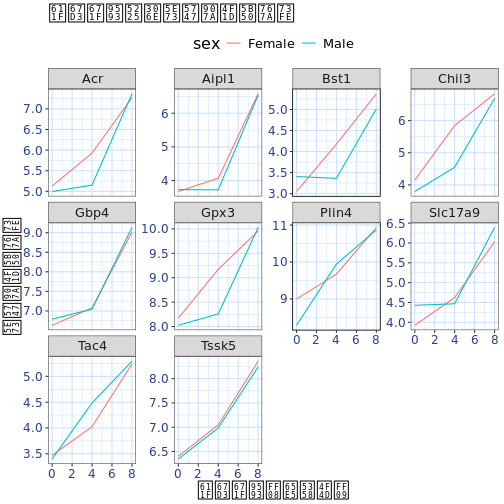
作成した変更がデフォルトのテーマよりも気に入った場合は、 、オブジェクトとして保存して、作成した他の プロットに簡単に適用することができます。 以下は、 以前に作成したヒストグラムを使った例です。
R
blue_theme <- theme(axis.text.x = element_text(colour = "royalblue4",
size = 12),
axis.text.y = element_text(colour = "royalblue4",
size = 12),
text = element_text(size = 16),
panel.grid = element_line(colour="lightsteelblue1"))
ggplot(rna, aes(x = expression_log)) +
geom_histogram(bins = 20) +
blue_theme
チャレンジ
これらの情報を手に入れたら、 、この練習で作成したプロットのいずれかを改良するか、 、あなた自身の美しいグラフを作成してください。 RStudio ggplot2 を参考にしてください。 いくつかアイデアを挙げてみよう:
- 線の太さを変えてみてください。
- 伝説の名前を変える方法はありますか?
そのラベルについてはどうだろう? (ヒント:
scale_で始まる ggplot 関数を探す) - 別のカラーパレットを使用するか、線の カラーを手動で指定してみてください( http://www.cookbook-r.com/Graphs/Colors_(ggplot2)/ 参照)。
例えば、このプロットに基づくと
R
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex))+
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank())
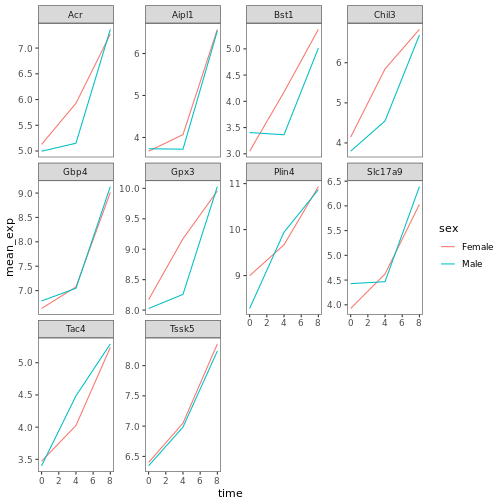
以下のようなカスタマイズが可能です:
R
# change the thickness of the lines
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex)) +
geom_line(size=1.5) +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank())
警告
Warning: Using `size` aesthetic for lines was deprecated in ggplot2 3.4.0.
ℹ Please use `linewidth` instead.
This warning is displayed once every 8 hours.
Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
generated.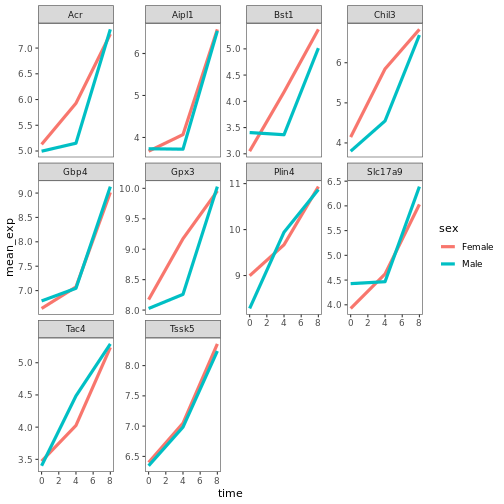
R
# change the name of the legend and the labels
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex)) +
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank()) +
scale_color_discrete(name = "Gender", labels = c("F", "M"))
R
# using a different color palette
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex)) +
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank()) +
scale_color_brewer(name = "Gender", labels = c("F", "M"), palette = "Dark2")
R
# manually specifying the colors
ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex)) +
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
theme_bw() +
theme(panel.grid = element_blank()) +
scale_color_manual(name = "Gender", labels = c("F", "M"),
values = c("royalblue", "deeppink"))
プロットの構成
ファセッティングは、1つのプロットを複数のサブプロットに分割するのに最適なツールです。 しかし、 複数の独立したプロット、すなわち異なる 変数、あるいは異なるデータフレームに基づくプロットを含む1つの図を作成したい場合があります。
、まず2つのプロットを作成する:
最初のグラフは、染色体ごとにユニークな遺伝子の数を数えている。
、まずchromosome_nameのレベルを並べ替え、
、染色体ごとにユニークな遺伝子をフィルタリングする必要がある。
また、読みやすくするために、Y軸のスケールを
log10スケールに変更した。
R
rna$chromosome_name <- factor(rna$chromosome_name,
levels = c(1:19,"X","Y"))
count_gene_chromosome <- rna %>% select(chromosome_name, gene) %>%
distinct() %>% ggplot() +
geom_bar(aes(x = chromosome_name), fill = "seagreen",
position = "dodge", stat = "count") +
labs(y = "log10(n genes)", x = "chromosome") +
scale_y_log10()
count_gene_chromosome
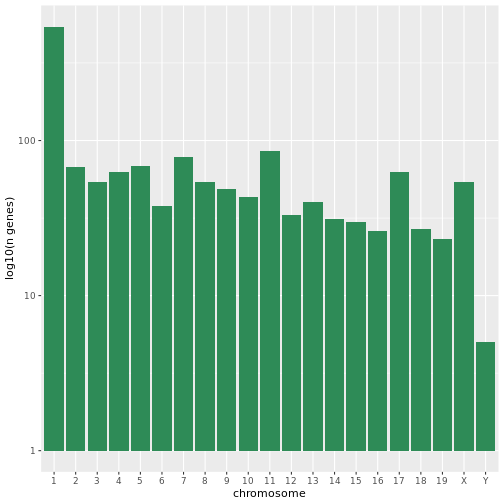
以下では、 legend.position を "none"
に設定することで、凡例を完全に削除することもできる。
R
exp_boxplot_sex <- ggplot(rna, aes(y=expression_log, x = as.factor(time),
color=sex)) +
geom_boxplot(alpha = 0) +
labs(y = "Mean gene exp",
x = "time") + theme(legend.position = "none")
exp_boxplot_sex
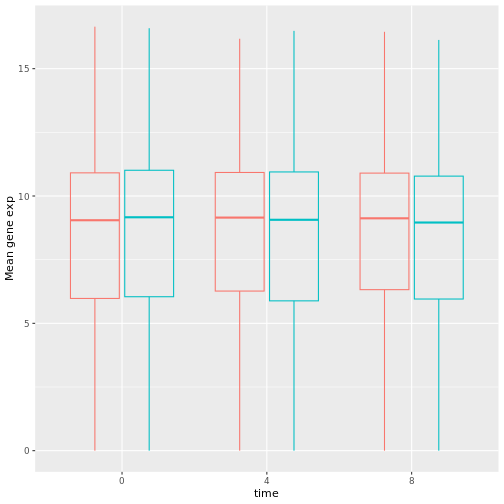
patchwork**](https://github.com/thomasp85/patchwork)パッケージ
は、+
を使って図形を組み合わせるエレガントなアプローチを提供し、
図形を(通常は横に並べて)並べます。 より具体的には、|
は明示的に横に並べ、/は 上に重ねる。
R
install.packages("patchwork")
R
library("patchwork")
エラー
Error in library("patchwork"): there is no package called 'patchwork'R
count_gene_chromosome + exp_boxplot_sex
エラー
Error in `ggplot_add()`:
! Can't add `exp_boxplot_sex` to a <ggplot> object.R
## or count_gene_chromosome | exp_boxplot_sex
R
count_gene_chromosome / exp_boxplot_sex
エラー
Error in count_gene_chromosome/exp_boxplot_sex: non-numeric argument to binary operatorplot_layout
と組み合わせることで、最終的なコンポジションのレイアウトをさらにコントロールし、より複雑なレイアウトを作成することができる:
R
count_gene_chromosome + exp_boxplot_sex + plot_layout(ncol = 1)
エラー
Error in `ggplot_add()`:
! Can't add `exp_boxplot_sex` to a <ggplot> object.R
count_gene_chromosome +
(count_gene_chromosome + exp_boxplot_sex) +
exp_boxplot_sex +
plot_layout(ncol = 1)
エラー
Error in `ggplot_add()`:
! Can't add `exp_boxplot_sex` to a <ggplot> object.最後のプロットは | と /
のコンポーザーを使っても作成できる:
R
count_gene_chromosome /
(count_gene_chromosome | exp_boxplot_sex) /
exp_boxplot_sex
エラー
Error in count_gene_chromosome | exp_boxplot_sex: operations are possible only for numeric, logical or complex typesパッチワークの詳細については、 ウェブページ、またはこちらの ビデオをご覧ください。
もう一つのオプションは
**gridExtra** パッケージで、 の別々の ggplot をgrid.arrange()`
を使って一つの図にまとめることができます:
R
install.packages("gridExtra")
R
library("gridExtra")
エラー
Error in library("gridExtra"): there is no package called 'gridExtra'R
grid.arrange(count_gene_chromosome, exp_boxplot_sex, ncol = 2)
エラー
Error in grid.arrange(count_gene_chromosome, exp_boxplot_sex, ncol = 2): could not find function "grid.arrange"引数 ncol と nrow は単純な
の配置を作るのに使われるが、それに加えて、より複雑な のレイアウトを作る
ためのツールもある。
プロットのエクスポート
プロットを作成したら、お好きな 形式でファイルに保存できます。 RStudioのPlotペインのExportタブでは、 のプロットが低解像度で保存されます。これは多くのジャーナルでは受け入れられませんし、 ではポスター用にうまく拡大縮小できません。
代わりに ggsave() 関数を使います。この関数を使うと、
の適切な引数(width, height,
dpi)を調整することで、プロットの
次元と解像度を簡単に変更することができます。
作業ディレクトリに fig_output/
フォルダがあることを確認してください。
R
my_plot <- ggplot(data = mean_exp_by_time_sex,
mapping = aes(x = time, y = mean_exp, color = sex)) +
geom_line() +
facet_wrap(~ gene, scales = "free_y") +
labs(title = "Mean gene expression by duration of the infection",
x = "Duration of the infection (in days)",
y = "Mean gene expression") +
guides(color=guide_legend(title="Gender")) +
theme_bw() +
theme(axis.text.x = element_text(colour = "royalblue4", size = 12),
axis.text.y = element_text(colour = "royalblue4", size = 12),
text = element_text(size = 16),
panel.grid = element_line(colour="lightsteelblue1"),
legend.position = "top")
ggsave("fig_output/mean_exp_by_time_sex.png", my_plot, width = 15,
height = 10)
# This also works for grid.arrange() plots
combo_plot <- grid.arrange(count_gene_chromosome, exp_boxplot_sex,
ncol = 2, widths = c(4, 6))
ggsave("fig_output/combo_plot_chromosome_sex.png", combo_plot,
width = 10, dpi = 300)
注意: パラメータ width と height
は、保存されたプロットのフォントサイズ も決定します。
その他のビジュアライゼーション用パッケージ
ggplot2`は非常に強力なパッケージで、我々の_tidy data_と_tidy tools_パイプラインにうまくフィットする。 Rには他にも無視できない可視化パッケージ がある。
ベースグラフィック
Rに付属するデフォルトのグラフィックス・システムは、しばしば_ベースR
グラフィックス_と呼ばれ、シンプルで高速である。
これは_画家またはキャンバスの
モデル_に基づいており、異なる出力がそれぞれ 互いに直接重ね合わされる(図@ref(fig:paintermodel)を参照)。
これはggplot2(および後述するlattice)との基本的な
の違いである。
は専用のオブジェクトを返し、それは画面上またはファイル上にレンダリングされ、
は更新することさえできる。
R
par(mfrow = c(1, 3))
plot(1:20, main = "First layer, produced with plot(1:20)")
plot(1:20, main = "A horizontal red line, added with abline(h = 10)")
abline(h = 10, col = "red")
plot(1:20, main = "A rectangle, added with rect(5, 5, 15, 15)")
abline(h = 10, col = "red")
rect(5, 5, 15, 15, lwd = 3)
もうひとつの主な違いは、ベース・グラフィックスのプロット関数は、入力タイプに基づいて、
、_正しい_ことをしようとする。 これは、
データフレームしか入力として受け付けないggplot2で、プロットをビットごとに構築する必要があるのとは、
異なる。
R
par(mfrow = c(2, 2))
boxplot(rnorm(100),
main = "rnorm(100) の Boxplot")
boxplot(matrix(rnorm(100), ncol = 10),
main = "matrix(rnorm(100), ncol = 10) の Boxplot")
hist(rnorm(100))
hist(matrix(rnorm(100), ncol = 10))
、plot、hist、
boxplot、…のような1行のコードと1つの関数で非常に素早く作成できる。
しかし、デフォルトは必ずしも最も魅力的なものではありません。
、図形のチューニングは、特に複雑になると(例えばファセットを生成するために
)、時間がかかり面倒になります。
格子パッケージ
lattice** パッケージはggplot2と似ているが、 データフレームを入力として使い、グラフィカルオブジェクトを返し、ファセットをサポートする。 しかし、lattice`はグラフィックの文法に基づいておらず、
より複雑なインターフェイスを持っている。
lattice`パッケージの良いリファレンスは@latticebookだ。
Content from 次のステップ
最終更新日:2024-09-08 | ページの編集
所要時間: 90分
概要
質問
-
SummarizedExperimentとは何でしょうか? - Bioconductor と何でしょうか?
目的
- Bioconductorプロジェクトを紹介してみましょう。
- データコンテナの概念を紹介してみましょう。
- オミックス解析で多用される
SummarizedExperimentの概要を説明する。
次のステップ
バイオインフォマティクスのデータはしばしば複雑です。 これに対処するため、 開発者は、扱う必要のあるデータのプロパティに マッチする、特別なデータコンテナ(クラスと呼ばれる)を定義する。
この側面は、パッケージ間で同じコア・データ・インフラを使用するバイオコンダクター[^バイオコンダクター]プロジェクト 。 この 、Bioconductorの成功に貢献したことは間違いない。 Bioconductor パッケージ 開発者は、 プロジェクト全体に一貫性、相互運用性、安定性を提供するために、既存のインフラストラクチャを利用することをお勧めします 。
このようなオミックス・データ・コンテナを説明するために、
SummarizedExperimentクラスを紹介する。
実験概要
下図は、SummarizedExperimentクラスの構造を表しています。
SummarizedExperimentクラスのオブジェクトには、:
**定量的オミックスデータ (発現データ)を含む1つ(または複数)のアッセイ **、マトリックス状のオブジェクトとして格納されている。 特徴(遺伝子、 転写物、タンパク質、…) は行に沿って定義され、 は列に沿って定義される。
データフレームとして格納された、サンプルの共変量を含む sample metadata スロット。 この表の行はサンプルを表す(行は発現データの 列と正確に一致する)。
データフレームとして格納される、特徴共変量を含む 特徴メタデータ スロット。 このデータフレームの行は、 式データの行と完全に一致する。
SummarizedExperiment`の調整された性質は、データ操作中に 、異なるスロットの次元が 、常に一致することを保証する(すなわち、発現データの列と サンプルメタデータの行、および発現データと 特徴メタデータの行)。 例えば、 、アッセイから1つのサンプルを除外しなければならない場合、同じ操作でサンプルメタデータから 、自動的に除外される。
メタデータ・スロットは、他の構造に影響を与えることなく、 (カラム)の共変数を追加で増やすことができる。
SummarizedExperimentの作成
SummarizedExperiment`を作成するために、 の各コンポーネント、すなわちカウントマトリックス、サンプル、遺伝子 のメタデータをcsvファイルから作成する。 これらは通常、RNA-Seqデータが (生データが処理された後)提供される方法である。
-
An expression matrix: カウント行列をロードし、
最初の列が行/遺伝子名を含むことを指定し、
data.frameをmatrixに変換する。 ダウンロードは こちら。
R
count_matrix <- read.csv("data/count_matrix.csv",
row.names = 1) %>%
as.matrix()
count_matrix[1:5, ]
出力
GSM2545336 GSM2545337 GSM2545338 GSM2545339 GSM2545340 GSM2545341
Asl 1170 361 400 586 626 988
Apod 36194 10347 9173 10620 13021 29594
Cyp2d22 4060 1616 1603 1901 2171 3349
Klk6 287 629 641 578 448 195
Fcrls 85 233 244 237 180 38
GSM2545342 GSM2545343 GSM2545344 GSM2545345 GSM2545346 GSM2545347
Asl 836 535 586 597 938 1035
Apod 24959 13668 13230 15868 27769 34301
Cyp2d22 3122 2008 2254 2277 2985 3452
Klk6 186 1101 537 567 327 233
Fcrls 68 375 199 177 89 67
GSM2545348 GSM2545349 GSM2545350 GSM2545351 GSM2545352 GSM2545353
Asl 494 481 666 937 803 541
Apod 11258 11812 15816 29242 20415 13682
Cyp2d22 1883 2014 2417 3678 2920 2216
Klk6 742 881 828 250 798 710
Fcrls 300 233 231 81 303 285
GSM2545354 GSM2545362 GSM2545363 GSM2545380
Asl 473 748 576 1192
Apod 11088 15916 11166 38148
Cyp2d22 1821 2842 2011 4019
Klk6 894 501 598 259
Fcrls 248 179 184 68R
dim(count_matrix)
出力
[1] 1474 22- サンプルを説明する表、 こちら。
R
sample_metadata <- read.csv("data/sample_metadata.csv")
sample_metadata
出力
sample organism age sex infection strain time tissue mouse
1 GSM2545336 Mus musculus 8 Female InfluenzaA C57BL/6 8 Cerebellum 14
2 GSM2545337 Mus musculus 8 Female NonInfected C57BL/6 0 Cerebellum 9
3 GSM2545338 Mus musculus 8 Female NonInfected C57BL/6 0 Cerebellum 10
4 GSM2545339 Mus musculus 8 Female InfluenzaA C57BL/6 4 Cerebellum 15
5 GSM2545340 Mus musculus 8 Male InfluenzaA C57BL/6 4 Cerebellum 18
6 GSM2545341 Mus musculus 8 Male InfluenzaA C57BL/6 8 Cerebellum 6
7 GSM2545342 Mus musculus 8 Female InfluenzaA C57BL/6 8 Cerebellum 5
8 GSM2545343 Mus musculus 8 Male NonInfected C57BL/6 0 Cerebellum 11
9 GSM2545344 Mus musculus 8 Female InfluenzaA C57BL/6 4 Cerebellum 22
10 GSM2545345 Mus musculus 8 Male InfluenzaA C57BL/6 4 Cerebellum 13
11 GSM2545346 Mus musculus 8 Male InfluenzaA C57BL/6 8 Cerebellum 23
12 GSM2545347 Mus musculus 8 Male InfluenzaA C57BL/6 8 Cerebellum 24
13 GSM2545348 Mus musculus 8 Female NonInfected C57BL/6 0 Cerebellum 8
14 GSM2545349 Mus musculus 8 Male NonInfected C57BL/6 0 Cerebellum 7
15 GSM2545350 Mus musculus 8 Male InfluenzaA C57BL/6 4 Cerebellum 1
16 GSM2545351 Mus musculus 8 Female InfluenzaA C57BL/6 8 Cerebellum 16
17 GSM2545352 Mus musculus 8 Female InfluenzaA C57BL/6 4 Cerebellum 21
18 GSM2545353 Mus musculus 8 Female NonInfected C57BL/6 0 Cerebellum 4
19 GSM2545354 Mus musculus 8 Male NonInfected C57BL/6 0 Cerebellum 2
20 GSM2545362 Mus musculus 8 Female InfluenzaA C57BL/6 4 Cerebellum 20
21 GSM2545363 Mus musculus 8 Male InfluenzaA C57BL/6 4 Cerebellum 12
22 GSM2545380 Mus musculus 8 Female InfluenzaA C57BL/6 8 Cerebellum 19R
dim(sample_metadata)
出力
[1] 22 9- 遺伝子を説明する表、 こちら。
R
gene_metadata <- read.csv("data/gene_metadata.csv")
gene_metadata[1:10, 1:4]
出力
gene ENTREZID
1 Asl 109900
2 Apod 11815
3 Cyp2d22 56448
4 Klk6 19144
5 Fcrls 80891
6 Slc2a4 20528
7 Exd2 97827
8 Gjc2 118454
9 Plp1 18823
10 Gnb4 14696
product
1 argininosuccinate lyase, transcript variant X1
2 apolipoprotein D, transcript variant 3
3 cytochrome P450, family 2, subfamily d, polypeptide 22, transcript variant 2
4 kallikrein related-peptidase 6, transcript variant 2
5 Fc receptor-like S, scavenger receptor, transcript variant X1
6 solute carrier family 2 (facilitated glucose transporter), member 4
7 exonuclease 3'-5' domain containing 2
8 gap junction protein, gamma 2, transcript variant 1
9 proteolipid protein (myelin) 1, transcript variant 1
10 guanine nucleotide binding protein (G protein), beta 4, transcript variant X2
ensembl_gene_id
1 ENSMUSG00000025533
2 ENSMUSG00000022548
3 ENSMUSG00000061740
4 ENSMUSG00000050063
5 ENSMUSG00000015852
6 ENSMUSG00000018566
7 ENSMUSG00000032705
8 ENSMUSG00000043448
9 ENSMUSG00000031425
10 ENSMUSG00000027669R
dim(gene_metadata)
出力
[1] 1474 9これらのテーブルから SummarizedExperiment
を作成する:
として使用されるカウント行列。
サンプルを記述したテーブルは、サンプル メタデータスロットとして使用される。
遺伝子を記述したテーブルは、features メタデータスロットとして使用される。
これを行うには、 SummarizedExperiment
コンストラクタを使って、異なるパーツをまとめることができる:
R
## BiocManager::install("SummarizedExperiment")
library("SummarizedExperiment")
エラー
Error in library("SummarizedExperiment"): there is no package called 'SummarizedExperiment'まず、 カウントマトリックスとサンプルアノテーションにおいて、サンプルの順番が同じであることを確認する。また、 カウントマトリックスと遺伝子アノテーションにおいて、遺伝子の順番が同じであることを確認する。
R
stopifnot(rownames(count_matrix) == gene_metadata$gene)
stopifnot(colnames(count_matrix) == sample_metadata$sample)
R
se <- SummarizedExperiment(assays = list(counts = count_matrix),
colData = sample_metadata,
rowData = gene_metadata)
エラー
Error in SummarizedExperiment(assays = list(counts = count_matrix), colData = sample_metadata, : could not find function "SummarizedExperiment"R
se
エラー
Error in eval(expr, envir, enclos): object 'se' not foundデータの保存
以前のエピソードで行ったように、データをスプレッドシートにエクスポートするには、
、第1章
(小数点以下の区切り文字に,と.を使った場合の不整合の可能性、
変数型の定義の欠如)で説明したようないくつかの制限がある。 さらに、
スプレッドシートへのデータエクスポートは、データフレーム
や行列のような長方形のデータにのみ関係する。
データを保存するより一般的な方法は、Rに特有であり、
どのオペレーティングシステムでも動作することが保証されている
saveRDS 関数を使用することである。
このようにオブジェクトを保存すると、ディスク上にバイナリ
表現が生成されます(ここでは rds
ファイル拡張子を使用します)。 readRDS 関数を使用して R
にロードし直すことができます。
R
saveRDS(se, file = "data_output/se.rds")
rm(se)
se <- readRDS("data_output/se.rds")
head(se)
結論として、Rからデータを保存し、
Rで再度ロードする場合、saveRDSとreadRDSで保存とロードを行うのが
。 表形式のデータを、Rを使用していない誰か(
)と共有する必要がある場合は、テキストベースのスプレッドシートにエクスポートするのが、
良い選択肢である。
このデータ構造を使って、
assay関数で発現行列にアクセスすることができる:
R
head(assay(se))
エラー
Error in assay(se): could not find function "assay"R
dim(assay(se))
エラー
Error in assay(se): could not find function "assay"colData`関数を使ってサンプルのメタデータにアクセスすることができる:
R
colData(se)
エラー
Error in colData(se): could not find function "colData"R
dim(colData(se))
エラー
Error in colData(se): could not find function "colData"また、rowData関数を使ってフィーチャーのメタデータにアクセスすることもできる:
R
head(rowData(se))
エラー
Error in rowData(se): could not find function "rowData"R
dim(rowData(se))
エラー
Error in rowData(se): could not find function "rowData"SummarizedExperimentをサブセットする
SummarizedExperiment は、データフレームと同じように、 数値または論理の文字でサブセットできる。
以下では、 、3つの最初のサンプルの5つの最初の特徴のみを含む、SummarizedExperimentクラスの新しいインスタンスを作成します。
R
se1 <- se[1:5, 1:3]
エラー
Error in eval(expr, envir, enclos): object 'se' not foundR
se1
エラー
Error in eval(expr, envir, enclos): object 'se1' not foundR
colData(se1)
エラー
Error in colData(se1): could not find function "colData"R
rowData(se1)
エラー
Error in rowData(se1): could not find function "rowData"また、colData() 関数を使用して、
サンプルメタデータから何かをサブセットしたり、rowData()
関数を使用して、
フィーチャーメタデータから何かをサブセットすることもできます。
例えば、ここではmiRNAと に感染していないサンプルだけを残している:
R
se1 <- se[rowData(se)$gene_biotype == "miRNA",
colData(se)$infection == "NonInfected"]
エラー
Error in eval(expr, envir, enclos): object 'se' not foundR
se1
エラー
Error in eval(expr, envir, enclos): object 'se1' not foundR
assay(se1)
エラー
Error in assay(se1): could not find function "assay"R
colData(se1)
エラー
Error in colData(se1): could not find function "colData"R
rowData(se1)
エラー
Error in rowData(se1): could not find function "rowData"R
assay(se)[1:3, colData(se)$time != 4]
エラー
Error in assay(se): could not find function "assay"R
# Equivalent to
assay(se)[1:3, colData(se)$time == 0 | colData(se)$time == 8]
エラー
Error in assay(se): could not find function "assay"R
rna |>
filter(gene %in% c("Asl", "Apod", "Cyd2d22"))|>
filter(time != 4) |> select(expression)
出力
# A tibble: 28 × 1
expression
<dbl>
1 1170
2 36194
3 361
4 10347
5 400
6 9173
7 988
8 29594
9 836
10 24959
# ℹ 18 more rows長いテーブルとSummarizedExperimentは同じ
情報を含むが、単に構造が異なるだけである。 各アプローチにはそれぞれ
独自の利点がある。前者は tidyverse パッケージに適しており、
一方、後者は多くのバイオインフォマティクスと
統計処理ステップに適した構造である。 例えば、
DESeq2パッケージを使用した典型的なRNA-Seq分析である。
メタデータに変数を追加する
メタデータに情報を追加することもできる。 サンプルが採取されたセンターを追加したいとします…
R
colData(se)$center <- rep("University of Illinois", nrow(colData(se)))
エラー
Error in colData(se): could not find function "colData"R
colData(se)
エラー
Error in colData(se): could not find function "colData"これは、メタデータ・スロットが、 、他の構造に影響を与えることなく、無限に成長できることを示している!
tidySummarizedExperiment
SummarizedExperiment オブジェクトを操作するために
tidyverse コマンドを使うことはできるのだろうか?
tidySummarizedExperiment パッケージを使えば可能です。
SummarizedExperimentオブジェクトがどのようなものか思い出してください:
R
シー
エラー
Error in eval(expr, envir, enclos): object 'シー' not foundtidySummarizedExperiment`をロードし、seオブジェクト 。
R
#BiocManager::install("tidySummarizedExperiment")
library("tidySummarizedExperiment")
エラー
Error in library("tidySummarizedExperiment"): there is no package called 'tidySummarizedExperiment'R
se
エラー
Error in eval(expr, envir, enclos): object 'se' not foundこれはまだSummarizedExperimentオブジェクトなので、効率的な
構造を維持しているが、これでティブルとして見ることができる。
の最初の行に注目してほしい。出力にはこう書いてあるが、これは
SummarizedExperiment-tibble の抽象化である。
また、出力の2行目には、
のトランスクリプトとサンプルの数を見ることができる。
標準のSummarizedExperimentビューに戻したい場合は、
。
R
options("restore_SummarizedExperiment_show" = TRUE)
se
エラー
Error in eval(expr, envir, enclos): object 'se' not foundしかし、ここではティブル・ビューを使う。
R
options("restore_SummarizedExperiment_show" = FALSE)
se
エラー
Error in eval(expr, envir, enclos): object 'se' not foundSummarizedExperiment
オブジェクトと対話するために、tidyverse
コマンドを使用できるようになりました。
filter`を使用すると、条件を使って行をフィルタリングすることができる。例えば、 、あるサンプルのすべての行を表示することができる。
R
se %>% filter(.sample == "GSM2545336")
エラー
Error in eval(expr, envir, enclos): object 'se' not foundselect`を使って表示したいカラムを指定することができる。
R
se %>% select(.sample)
エラー
Error in eval(expr, envir, enclos): object 'se' not foundmutate`を使ってメタデータ情報を追加することができる。
R
se %>% mutate(center = "ハイデルベルク大学")
エラー
Error in eval(expr, envir, enclos): object 'se' not foundtidyverseパイプ %>%
を使ってコマンドを組み合わせることもできます。
の例では、group_by と summarise
を組み合わせて、各サンプルの カウントの合計を得ることができる。
R
se %>%
group_by(.sample) %>%
summarise(total_counts=sum(counts))
エラー
Error in eval(expr, envir, enclos): object 'se' not found整頓されたSummarizedExperimentオブジェクトを、プロット用の通常のtibble として扱うことができる。
ここでは、サンプルごとのカウント数分布をプロットしている。
R
se %>%
ggplot(aes(counts + 1, group=.sample, color=infection))+
geom_density() +
scale_x_log10() +
theme_bw()
エラー
Error in eval(expr, envir, enclos): object 'se' not foundtidySummarizedExperimentの詳細については、パッケージ ウェブサイト こちらを参照してください。
**テイクホーム・メッセージ
SummarizedExperiment`は、オミックスデータを効率的に保存し、 。
これらは多くのBioconductorパッケージで使用されている。
RNAシーケンス解析に焦点を当てた次のトレーニング、 、Bioconductor
DESeq2パッケージを使って、 差分発現解析を行う方法を学ぶ。
DESeq2パッケージの全解析はSummarizedExperiment`
で処理される。