Content from はじめに
最終更新日:2025-04-15 | ページの編集
概要
質問
- 再現性を重視すべき理由は何ですか?
-
targetsはどのようにして再現性の達成を助けますか?
目的
- 再現性が科学にとってなぜ重要なのかを説明する
- 再現性を高める
targetsの機能について説明する
再現性とは何ですか?
再現性とは、他の人(将来の自分自身を含む)があなたの分析を再現できる能力のことです。
科学的な分析の結果に自信を持つためには、それらが再現可能でなければなりません。
しかし、再現性は二元的な概念(再現不可能 vs. 再現可能)ではなく、再現性が低いから再現性が高いまでのスケールがあります。
targets は分析を より再現性の高いもの
にするために大きく貢献します。
再現性をさらに高めるために使用できる他の方法としては、Docker、conda、renv などのツールを使用してコンピューティング環境を管理することがありますが、このワークショップではそれらを扱う時間がありません。
targets とは何ですか?
targets は、Will Landau
氏によって開発および維持されている R
プログラミング言語向けのワークフローマネジメントパッケージです。
targets の主な機能には以下が含まれます:
- ワークフローの自動化
- ワークフローのステップのキャッシング
- ワークフローのステップのバッチ作成
- ワークフローのレベルでの並列化
これにより、以下のことが可能になります:
- 他の作業をした後にプロジェクトに戻り、混乱や何をしていたかを思い出すことなくすぐに作業を再開できる
- ワークフローを変更した場合、変更の影響を受ける部分のみを再実行できる
- 個々の関数を変更することなくワークフローを大規模に拡張できる
… そしてもちろん、他の人があなたの分析を再現するのに役立ちます。
誰が targets を使用すべきですか?
targets
はワークフローマネジメントソフトウェアの唯一のものではありません。
類似のツールは数多く存在し、それぞれに異なる機能やユースケースがあります。
例えば、snakemake は
Python 向けの人気のあるワークフローツールであり、make
は長い歴史を持つ Bash スクリプトの自動化ツールです。
targets は特に R と連携するように設計されているため、主に R
を使用する場合、または R を使用する予定がある場合に最も適しています。
他のツールを主に使用する場合は、代替手段を検討することをお勧めします。
このワークショップの目標は、R
で再現可能なデータ分析を行うために targets
の使い方を学ぶことです。
さらに情報を得るには
targets
は高度なパッケージであり、このワークショップでカバーできる以上に学ぶことがたくさんあります。
targets
の学習を続けるためのおすすめリソースは以下の通りです:
-
targetsの作者である Will Landau氏 による The targets R package user manual は、targetsに真剣に興味がある人にとって必読とすべきです。 - targets discussion board は質問をしたり助けを得たりするのに最適な場所です。ただし、質問をする前に必ず help のポリシーを読む ことを確認してください。
-
targetsのウエブサイト
には、すべての
targets関数のドキュメントが含まれています。 -
tarchetypesのウエブサイト
には、すべての
tarchetypes関数のドキュメントが含まれています。tarchetypesはほぼ確実にtargetsと一緒に使用するため、両方を参照することをお勧めします。 - Reproducible computation at scale in R with targets は、Keras を用いて顧客の離脱を分析する Will Landau氏 によるチュートリアルです。
-
Recorded
talks および example
projects は、
targetsの README に記載されています。
例示データセットについて
このワークショップでは、南極のパルマー群島の島々で観察された成体の採餌アデリーペンギン、チンストラップペンギン、ジェンツーペンギンの測定データを分析します。
データは palmerpenguins R
パッケージから入手可能です。データに関する詳細情報は
?palmerpenguins を実行することで得られます。

palmerpenguins
データセットの3種のペンギン。アートワーク:@allison_horst.分析の目標は、線形モデルを使用してくちばしの長さと深さの関係を明らかにすることです。
このレッスンを通じて分析を段階的に構築しますが、最終版は https://github.com/joelnitta/penguins-targets で見ることができます。
まとめ
- 科学的分析の結果に自信を持つためには、他の人(将来の自分自身を含む)がそれを再現できなければならない
-
targetsはワークフローの自動化によって再現性の達成を助ける -
targetsは R プログラミング言語と連携するように設計されている - このワークショップの例示データセットには、南極のペンギンの測定データが含まれている
Content from 初めての targets ワークフロー
最終更新日:2025-04-15 | ページの編集
概要
質問
- 分析を整理するためのベストプラクティスは何ですか?
-
_targets.Rファイルは何のためのものですか? -
_targets.Rファイルの内容は何ですか? - ワークフローを実行するにはどうしますか?
目的
- RStudioでプロジェクトを作成する
-
_targets.Rファイルの目的を説明する - 基本的な
_targets.Rファイルを書く -
_targets.Rファイルを使用してワークフローを実行する
プロジェクトの作成
プロジェクトについて
targets
は分析を整理するために「プロジェクト」の概念を使用します。特定のプロジェクトに必要なすべてのファイルを1つのフォルダ、プロジェクトフォルダにまとめます。
プロジェクトフォルダには、データ、コード、結果用のフォルダなど、整理のための追加のサブフォルダがあります。
プロジェクトを使用することで、他の作業に時間を費やした後に分析に戻った際に、簡単に再び方向付けることができます。
もし一度に1つの作業のみを完了させる場合は問題になりませんが、実際にはほとんどの場合そうではありません。
他の作業をした後にプロジェクトに戻るときに、何をしていたかを覚えておくのは難しいです(「コンテキストスイッチング」と呼ばれる現象)。
標準化された整理システムを使用することで、混乱や時間の浪費を減らすことができます。つまり、再現性を高めることになります!
このワークショップでは、プロジェクトの整理概念ともうまく連携する RStudio を使用します。
RStudioでプロジェクトを作成する
RStudioを使用して新しいプロジェクトを開始しましょう。
「ファイル」をクリックし、「新しいプロジェクト」を選択します。
これにより、新しいプロジェクトウィザードが開き、プロジェクトの設定を手助けする一連のメニューが表示されます。
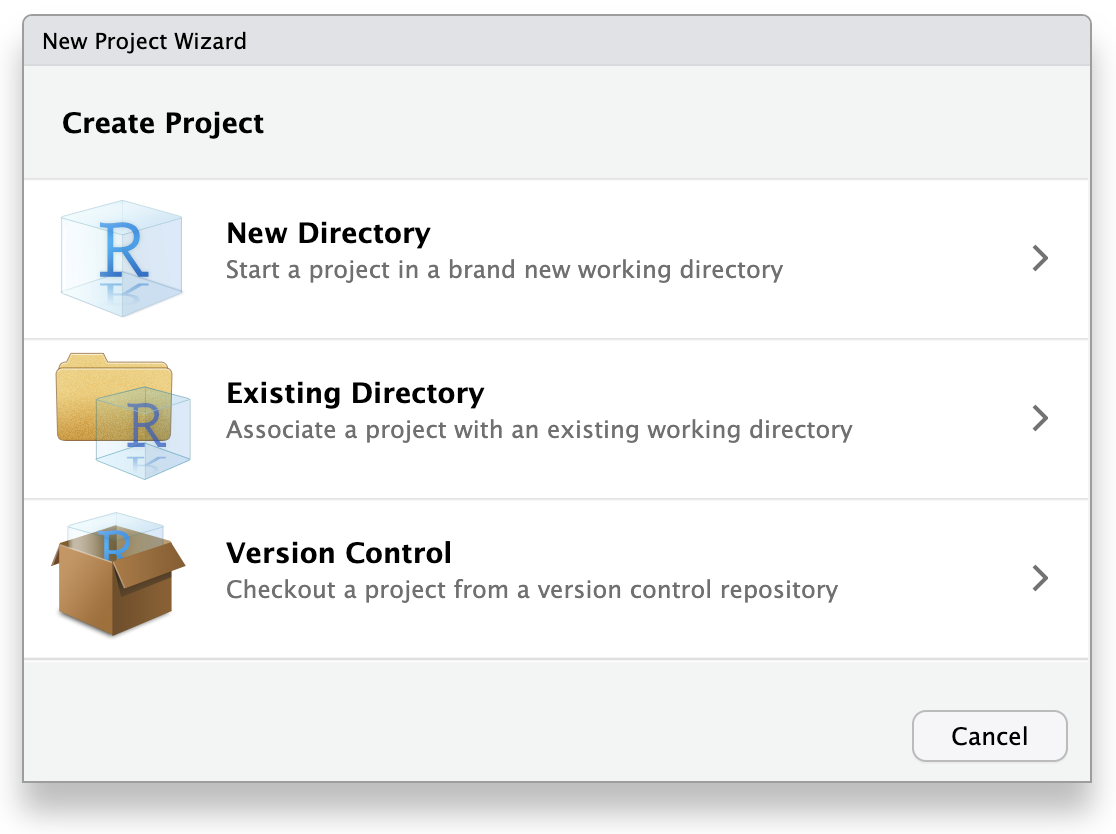
ウィザードで最初のオプション「新しいディレクトリ」をクリックします。これは、新しいプロジェクトをゼロから作成するためです。
次のメニューで「新しいプロジェクト」をクリックします。
「ディレクトリ名」には、プロジェクトの目的を思い出しやすい名前を入力します。例えば「targets-demo」(ファイルやフォルダの命名のベストプラクティスに従ってください)。
「プロジェクトをサブディレクトリとして作成する…」の下で、「参照」ボタンをクリックしてプロジェクトを配置するディレクトリを選択します。
プロジェクトを簡単に見つけられるように、デスクトップに配置することをお勧めします。
「Gitリポジトリを作成」と「このプロジェクトで renv を使用する」はチェックを外したままにできますが、これらは再現性を向上させる優れたツールです。もしまだであれば、将来的に学習して使用することを検討してください。
これらは後からでも有効にできるため、すぐに使用しようと心配する必要はありません。
これらの手順を進めると、RStudioのセッションは次のようになります:
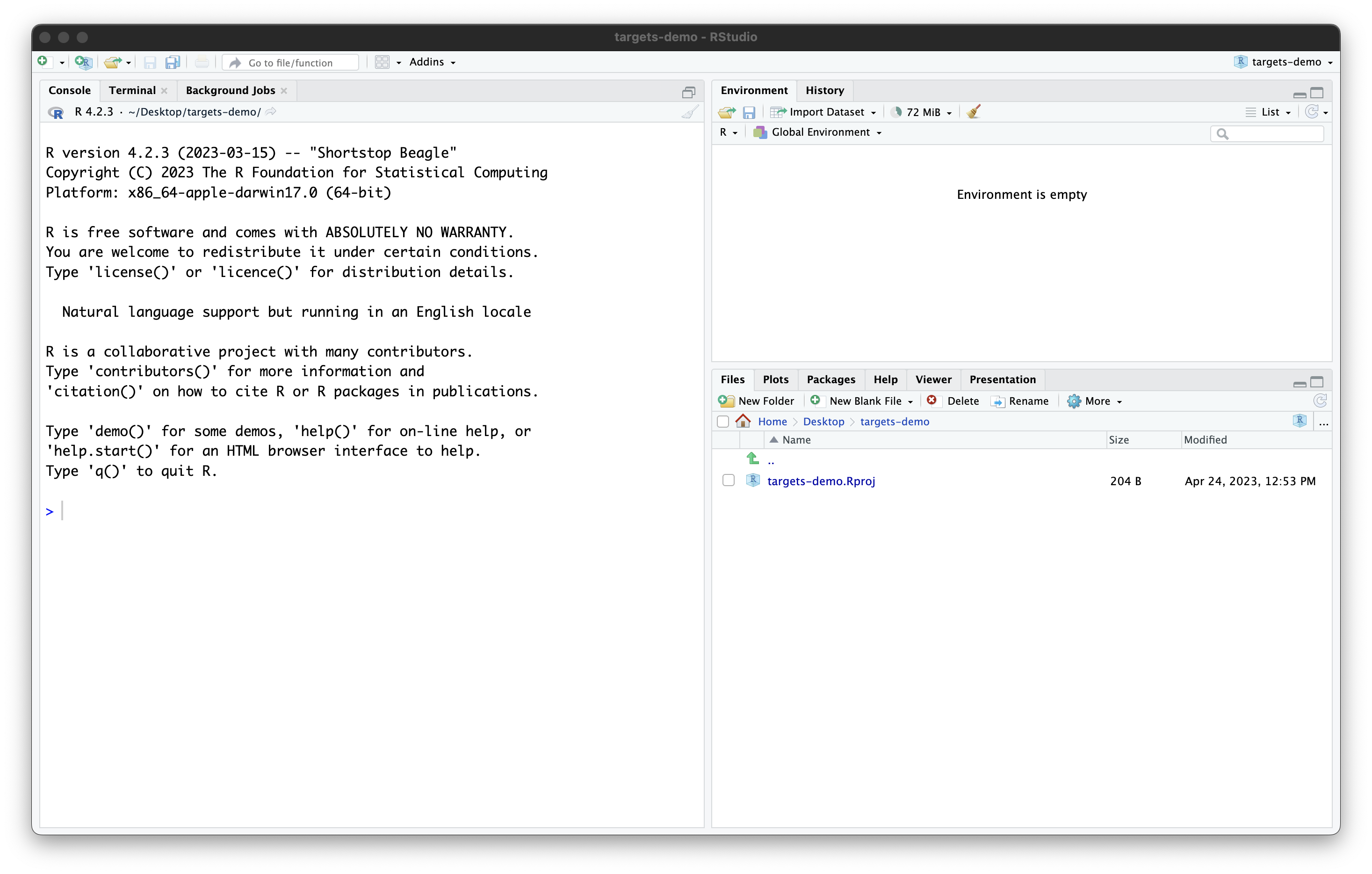
プロジェクトには現在、RStudioによって作成された1つのファイル
targets-demo.Rproj
が含まれています。このファイルを手動で編集しないでください。その目的は、RStudioにこのフォルダがプロジェクトフォルダであることを伝え、いくつかのRStudio設定を保存することです(バージョン管理ソフトウェアを使用している場合は、このファイルをコミットしても構いません)。また、ファイルエクスプローラーで
.Rproj
ファイルをダブルクリックすることでプロジェクトを開くことができます(RStudioを終了してからファイルブラウザでデスクトップに移動し、「targets-demo」フォルダを開いて
targets-demo.Rproj
をダブルクリックして試してください)。
さて、プロジェクトが設定されたので、targets
の使用を開始する準備ができました!
_targets.R ファイルの作成
すべての targets
プロジェクトには、メインプロジェクトフォルダ(「プロジェクトルート」)にある特別なファイル
_targets.R を含める必要があります。
_targets.R
ファイルにはワークフローの仕様が含まれており、Rに分析を実行する指示が記述されています。これはレシピのようなものです。
_targets.R
ファイルを使用することで、特定のスクリプトを特定の順序で実行することを覚えておく必要がなくなります。
代わりに、Rがそれを自動的に実行してくれます(再現性が向上します)!
_targets.R ファイルの構成
これから _targets.R
ファイルの作成を開始します。幸い、targets
にはこれを手助けする関数が用意されています。
Rコンソールで、まず library(targets) で
targets パッケージを読み込み、次に
tar_script() コマンドを実行します。
R
library(targets)
tar_script()
コンソールでは何も起こりませんが、ファイルビューアに新しいファイル
_targets.R
が表示されます。ファイルメニューを使用するか、クリックして開いてください。
このデフォルトの _targets.R
ファイルには3つの主要な部分が含まれています:
-
library()を使用したパッケージの読み込み -
function()を使用したカスタム関数の定義 -
list()を使用したリストの定義
最後の部分であるリストは、_targets.R
ファイルの中で最も重要な部分です。
ワークフローのステップを定義します。
_targets.R
ファイルは常にこのリストで終わらなければなりません。
さらに、リスト内の各項目は tar_target()
関数の呼び出しです。
tar_target()
の最初の引数はビルドするターゲットの名前で、2番目の引数はそれをビルドするために使用するコマンドです。
ターゲットの名前は引用符なし、つまり、引用符で囲まれていないことに注意してください。
例示分析を実行するための _targets.R ファイルの設定
背景: targets を使用しないバージョン
このテンプレートを使用して、ペンギンのくちばしの形状の分析を構築し始めます。
しかしまず、使用する関数やパッケージに慣れるために、targets
を使用せずに「通常の」Rスクリプトでコードを実行してみましょう。
データを取得するために palmerpenguins
Rパッケージを使用していることを思い出してください。
このパッケージには実際にデータセットの2つのバリエーションが含まれています。1つは生データを含む外部CSVファイルで、もう1つはRに読み込まれたクリーンなデータです。
実際のところ、生データは外部に保存されていることが多いため、生のペンギンデータ を分析の出発点として使用しましょう。
palmerpenguins の path_to_file()
関数は、生データCSVファイルへのパスを提供します(これは、パッケージをインストールしたときにコンピュータにダウンロードされた
palmerpenguins
Rパッケージのソースコード内にあります)。
R
library(palmerpenguins)
# Get path to CSV file
penguins_csv_file <- path_to_file("penguins_raw.csv")
penguins_csv_file
出力
[1] "/home/runner/.local/share/renv/cache/v5/linux-ubuntu-jammy/R-4.4/x86_64-pc-linux-gnu/palmerpenguins/0.1.1/6c6861efbc13c1d543749e9c7be4a592/palmerpenguins/extdata/penguins_raw.csv"データの読み込みと操作には、tidyverse
パッケージ群を使用します。
今は tidyverse
の使用方法のすべての詳細をカバーする時間がありませんが、詳細を学びたい場合は、“tidyverse
を使用したデータの操作、分析、およびエクスポート” レッスン
を参照してください。
read_csv() を使用してデータを読み込みましょう。
R
library(tidyverse)
# Read CSV file into R
penguins_data_raw <- read_csv(penguins_csv_file)
penguins_data_raw
出力
Rows: 344 Columns: 17
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (9): studyName, Species, Region, Island, Stage, Individual ID, Clutch C...
dbl (7): Sample Number, Culmen Length (mm), Culmen Depth (mm), Flipper Leng...
date (1): Date Egg
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.出力
# A tibble: 344 × 17
studyName `Sample Number` Species Region Island Stage `Individual ID`
<chr> <dbl> <chr> <chr> <chr> <chr> <chr>
1 PAL0708 1 Adelie Penguin… Anvers Torge… Adul… N1A1
2 PAL0708 2 Adelie Penguin… Anvers Torge… Adul… N1A2
3 PAL0708 3 Adelie Penguin… Anvers Torge… Adul… N2A1
4 PAL0708 4 Adelie Penguin… Anvers Torge… Adul… N2A2
5 PAL0708 5 Adelie Penguin… Anvers Torge… Adul… N3A1
6 PAL0708 6 Adelie Penguin… Anvers Torge… Adul… N3A2
7 PAL0708 7 Adelie Penguin… Anvers Torge… Adul… N4A1
8 PAL0708 8 Adelie Penguin… Anvers Torge… Adul… N4A2
9 PAL0708 9 Adelie Penguin… Anvers Torge… Adul… N5A1
10 PAL0708 10 Adelie Penguin… Anvers Torge… Adul… N5A2
# ℹ 334 more rows
# ℹ 10 more variables: `Clutch Completion` <chr>, `Date Egg` <date>,
# `Culmen Length (mm)` <dbl>, `Culmen Depth (mm)` <dbl>,
# `Flipper Length (mm)` <dbl>, `Body Mass (g)` <dbl>, Sex <chr>,
# `Delta 15 N (o/oo)` <dbl>, `Delta 13 C (o/oo)` <dbl>, Comments <chr>生データにはスペースを含む扱いにくい列名があり(これらはタイプミスしやすくコードのミスにつながりやすい)、必要なものよりもはるかに多くの列があります。
この分析の目的では、種名、くちばしの長さ、くちばしの深さのみが必要です。
生データでは、「culmen」というやや技術的な用語がくちばしを指すために使用されています。
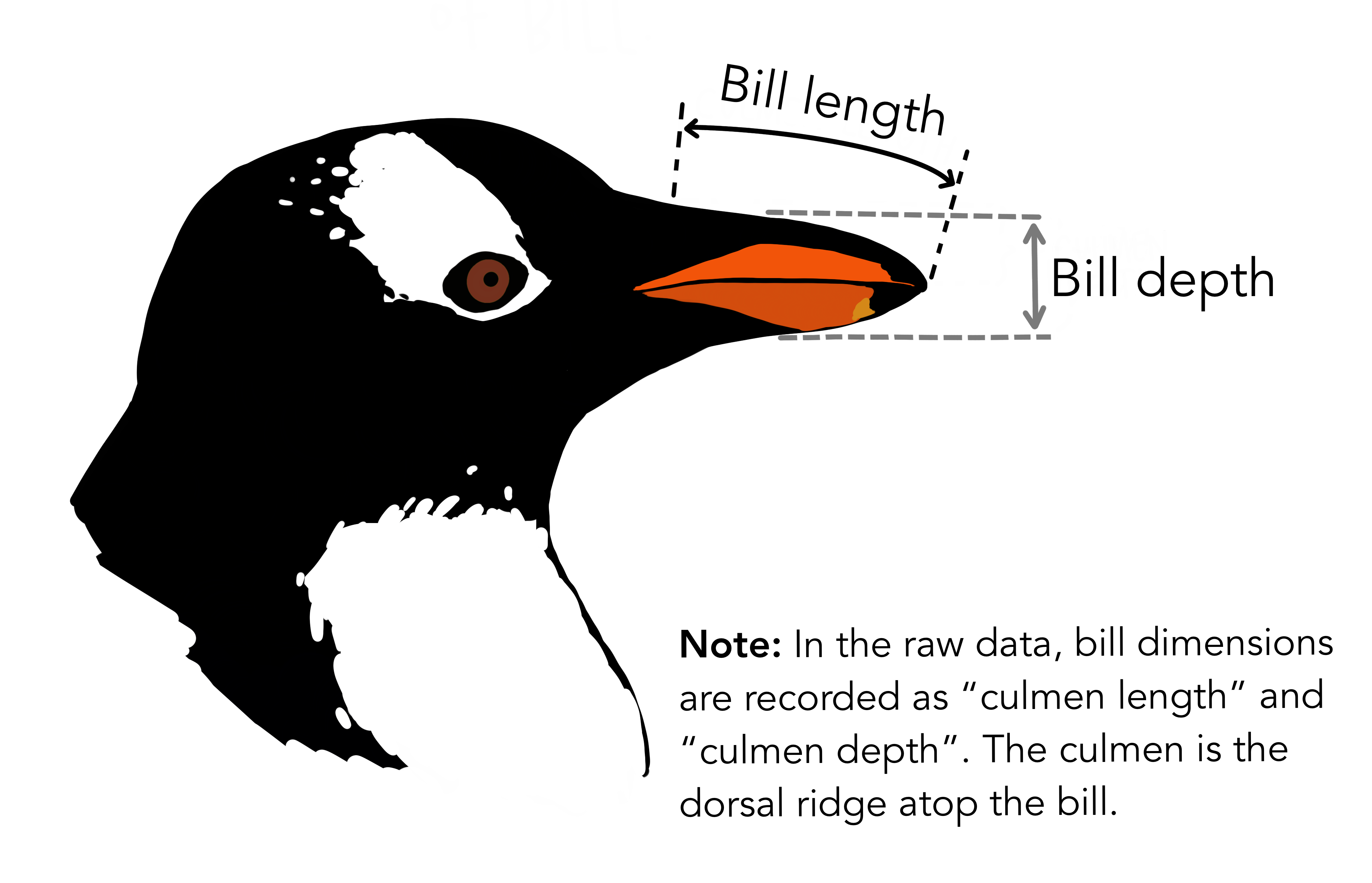
下流の分析で使用しやすくするためにデータを整理しましょう。
後で一部の関数でエラーを引き起こす可能性があるため、欠損データを含む行も削除します。
R
# Clean up raw data
penguins_data <- penguins_data_raw |>
# Rename columns for easier typing and
# subset to only the columns needed for analysis
select(
species = Species,
bill_length_mm = `Culmen Length (mm)`,
bill_depth_mm = `Culmen Depth (mm)`
) |>
# Delete rows with missing data
remove_missing(na.rm = TRUE)
penguins_data
出力
# A tibble: 342 × 3
species bill_length_mm bill_depth_mm
<chr> <dbl> <dbl>
1 Adelie Penguin (Pygoscelis adeliae) 39.1 18.7
2 Adelie Penguin (Pygoscelis adeliae) 39.5 17.4
3 Adelie Penguin (Pygoscelis adeliae) 40.3 18
4 Adelie Penguin (Pygoscelis adeliae) 36.7 19.3
5 Adelie Penguin (Pygoscelis adeliae) 39.3 20.6
6 Adelie Penguin (Pygoscelis adeliae) 38.9 17.8
7 Adelie Penguin (Pygoscelis adeliae) 39.2 19.6
8 Adelie Penguin (Pygoscelis adeliae) 34.1 18.1
9 Adelie Penguin (Pygoscelis adeliae) 42 20.2
10 Adelie Penguin (Pygoscelis adeliae) 37.8 17.1
# ℹ 332 more rowsこれで良くなりました!
targets バージョン
targets を使用するとどのようになりますか?
最大の違いは、ワークフローの各ステップを最後のリストに追加する必要があることです。
データクリーニングステップのためにカスタム関数も定義します。
これは、最後のターゲットのリストが分析の高レベルな要約のように見えるべきだからです。
ターゲットを定義するときに長いコードの塊を避けたいです。代わりに、そのコードをカスタム関数に入れます。
他のステップ(ファイルパスの設定とデータの読み込み)はそれぞれ1つの関数呼び出しだけなので、それらを独自のカスタム関数に入れる意味はあまりありません。
最後に、ワークフローの各ステップは tar_target()
関数で定義されます。
R
library(targets)
library(tidyverse)
library(palmerpenguins)
clean_penguin_data <- function(penguins_data_raw) {
penguins_data_raw |>
select(
species = Species,
bill_length_mm = `Culmen Length (mm)`,
bill_depth_mm = `Culmen Depth (mm)`
) |>
remove_missing(na.rm = TRUE)
}
list(
tar_target(penguins_csv_file, path_to_file("penguins_raw.csv")),
tar_target(penguins_data_raw, read_csv(
penguins_csv_file, show_col_types = FALSE)),
tar_target(penguins_data, clean_penguin_data(penguins_data_raw))
)
read_csv() で show_col_types = FALSE
に設定したのは、先ほどのコードから列の型がデフォルトで正しく設定されている(種には文字列、くちばしの長さと深さには数値)ことがわかっているためです。したがって、通常発生する警告を表示する必要はありません。
ワークフローの実行
ワークフローができたので、tar_make()
関数を使用して実行できます。
それを実行してみてください。次のようなものが表示されるはずです:
R
tar_make()
出力
▶ dispatched target penguins_csv_file
● completed target penguins_csv_file [0.001 seconds]
▶ dispatched target penguins_data_raw
● completed target penguins_data_raw [0.098 seconds]
▶ dispatched target penguins_data
● completed target penguins_data [0.007 seconds]
▶ ended pipeline [0.236 seconds]おめでとうございます、targets
を使って最初のワークフローを実行しました!
まとめ
- プロジェクトは分析を整理しておくのに役立ち、後で簡単に再実行できます
- RStudioのプロジェクトウィザードを使用してプロジェクトを作成する
-
_targets.Rファイルはすべてのtargetsプロジェクトに含める必要がある特別なファイルであり、ワークフローを定義します -
tar_script()を使用してデフォルトの_targets.Rファイルを作成する -
tar_make()を使用してワークフローを実行する
Content from ワークフローオブジェクトの読み込み
最終更新日:2025-04-15 | ページの編集
概要
質問
- ワークフローはどこで実行されますか?
- ワークフローによって作成されたオブジェクトをどのように検査できますか?
目的
-
targetsがワークフローを実行する場所とその理由を説明する - ワークフローによって作成されたオブジェクトをRセッションにロードできるようにする
ワークフローはどこで実行されますか?
私たちはちょうど最初のワークフローを実行しました。
今、出力を見たいと思うでしょう。 しかし、オブジェクトの名前(例えば
penguins_data)を呼び出すだけではエラーが発生します。
R
penguins_data
エラー
Error in eval(expr, envir, enclos): object 'penguins_data' not foundワークフローの結果はどこにありますか?
ワークフローの結果が見えないのは、targets
がワークフローを別のRセッションで実行するためであり、そのセッションと対話することができないからです。
これは再現性のためです—ワークフローによって作成されたオブジェクトは、プロジェクト内のコードのみに依存すべきであり、Rに対してインタラクティブに与えたコマンドには依存すべきではありません。
幸いにも、targets
にはワークフローによって作成されたオブジェクトを現在のセッションにロードするために使用できる2つの関数、tar_load()
と tar_read() があります。
これらがどのように機能するか見てみましょう。
tar_load()
tar_load()
は、ワークフローによって作成されたオブジェクトを現在のセッションにロードします。
最初の引数はロードしたいオブジェクトの名前です。 これを使用して
penguins_data をロードし、summary()
でデータの概要を取得しましょう。
R
tar_load(penguins_data)
summary(penguins_data)
出力
species bill_length_mm bill_depth_mm
Length:342 Min. :32.10 Min. :13.10
Class :character 1st Qu.:39.23 1st Qu.:15.60
Mode :character Median :44.45 Median :17.30
Mean :43.92 Mean :17.15
3rd Qu.:48.50 3rd Qu.:18.70
Max. :59.60 Max. :21.50 tar_load()
は副作用—望むオブジェクトを現在のRセッションにロードするために使用されます。
実際には値を返しません。
tar_read()
tar_read()
は、ワークフローによって作成されたオブジェクトを取得するために使用される点では
tar_load() と似ていますが、tar_load()
とは異なり、それらを直接出力として返します。
penguins_csv_file で試してみましょう。
R
tar_read(penguins_csv_file)
出力
[1] "/home/runner/.local/share/renv/cache/v5/linux-ubuntu-jammy/R-4.4/x86_64-pc-linux-gnu/palmerpenguins/0.1.1/6c6861efbc13c1d543749e9c7be4a592/palmerpenguins/extdata/penguins_raw.csv"penguins_csv_file の内容がすぐに表示されます。
しかし、それは環境にロードされていません。 今
penguins_csv_file
を実行しようとすると、エラーが発生します:
R
penguins_csv_file
エラー
Error in eval(expr, envir, enclos): object 'penguins_csv_file' not foundどの関数をいつ使用するか
tar_load()
はオブジェクトをロードしてそれらを操作したいときにより便利です。
tar_read()
はオブジェクトを即座に検査したいときにより便利です。
targets キャッシュ
Rセッションを終了し、再起動して tar_load() または
tar_read()
を使用すると、ワークフローオブジェクトをまだロードできることに気づくでしょう。
言い換えれば、ワークフローの出力はRセッション間で保存されています。
これはどのように可能なのでしょうか?
プロジェクトに新しいフォルダ _targets
が現れたことに気づいたかもしれません。 これはtargets
キャッシュです。
ワークフローの出力すべてが含まれています。これにより、Rを終了して再起動した後でもワークフローによって作成されたターゲットをロードできるのです。
キャッシュの内容を手動で編集してはいけません(1つの例外を除く)。 そうすると、分析の再現性が失われます。
このルールの唯一の例外は、_targets/user
という特別なサブフォルダです。
このフォルダはデフォルトでは存在しません。
必要であれば作成し、任意のものを中に入れることができます。
一般的に、_targets/user
はデータや出力のようなコードではないファイルを保存するのに適しています。
もし _targets/user
に保持する必要があるものが何もない場合、単に _targets
フォルダ全体を削除することでワークフローを「リセット」することが可能です。
もちろん、これはすべてを再実行する必要があることを意味するため、軽率に行わないでください!
まとめ
-
targetsのワークフローは別の非対話型Rセッションで実行されます -
tar_load()はワークフローオブジェクトを現在のRセッションにロードします -
tar_read()はワークフローオブジェクトを読み取り、その値を返します -
_targetsフォルダはキャッシュであり、一般的には手動で編集すべきではありません
Content from ワークフローのライフサイクル
最終更新日:2025-04-15 | ページの編集
概要
質問
- ワークフローを再実行するとどうなりますか?
-
targetsはどのようにして再実行すべきステップを認識しますか? - ワークフローの状態をどのように検査できますか?
目的
-
targetsが効率性を向上させる方法を説明する - ワークフローの古くなった部分を確認する方法を習得する
ワークフローの再実行
targets
の特徴の一つは、実行が必要なワークフローの部分のみを実行することで効率性を最大化することです。
これは実際に試してみると理解しやすいです。ワークフローをもう一度実行してみましょう:
R
tar_make()
出力
✔ skipped target penguins_csv_file
✔ skipped target penguins_data_raw
✔ skipped target penguins_data
✔ skipped pipeline [0.129 seconds]最初にパイプラインを実行したとき、targets
がビルドされている各ターゲットのリストを出力したことを覚えていますか?
今回は、それらのターゲットをスキップしていると教えてくれます。すでにビルドされているため、同じコードを再度実行する必要がないからです。
覚えておいてください、最速のコードは実行しなくて済むコードです!
変更後のワークフローの再実行
ワークフローの一部を変更してから再度実行するとどうなりますか?
例えば、種名を短くすることに決めたとしましょう。 現在は一般名と学名が含まれていますが、区別するために一般名の最初の部分のみが必要です。
_targets.R を編集して、clean_penguin_data()
関数を以下のようにします:
R
clean_penguin_data <- function(penguins_data_raw) {
penguins_data_raw |>
select(
species = Species,
bill_length_mm = `Culmen Length (mm)`,
bill_depth_mm = `Culmen Depth (mm)`
) |>
remove_missing(na.rm = TRUE) |>
# Split "species" apart on spaces, and only keep the first word
separate(species, into = "species", extra = "drop")
}
そして、再度実行します。
R
tar_make()
出力
✔ skipped target penguins_csv_file
✔ skipped target penguins_data_raw
▶ dispatched target penguins_data
● completed target penguins_data [0.014 seconds]
▶ ended pipeline [0.165 seconds]何が起こったでしょうか?
今回は、penguins_csv_file と
penguins_data_raw をスキップし、penguins_data
のみを実行しました。
もちろん、私たちの例示ワークフローは非常に短いため、節約された時間を感じ取ることはほとんどありません。 しかし、計算集約的な一連の分析ステップでこれを使用することを想像してみてください。 ステップを自動的にスキップする能力は、効率性を大幅に向上させます。
チャレンジ 1: 出力を検査する
penguins_data の内容をどのように検査できますか?
tar_read(penguins_data)
を使用するか、tar_load(penguins_data) を実行した後に
penguins_data を実行します。
内部動作
targets
は、どのターゲットが最新であり、どれが古くなっているかをどのように追跡していますか?
ワークフロー内の各ターゲット(_targets.R
ファイルの最後のリスト内の項目)およびワークフローで使用されるカスタム関数ごとに、targets
はハッシュ値、つまりコンピュータのメモリ内のオブジェクトを表す一意の文字と数字の組み合わせを計算します。
ハッシュ値(略して「ハッシュ」)をターゲットや関数の一意の指紋と考えることができます。
最初に tar_make() を実行すると、targets
はコードを実行しながら各ターゲットと関数のハッシュを計算し、それらを
targets キャッシュ(_targets フォルダ)に保存します。
その後、tar_make()
を呼び出すたびに、再度ハッシュを計算し、保存された値と比較します。
どれが変更されたかを検出し、これがどのターゲットが古くなっているかを知る方法です。
ハッシュが存在する場所
ハッシュがどのように見えるか気になる場合は、ファイル
_targets/meta/meta
に見ることができますが、このファイルを手動で編集しないでください—ワークフローが壊れてしまいます!
この情報は、依存関係の関係(つまり、各ターゲットが他のターゲットにどのように依存しているか)と組み合わせて使用され、ワークフローを可能な限り効率的に再実行します:再構築が必要なターゲットに対してのみコードが実行され、他のターゲットはスキップされます。
ワークフローの可視化
通常、コードのさまざまな場所を編集し、新しいターゲットを追加し、定期的にワークフローを実行することになります。 ワークフローの状態を可視化できることは良いことです。
これは tar_visnetwork()
を使用して行うことができます。
R
tar_visnetwork()
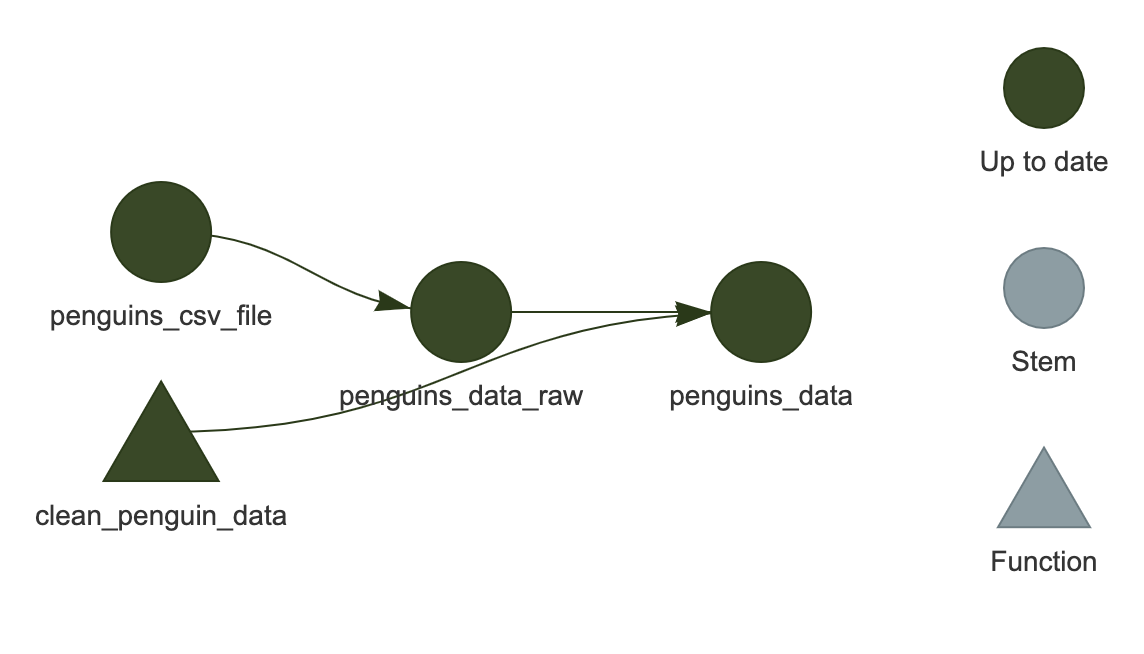
ネットワークが RStudio のプロット領域に表示されるはずです。
これは HTML ウィジェットなので、ズームインやズームアウトが可能です(現在の例では非常に小さいため重要ではありませんが、より大きな「実際の」ワークフローでは役立ちます)。
ここでは、すべてのターゲットが濃い緑色で表示されており、次にワークフローを実行したときにスキップされることを示しています。
visNetwork のインストール
The package "visNetwork" is required.
というエラーメッセージが表示される場合があります。
この場合、まず install.packages("visNetwork")
を実行してインストールしてください。
チャレンジ 2: 可視化は他に何を教えてくれますか?
_targets.R
でワークフローを変更し、tar_make() を実行せずに再度
tar_visnetwork() を実行してください。
ターゲットが古くなっていることを示す色は何ですか?
薄い青色がターゲットが古くなっていることを示します。
コードをどのように変更したかによりますが、ターゲットのいくつかまたはすべてが薄い青色になる場合があります。
‘古くなっている’ は必ずしも ‘実行される’ ことを意味しない
ターゲットがネットワークの可視化で薄い青色(「古くなっている」)として表示されているからといって、次回の実行時に必ず再構築されるわけではありません。これは、依存しているターゲットの少なくとも一つが変更されたことを意味します。
例えば、ワークフローの状態が以下のようになっているとします:
A -> B* -> C -> D
ここで * は B
が最後にワークフローを実行したときと比較して変更されたことを示します。ネットワークの可視化では
B, C, D
がすべて薄い青色で表示されます。
しかし、ワークフローを再実行して C
が以前と全く同じ値になる場合、D
は再実行されません(「スキップ」されます)。
ほとんどの場合、単一の変更が下流のターゲット全体に影響を及ぼし、再構築を引き起こしますが、必ずしもそうではありません。targets
は事前に実際の出力がどうなるかを知る方法がないため、将来を完全に予測するネットワーク可視化を提供することはできません!
ワークフローの状態を確認する他の方法
可視化は非常に便利ですが、時にはグラフィカルな出力を提供しないサーバー上で作業している場合や、ワークフローの迅速なテキストサマリーを望む場合があります。 それを行うための他の有用な関数があります。
tar_outdated()
は古くなったターゲットのみをリストします。つまり、次回の実行時にビルドされるターゲット、またはそのようなターゲットに依存するターゲットです。
すべてが最新の場合、ゼロ長の文字ベクトル (character(0))
を返します。
R
tar_outdated()
出力
character(0)tar_progress()
はワークフローの現在の状態をデータフレームとして表示します。
ワークフローの有用なサマリーを取得するためにデータフレームをさらに操作することが役立つ場合があります。例えば、dplyr
を使用するなど(このデータ操作はこのレッスンの範囲外ですが、インストラクターがその使用を示すかもしれません)。
R
tar_progress()
出力
# A tibble: 3 × 2
name progress
<chr> <chr>
1 penguins_csv_file skipped
2 penguins_data_raw skipped
3 penguins_data completedターゲットの細かい制御
特定のターゲットのみをビルドし、ワークフロー全体を実行しないことが可能です。
これを行うには、tar_make()
の後にビルドしたいターゲットの名前を入力します(指定したターゲットに必要な他のターゲットもビルドされることに注意してください)。
例えば、tar_make(penguins_data_raw) は
penguins_data_raw
のみをビルドし、penguins_data はビルドしません。
さらに、ターゲットを手動で「リセット」し、古くなったように見せるには、tar_invalidate()
を使用できます。これは、次回の実行時にそのターゲット(およびそれに依存するターゲット)が再実行されることを意味します。
これを試してみましょう。現在、私たちのパイプラインは最新の状態なので、tar_make()
はすべてをスキップします:
R
tar_make()
出力
✔ skipped target penguins_csv_file
✔ skipped target penguins_data_raw
✔ skipped target penguins_data
✔ skipped pipeline [0.144 seconds]penguins_data を無効化し、再度実行してみましょう:
R
tar_invalidate(penguins_data)
tar_make()
出力
✔ skipped target penguins_csv_file
✔ skipped target penguins_data_raw
▶ dispatched target penguins_data
● completed target penguins_data [0.015 seconds]
▶ ended pipeline [0.165 seconds]もしすべてをリセットして新たに開始したい場合は、tar_invalidate(everything())
を使用して _targets
フォルダ全体を削除することができます(tar_invalidate()
はターゲット名を指定するために tidyselect
式を受け入れます)。
しかし、これはすべてを再実行する必要があることを意味するため、軽率に行わないでください!
実際の運用での動作
実際には、tar_make()
を使用してワークフローを実行し、tar_load()
を使用してビルドしたターゲットをロードし、インタラクティブな R
セッションでコードを実行してカスタム関数を編集することを行き来することが多いでしょう。慣れるまで時間がかかりますが、すぐに
targets
ワークフローに組み込まれていないコードは「本物ではない」と感じるようになるでしょう。
まとめ
-
targetsはコードの変更に影響を受けたステップのみを実行します -
tar_visnetwork()はワークフローの現在の状態をネットワークとして表示します -
tar_progress()はワークフローの現在の状態をデータフレームとして表示します -
tar_outdated()は古くなったターゲットをリストします -
tar_invalidate()は特定のターゲットを無効化(再実行)するために使用できます
Content from targetsプロジェクト組織のベストプラクティス
最終更新日:2025-04-15 | ページの編集
概要
質問
-
targetsプロジェクトを整理するためのベストプラクティスは何ですか? -
targetsのワークフローの組織はスクリプトベースの分析とどのように異なりますか?
目的
- 最大限の再現性のために
targetsプロジェクトをどのように整理するかを説明する -
targetsの文脈で関数をどのように使用するかを理解する
ワークフロープランをより簡単に書く方法
プラン内でターゲットを指定するデフォルトの方法は、tar_target()
関数を使うことです。
しかし、この書き方は少し冗長に感じるかもしれません。
その代わりに、targetsの開発者であるWill
Landauによって作成された tarchetypes
パッケージを使う方法があります。
tarchetypes のインストール
まだインストールしていない場合は、install.packages("tarchetypes")
で tarchetypes をインストールしてください。
tarchetypes の目的は、targets
パイプラインの記述を容易にするさまざまなショートカットを提供することです。
今回はそのうちの一つ、tar_plan() を紹介します。これは
_targets.R スクリプトの最後にある list()
の代わりに使用されます。
tar_plan() を使用することで、tar_target()
を使用してターゲットを指定する代わりに、target_name = target_command
のような構文を使用できます。
ペンギンのワークフローを tar_plan()
構文を使用するように編集しましょう:
R
library(targets)
library(tarchetypes)
library(palmerpenguins)
library(tidyverse)
clean_penguin_data <- function(penguins_data_raw) {
penguins_data_raw |>
select(
species = Species,
bill_length_mm = `Culmen Length (mm)`,
bill_depth_mm = `Culmen Depth (mm)`
) |>
remove_missing(na.rm = TRUE) |>
# Split "species" apart on spaces, and only keep the first word
separate(species, into = "species", extra = "drop")
}
tar_plan(
penguins_csv_file = path_to_file("penguins_raw.csv"),
penguins_data_raw = read_csv(penguins_csv_file, show_col_types = FALSE),
penguins_data = clean_penguin_data(penguins_data_raw)
)
読みやすくなったと思いませんか?
tar_plan()
を使用するからといって、すべてのターゲットをこの方法で書かなければならないわけではありません。tar_plan()
内で tar_target()
フォーマットを使用することもできます。
これは、= が短く読みやすい一方で、targets
が提供できるすべてのカスタマイズを提供しないためです。
今のところあまり重要ではありませんが、より高度な targets
ワークフローを作成し始めると重要になります。
ファイルとフォルダの整理
これまで、すべてを単一の _targets.R
ファイルで行ってきました。
これは小規模なワークフローには問題ありませんが、ワークフローが大きくなるとあまりうまく機能しません。
コードを整理するためのより良い方法があります。
まず、_targets.R 以外の R コードを保存するために
R
というディレクトリを作成しましょう(_targets.R
はサブディレクトリではなく、プロジェクト全体のディレクトリに配置する必要があることを覚えておいてください)。
R/ 内に functions.R という新しい R
ファイルを作成します。 ここにカスタム関数を配置します。 今すぐ
clean_penguin_data() をそこに入れて保存しましょう。
同様に、library() 呼び出しを R/ 内の
packages.R
という独自のスクリプトに配置しましょう(ただし、これは唯一の方法ではありません。“パッケージの管理”
エピソード を参照してください)。
また、_targets.R スクリプトをこれらのスクリプトを
source で呼び出すように修正する必要があります:
R
source("R/packages.R")
source("R/functions.R")
tar_plan(
penguins_csv_file = path_to_file("penguins_raw.csv"),
penguins_data_raw = read_csv(penguins_csv_file, show_col_types = FALSE),
penguins_data = clean_penguin_data(penguins_data_raw)
)
これで _targets.R
はずっとスリムになりました:ワークフローに集中し、各ステップで何が起こるかをすぐに教えてくれます。
最後に、データや出力など、コードではないファイルを保存するためのディレクトリを作成しましょう。
ターゲットキャッシュ内に user
という新しいディレクトリを作成します:_targets/user。
user 内にさらに data と results
の2つのディレクトリを作成します。
(バージョン管理を使用している場合は、_targets
ディレクトリを無視することをおそらく望むでしょう)。
関数についての一言
このレッスンの前半でカスタム関数について触れましたが、これはさらに明確化が必要な重要なトピックです。
targets
のような単一のワークフローではなく、複数のスクリプトを使用して R
でデータを分析することに慣れている場合、多くの関数(function()
関数を使用)を書かないかもしれません。
これは targets との大きな違いです。
カスタム関数を使用せずに効率的な targets
パイプラインを書くのは非常に難しいでしょう。なぜなら、ビルドする各ターゲットが単一のコマンドの出力でなければならないからです。
このカリキュラムでは R での関数の書き方をカバーする時間がありませんが、このトピックを復習するためには Software Carpentry のレッスン をお勧めします。
もう一つの大きな違いは、各ターゲットが一意の名前を持たなければならない ということです。 以下のようなコードを書くことに慣れているかもしれません:
R
# 人の身長をcmで保存し、インチに変換する
height <- 160
height <- height / 2.54
同等の targets
パイプラインを実行しようとするとエラーが発生します:
R
tar_plan(
height = 160,
height = height / 2.54
)
エラー
Error:
! Error running targets::tar_make()
Error messages: targets::tar_meta(fields = error, complete_only = TRUE)
Debugging guide: https://books.ropensci.org/targets/debugging.html
How to ask for help: https://books.ropensci.org/targets/help.html
Last error message:
duplicated target names: height
Last error traceback:
base::tryCatch(base::withCallingHandlers({ NULL base::saveRDS(base::do.c...
tryCatchList(expr, classes, parentenv, handlers)
tryCatchOne(tryCatchList(expr, names[-nh], parentenv, handlers[-nh]), na...
doTryCatch(return(expr), name, parentenv, handler)
tryCatchList(expr, names[-nh], parentenv, handlers[-nh])
tryCatchOne(expr, names, parentenv, handlers[[1L]])
doTryCatch(return(expr), name, parentenv, handler)
base::withCallingHandlers({ NULL base::saveRDS(base::do.call(base::do.ca...
base::saveRDS(base::do.call(base::do.call, base::c(base::readRDS("/tmp/R...
base::do.call(base::do.call, base::c(base::readRDS("/tmp/RtmpskhYdk/call...
(function (what, args, quote = FALSE, envir = parent.frame()) { if (!is....
(function (targets_function, targets_arguments, options, envir = NULL, s...
tryCatch(out <- withCallingHandlers(targets::tar_callr_inner_try(targets...
tryCatchList(expr, classes, parentenv, handlers)
tryCatchOne(expr, names, parentenv, handlers[[1L]])
doTryCatch(return(expr), name, parentenv, handler)
withCallingHandlers(targets::tar_callr_inner_try(targets_function = targ...
targets::tar_callr_inner_try(targets_function = targets_function, target...
pipeline_from_list(targets)
pipeline_from_list.default(targets)
pipeline_init(out)
pipeline_targets_init(targets, clone_targets)
tar_assert_unique_targets(names)
tar_throw_validate(message)
tar_error(message = paste0(...), class = c("tar_condition_validate", "ta...
rlang::abort(message = message, class = class, call = tar_empty_envir)
signal_abort(cnd, .file)targets
パイプラインで作業する大部分は、適切なサイズのカスタム関数を書くことです。
それらは単一行のコードだけになるほど小さくてはいけません;そうするとパイプラインが理解しにくくなり、維持管理が難しくなります。
一方で、変更に過度に敏感になるほど大きくしてはいけません。
このバランスを取ることは科学というよりもアートであり、練習を通じてしか習得できません。私が見つけた良い経験則は、ターゲットごとに3つを超える入力を持たないことです。
まとめ
- コードを
R/フォルダに配置する - 関数を
R/functions.Rに配置する - パッケージを
R/packages.Rに指定する - その他の雑多なファイルを
_targets/userに配置する - 関数を書くことは
targetsパイプラインの重要なスキルである
Content from パッケージの管理
最終更新日:2025-04-15 | ページの編集
概要
質問
-
targetsプロジェクトのパッケージをどのように管理すべきですか?
目的
- パッケージ管理のベストプラクティスを実演する
パッケージの読み込み
ほとんどすべての R 分析は、base R で利用可能な機能を超える関数を提供するパッケージに依存しています。
targets
ワークフローでパッケージを読み込む主な方法は3つあります。
方法1: library()
これはおそらく最も馴染みのある方法であり、これまでデフォルトで使用してきた方法です。
他の R スクリプトと同様に、_targets.R
スクリプトの上部近くに library()
呼び出しを含めます。あるいは(プロジェクト組織の推奨ベストプラクティスとして)、すべての
library()
呼び出しを別のスクリプトに配置することもできます—通常これは
packages.R と呼ばれ、プロジェクトの R/
ディレクトリに保存されます。
このアプローチの潜在的な欠点は、読み込むパッケージのリストが長い場合、tar_visnetwork()、tar_outdated()
などの特定の関数がすべてのパッケージを読み込まなければならないため、必ずしもそれらを使用しなくても不必要に長い時間がかかる可能性があることです。
方法2: tar_option_set()
この方法では、ワークフローを実行するときに読み込むパッケージを指定するために、_targets.R
内で tar_option_set() 関数を使用します。
これは palmerpenguins
パッケージの事前にクリーンアップされたデータセットを使用して実演されます。例えば、アデリーペンギンのデータのみにフィルタリングしたいとしましょう。
進捗の保存
1つのプロジェクト内でアクティブな _targets.R
ファイルは1つだけです。
新しい _targets.R
ファイルを作成しようとしていますが、これまで作業してきたもの(ペンギンのくちばし分析)の進捗を失いたくないでしょう。そのファイルを一時的に
_targets_old.R
のような名前に変更することで、以下の新しい例の _targets.R
ファイルを上書きしないようにできます。再び作業を再開するときに名前を戻してください。
これが tar_option_set()
メソッドを使用したときの例です:
R
library(targets)
library(tarchetypes)
tar_option_set(packages = c("dplyr", "palmerpenguins"))
tar_plan(
adelie_data = filter(penguins, species == "Adelie")
)
出力
▶ dispatched target adelie_data
● completed target adelie_data [0.024 seconds]
▶ ended pipeline [0.183 seconds]この方法は、方法1で時々経験するかもしれない遅延を回避します。
方法3: tar_target() の packages 引数
ターゲットを定義するための主要な関数である tar_target()
には、指定したターゲットのためにのみ指定されたパッケージを読み込む
packages 引数があります。
これは、上記と同じ例から修正したこの方法の使用例です。
R
library(targets)
library(tarchetypes)
tar_plan(
tar_target(
adelie_data,
filter(penguins, species == "Adelie"),
packages = c("dplyr", "palmerpenguins")
)
)
出力
▶ dispatched target adelie_data
● completed target adelie_data [0.023 seconds]
▶ ended pipeline [0.183 seconds]これは、すべてのパッケージを読み込むよりもメモリ効率が良い場合があります。なぜなら、ワークフローの通常の実行中にすべてのターゲットが常に作成されるわけではないからです。 しかし、ターゲットごとに必要なパッケージを覚えて指定するのは手間がかかることがあります。
もう一つのオプション
実際にパッケージを読み込むことなく、::
記法を使用して各関数に関連付けられたパッケージを指定する方法があります。例えば、dplyr::mutate()
です。
これにより、パッケージの読み込みを完全に避けることができます。
この方法を使用してプランを書く方法は以下の通りです:
R
library(targets)
library(tarchetypes)
tar_plan(
adelie_data = dplyr::filter(palmerpenguins::penguins, species == "Adelie")
)
出力
▶ dispatched target adelie_data
● completed target adelie_data [0.015 seconds]
▶ ended pipeline [0.175 seconds]このアプローチの利点は、すべての関数の起源が明確になることです。例えば、GitHub でソースを見たりすることで、すぐに関数がどこから来ているかを知ることができます。 欠点は、関数を使用するたびにパッケージ名を入力する必要があるため、やや冗長になることです。
パッケージバージョンの維持
カスタム関数とパッケージの関数の追跡
targets
について理解しておくべき重要な点は、カスタム関数とターゲットのみを追跡し、パッケージによって提供される関数は追跡しない
ということです。
しかし、パッケージの内容は変更されることがあり、パッケージは通常定期的に更新されます。ワークフローの出力は、使用するパッケージだけでなく、そのバージョンにも依存する可能性があります。
したがって、パッケージバージョンを追跡することは良い考えです。
renv について
幸いなことに、これを手動で行う必要はありません。このプロセスを自動化するのに役立つ
R パッケージが利用可能です。私たちは renv
をお勧めしますが、他にも groundhog
などがあります。このレッスンでは renv
の詳細な使用方法をカバーする時間がありません。renv
を始めるには、“Introduction
to renv” ビネット を参照してください。
一般的に、renv は他の R プロジェクトと同様に
targets
プロジェクトでも使用できます。しかし、1つの例外があります:tar_option_set()
や tar_target() の packages 引数(それぞれ方法2 または方法3)を使用してパッケージを読み込む場合、renv
はそれらを検出しません(なぜなら、renv は
library()、require()
などでパッケージが読み込まれることを期待しているからです)。
この場合の解決策は、tar_renv()
関数
を使用することです。これにより、ワークフローで使用される各パッケージの
library()
呼び出しを含む別のファイルが書き出され、renv
がそれらを正しく検出できるようになります。
パッケージの関数の選択的追跡
targets
はパッケージの関数を追跡しないため、パッケージを更新してその関数の内容が変更された場合、targets
その関数によって生成されたターゲットを再構築しません。
しかし、この動作をパッケージごとに変更することは可能です。
これは、依存関係を計算する際に targets
に過度な計算負荷をかけないため、少数のパッケージに対してのみ行うのが最適です。
例えば、頻繁に更新する独自のカスタムパッケージを使用している場合にこれを行いたいかもしれません。
これを行う方法は、tar_option_set()
を使用し、packages と imports
の両方に同じパッケージ名を指定することです。以下は、dplyr
と palmerpenguins
に対してこれを示す前のコードを修正したバージョンです。
R
library(targets)
library(tarchetypes)
tar_option_set(
packages = c("dplyr", "palmerpenguins"),
imports = c("dplyr", "palmerpenguins")
)
tar_plan(
adelie_data = filter(penguins, species == "Adelie")
)
もし dplyr または palmerpenguins
を再インストールし、パイプラインで使用されるそれらの関数の一つ(例えば
filter())が変更された場合、その関数に依存するターゲットは再構築されます。
名前空間の競合の解決
パッケージに関連して言及すべき最後のベストプラクティスがあります:名前空間の競合の解決です。
「名前空間」とは、一連の一意の名前が特定のコンテキスト内でのみ一意であるという考え方を指します。 例えば、パッケージのすべての関数名は一意でなければなりませんが、そのパッケージ内でのみです。 関数名は複数のパッケージで重複する可能性があります。
ご想像の通り、これは混乱を招く可能性があります。
例えば、filter() 関数は stats パッケージと
dplyr
パッケージの両方に存在しますが、それぞれで全く異なる動作をします。
これは名前空間の競合です:私たちはどの
filter() を指しているのか、どうやって知るのでしょうか?
conflicted
パッケージは、曖昧な関数を使用しようとした場合に停止させ、どのパッケージを使用するかを明確にするのを助けることで、このような混乱を防ぐのに役立ちます。
ここでは詳細をカバーする時間がありませんが、conflicted
の使用方法については 公式サイト
を参照してください。
conflicted を使用するとき、通常は
conflicts_prefer(dplyr::filter)
のように名前空間の競合を明示的に解決する一連のコマンドを実行します(これは、dplyr
の filter を使用したいことを R
に伝えます、stats の filter
ではありません)。
これを targets
ワークフローで使用するには、.Rprofile
と呼ばれる特別なファイルに conflicts_prefer
のすべての呼び出しを配置する必要があります。このファイルはプロジェクトのメインフォルダにあります。これにより、各ターゲットに対して常に競合が解決されることが保証されます。
.Rprofile
を編集する推奨方法は、usethis::edit_r_profile("project")
を使用することです。 これにより、エディタで .Rprofile
が開かれ、そこで編集して保存できます。
例えば、あなたの .Rprofile は以下のようになります:
R
library(conflicted)
conflicts_prefer(dplyr::filter)
.Rprofile のコードを実行するために source()
を実行する必要はありません。 それは各 R
セッションの開始時に自動的に実行されます。
まとめ
-
targetsでパッケージをロードする方法は複数あります -
targetsはユーザー定義の関数のみを追跡し、パッケージは追跡しません -
renvを使用してパッケージバージョンを管理する -
conflictedパッケージを使用して名前空間の競合を管理する
Content from 外部ファイルの取り扱い
最終更新日:2025-04-15 | ページの編集
概要
質問
- 外部データをどのようにロードできますか?
目的
- ワークフローに外部データをロードできるようにする
- 外部データの内容が変更された場合にワークフローを再実行するように設定する
外部ファイルを依存関係として扱う
ほとんどすべてのワークフローはデータのインポートから始まります。データは通常、外部ファイルとして保存されています。
簡単な例として、RStudioの「新しいファイル」メニューオプションを使用して外部データファイルを作成しましょう。「Hello
World」という一行のテキストを入力し、_targets/user/data/ に
“hello.txt” テキストファイルとして保存します。
次に、このファイルの内容を読み込み、ワークフロー内で
some_data として保存するために、以下のプランを書いて
tar_make() を実行します:
進捗の保存
1つのプロジェクト内でアクティブな _targets.R
ファイルは1つだけです。
新しい _targets.R
ファイルを作成しようとしていますが、これまで作業してきたもの(ペンギンのくちばし分析)の進捗を失いたくないでしょう。そのファイルを一時的に
_targets_old.R
のような名前に変更することで、以下の新しい例の _targets.R
ファイルを上書きしないようにできます。再び作業を再開するときに名前を戻してください。
R
library(targets)
library(tarchetypes)
tar_plan(
some_data = readLines("_targets/user/data/hello.txt")
)
出力
▶ dispatched target some_data
● completed target some_data [0 seconds]
▶ ended pipeline [0.122 seconds]tar_read(some_data) を使用して some_data
の内容を検査すると、期待通り "Hello World"
という文字列が含まれていることがわかります。
次に、“hello.txt” を編集して、テキストを追加します。例えば、「Hello World. How are you?」としましょう。これをRStudioのテキストエディタで編集して保存します。次にパイプラインを再実行します。
R
library(targets)
library(tarchetypes)
tar_plan(
some_data = readLines("_targets/user/data/hello.txt")
)
出力
✔ skipped target some_data
✔ skipped pipeline [0.125 seconds]ターゲット some_data
がスキップされましたが、これはファイルの内容が変更されたにもかかわらずです。
これは、現在のところ targets
がファイルの名前のみを追跡しており、その内容を追跡していないためです。これを行うには、tarchetypes
パッケージの tar_file()
関数を使用する必要があります。tar_file()
はファイルの「ハッシュ」を計算します。これはファイルの内容によって決定される一意のデジタル署名です。内容が変更されると、ハッシュも変更され、targets
によって検出されます。
R
library(targets)
library(tarchetypes)
tar_plan(
tar_file(data_file, "_targets/user/data/hello.txt"),
some_data = readLines(data_file)
)
出力
▶ dispatched target data_file
● completed target data_file [0.001 seconds]
▶ dispatched target some_data
● completed target some_data [0 seconds]
▶ ended pipeline [0.182 seconds]今回は、targets が期待通りに some_data
を再構築するのが確認できます。
ショートカット(または、ターゲットファクトリーについて)
しかし、これにより、一つのターゲットではなく二つのターゲットを書く必要があることにも気づきます。ファイルの内容を追跡するターゲット(data_file)と、ファイルからロードした内容を保存するターゲット(some_data)です。
これは targets
ワークフローでは一般的なパターンであるため、tarchetypes
はこれをより簡潔に表現するショートカット、tar_file_read()
を提供しています。
R
library(targets)
library(tarchetypes)
tar_plan(
tar_file_read(
hello,
"_targets/user/data/hello.txt",
readLines(!!.x)
)
)
このプランを tar_manifest() で検査してみましょう:
R
tar_manifest()
出力
# A tibble: 2 × 2
name command
<chr> <chr>
1 hello_file "\"_targets/user/data/hello.txt\""
2 hello "readLines(hello_file)" tar_file_read()
を使用してパイプラインに一つのターゲット(hello)のみを指定しましたが、実際には
二つ のターゲット、hello_file と
hello が含まれていることに気づきます。
これは tar_file_read() が
ターゲットファクトリー
と呼ばれる特別な関数だからです。ターゲットファクトリーは一度に複数のターゲットを作成します。tarchetypes
パッケージの主な目的の一つは、パイプラインの記述を容易にし、エラーを減らすためにターゲットファクトリーを提供することです。
非標準評価
!!.x
の意味は何でしょうか?これはRの使用に慣れていても馴染みがないかもしれません。これは「非標準評価」として知られ、特定のコンテキストで使用されます。詳細については時間がありませんが、tar_file_read()
を使用する際にはこの特別な記法を使用する必要があることを覚えておいてください。書き方を忘れた場合(これは頻繁に起こります!)、?tar_file_read
を実行してヘルプファイルの例を参照してください。
他のデータ読み込み関数
ここでは readLines()
を例として使用しましたが、readr::read_csv()、xlsx::read_excel()
など、外部ファイルからデータを読み込む他の関数でも同じパターンを使用できます(例えば、read_csv(!!.x)、read_excel(!!.x)
など)。
これは一般的に推奨されます。そうすることで、入力データとパイプラインが同期し、常に最新の状態に保たれます。
Challenge: penguins の例で
tar_file_read() を使用する
ペンギンのくちばし分析を開始したとき、まだ
tar_file_read() を知りませんでした。
tar_file_read()
を使用してCSVファイルを読み込み、その内容を追跡するにはどうすればよいですか?
R
source("R/packages.R")
source("R/functions.R")
tar_plan(
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
penguins_data = clean_penguin_data(penguins_data_raw)
)
出力
▶ dispatched target penguins_data_raw_file
● completed target penguins_data_raw_file [0.001 seconds]
▶ dispatched target penguins_data_raw
● completed target penguins_data_raw [0.096 seconds]
▶ dispatched target penguins_data
● completed target penguins_data [0.014 seconds]
▶ ended pipeline [0.25 seconds]データの書き出し
ファイルへの書き出しは、ファイルの読み込みと似ています。tar_file()
関数を使用します。ただし、重要な注意点があります:この場合、tar_file()
の第二引数(ターゲットをビルドするためのコマンド)はファイルへのパスを返さなければなりません。すべてのファイルを書き出す関数がこれを行うわけではありません(一部は何も返さず、ファイルの出力を関数の副作用として扱います)。そのため、ファイルを書き出し、そのパスを返すカスタム関数を定義する必要があるかもしれません。
R
x <- writeLines("some text", "test.txt")
x
出力
NULLここでは、文字データをファイルに書き出し、そのファイル名を返す修正済み関数を作成します(...
は「これらの引数の残りを writeLines()
に渡す」を意味します):
R
write_lines_file <- function(text, file, ...) {
writeLines(text = text, con = file, ...)
file
}
これを試してみましょう:
R
x <- write_lines_file("some text", "test.txt")
x
出力
[1] "test.txt"これで、この関数をパイプラインで使用できます。例えば、テキストを大文字に変換して再度書き出してみましょう:
R
library(targets)
library(tarchetypes)
source("R/functions.R")
tar_plan(
tar_file_read(
hello,
"_targets/user/data/hello.txt",
readLines(!!.x)
),
hello_caps = toupper(hello),
tar_file(
hello_caps_out,
write_lines_file(hello_caps, "_targets/user/results/hello_caps.txt")
)
)
出力
▶ dispatched target hello_file
● completed target hello_file [0.001 seconds]
▶ dispatched target hello
● completed target hello [0 seconds]
▶ dispatched target hello_caps
● completed target hello_caps [0 seconds]
▶ dispatched target hello_caps_out
● completed target hello_caps_out [0 seconds]
▶ ended pipeline [0.188 seconds]results フォルダ内の hello_caps.txt
を見て、期待通りであることを確認してください。
Challenge: What happens to file output if its modified?
Delete or change the contents of hello_caps.txt in the
results folder. What do you think will happen when you run
tar_make() again? Try it and see.
targets は hello_caps_out
が変更された(「無効化された」)ことを検出し、再構築のためにコードを再実行します。これにより、hello_caps.txt
が再度 results に書き出されます。
この方法で結果を出力することで、パイプラインがより堅牢になります。つまり、results
内のファイルの内容がプラン内のコードによってのみ生成されることが保証されます。
まとめ
-
tarchetypes::tar_file()はファイルの内容を追跡します -
tarchetypes::tar_file_read()をデータ読み込み関数(例えばread_csv())と組み合わせて使用し、入力データとパイプラインを同期させる -
tarchetypes::tar_file()をファイルに書き出す関数(ファイルに書き込んでパスを返す関数)と組み合わせてデータを書き出す
Content from ブランチング
最終更新日:2025-04-15 | ページの編集
概要
質問
- すべてを入力せずに多くのターゲットをどのように指定できますか?
目的
- ブランチングを使用してターゲットを指定できるようにする
なぜブランチングなのか?
targets
の大きな強みの一つは、ブランチングと呼ばれる、単一のコード行から多くのターゲットを定義できる能力です。
これは入力を省くだけでなく、タイプミスの可能性が減るため、エラーのリスクも低減します。
ブランチングの種類
ブランチングには、動的ブランチングと静的ブランチングの二種類があります。
「ブランチング」とは、ターゲットを作成する方法(「パターン」)を単一に指定し、targets
がそれから複数のターゲット(「ブランチ」)を生成するという考え方を指します。
「動的」とは、パターンから生成されるブランチが事前に定義されている必要がなく、コードの結果として動的に生成されることを意味します。
このワークショップでは、動的ブランチングのみを扱います。静的ブランチングはメタプログラミングの使用を必要とするため、これは高度なトピックです。どちらをいつ使用するか(または両方の組み合わせ)についての詳細は、targets
パッケージマニュアルを参照してください。
ブランチングなしの例
これがどのように機能するかを理解するために、palmerpenguins
データセットの分析を続けましょう。
私たちの仮説は、くちばしの深さがくちばしの長さとともに減少するということです。 この仮説を線形モデルで検証します。
例えば、これはくちばしの長さに依存するくちばしの深さのモデルです:
R
lm(bill_depth_mm ~ bill_length_mm, data = penguins_data)
これをパイプラインに追加できます。すべての種を区別せずに結合しているため、combined_model
と呼びます:
R
source("R/packages.R")
source("R/functions.R")
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build model
combined_model = lm(
bill_depth_mm ~ bill_length_mm,
data = penguins_data
)
)
出力
✔ skipped target penguins_data_raw_file
✔ skipped target penguins_data_raw
✔ skipped target penguins_data
▶ dispatched target combined_model
● completed target combined_model [0.023 seconds]
▶ ended pipeline [0.171 seconds]モデルを見てみましょう。broom パッケージの
glance() 関数を使用します。これは base R の
summary() とは異なり、出力をティブル(データフレームの
tidyverse
相当)として返します。後で見るように、これは下流の分析に非常に便利です。
R
library(broom)
tar_load(combined_model)
glance(combined_model)
出力
# A tibble: 1 × 12
r.squared adj.r.squared sigma statistic p.value df logLik AIC BIC deviance df.residual nobs
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int>
1 0.0552 0.0525 1.92 19.9 0.0000112 1 -708. 1422. 1433. 1256. 340 342小さな P-値に注目してください。 これはモデルが非常に有意であることを示しているようです。
しかし、ちょっと待ってください… これは本当に適切なモデルでしょうか? データセットには3種類のペンギンがいることを思い出してください。くちばしの深さと長さの関係が種によって異なる可能性があります。
おそらく、種に対するパラメータを追加するモデルや、種とくちばしの長さの相互作用効果を追加するモデルなど、いくつかの代替モデルをテストする必要があります。
これでワークフローがより複雑になっています。これはブランチングなしでのそのような分析のワークフローの例です(packages.R
に library(broom)
を追加することを忘れないでください):
R
source("R/packages.R")
source("R/functions.R")
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build models
combined_model = lm(
bill_depth_mm ~ bill_length_mm,
data = penguins_data
),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species,
data = penguins_data
),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species,
data = penguins_data
),
# Get model summaries
combined_summary = glance(combined_model),
species_summary = glance(species_model),
interaction_summary = glance(interaction_model)
)
出力
✔ skipped target penguins_data_raw_file
✔ skipped target penguins_data_raw
✔ skipped target penguins_data
✔ skipped target combined_model
▶ dispatched target interaction_model
● completed target interaction_model [0.004 seconds]
▶ dispatched target species_model
● completed target species_model [0.001 seconds]
▶ dispatched target combined_summary
● completed target combined_summary [0.009 seconds]
▶ dispatched target interaction_summary
● completed target interaction_summary [0.002 seconds]
▶ dispatched target species_summary
● completed target species_summary [0.003 seconds]
▶ ended pipeline [0.188 seconds]モデルの一つのサマリーを見てみましょう:
R
tar_read(species_summary)
出力
# A tibble: 1 × 12
r.squared adj.r.squared sigma statistic p.value df logLik AIC BIC deviance df.residual nobs
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int>
1 0.769 0.767 0.953 375. 3.65e-107 3 -467. 944. 963. 307. 338 342この方法でパイプラインを書くと機能しますが、繰り返しが多くなります。各モデルのサマリー統計量を取得するたびに
glance() を呼び出さなければなりません。
さらに、各サマリータゲット(combined_summary
など)は明示的に名前が付けられ、手動で入力されています。
タイプミスをして間違ったモデルがサマリーされるのはかなり簡単です。
ブランチングを使用した例
最初の試み
動的ブランチングを使用して同じプランを書く方法を見てみましょう:
R
source("R/packages.R")
source("R/functions.R")
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build models
models = list(
combined_model = lm(
bill_depth_mm ~ bill_length_mm, data = penguins_data),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species, data = penguins_data),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species, data = penguins_data)
),
# Get model summaries
tar_target(
model_summaries,
glance(models[[1]]),
pattern = map(models)
)
)
ここで何が起こっているのでしょうか?
まず、tar_make()
が提供するメッセージを見てみましょう。
出力
✔ skipped target penguins_data_raw_file
✔ skipped target penguins_data_raw
✔ skipped target penguins_data
▶ dispatched target models
● completed target models [0.004 seconds]
▶ dispatched branch model_summaries_812e3af782bee03f
● completed branch model_summaries_812e3af782bee03f [0.008 seconds]
▶ dispatched branch model_summaries_2b8108839427c135
● completed branch model_summaries_2b8108839427c135 [0.003 seconds]
▶ dispatched branch model_summaries_533cd9a636c3e05b
● completed branch model_summaries_533cd9a636c3e05b [0.002 seconds]
● completed pattern model_summaries
▶ ended pipeline [0.198 seconds]一連の小さなターゲット(ブランチ)があり、それぞれが
model_summaries_812e3af782bee03f のように名前付けされ、その後に全体の
model_summaries ターゲットがあります。
これはブランチングを使用してターゲットを指定した結果です:小さなターゲットそれぞれが全体のターゲットを構成する「ブランチ」です。
targets
は、事前にどれだけのブランチが存在するか、またそれらが何を表しているかを知らないため、数字と文字の一連(「ハッシュ」)を使用して各ブランチに名前を付けます。
targets
は各ブランチを一つずつビルドし、それらを全体のターゲットに結合します。
次に、ワークフローがどのように設定されているか、モデルの定義から詳しく見てみましょう:
R
# Build models
models = list(
combined_model = lm(
bill_depth_mm ~ bill_length_mm, data = penguins_data),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species, data = penguins_data),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species, data = penguins_data)
),ブランチングなしのバージョンとは異なり、モデルをリスト内で定義しました(モデルごとに一つのターゲットではなく)。
これは動的ブランチングが base::apply() や purrrr::map()
のループ方法に似ているためです:リストの各要素に関数を適用します。
したがって、ループの入力としてリストを準備する必要があります。
次に、ターゲット model_summaries
をビルドするコマンドを見てみましょう。
R
# Get model summaries
tar_target(
model_summaries,
glance(models[[1]]),
pattern = map(models)
)
以前と同様に、最初の引数はビルドするターゲットの名前で、二つ目の引数はそれをビルドするコマンドです。
ここでは、glance() 関数を models
の各要素に適用しています([[1]]
が必要なのは、関数が適用されるとき、各要素が実際にはネストされたリストであり、ネストを一層取り除く必要があるためです)。
最後に、これまで見たことのない引数 pattern
があります。これは、このターゲットが動的ブランチングを使用してビルドされるべきことを示します。
map
は、入力リスト(models)の各要素に対して関数を順次適用することを意味します。
ブランチングワークフローの構築方法を理解したので、出力を検査してみましょう:
R
tar_read(model_summaries)
出力
# A tibble: 3 × 12
r.squared adj.r.squared sigma statistic p.value df logLik AIC BIC deviance df.residual nobs
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int>
1 0.0552 0.0525 1.92 19.9 1.12e- 5 1 -708. 1422. 1433. 1256. 340 342
2 0.769 0.767 0.953 375. 3.65e-107 3 -467. 944. 963. 307. 338 342
3 0.770 0.766 0.955 225. 8.52e-105 5 -466. 947. 974. 306. 336 342モデルのサマリー統計量がすべて一つのデータフレームに含まれています。
しかし、一つ問題があります:どの行がどのモデルから来たのか分かりません! モデルのリストと同じ順序であると仮定するのは賢明ではありません。
これは動的ブランチングの動作方法によるものです:デフォルトでは、各ターゲットの由来に関する情報が出力に保持されません。
これをどう修正すれば良いでしょうか?
第二の試み
ブランチングパイプラインから有用な出力を得るための鍵は、各ブランチの出力に必要な情報を含めることです。
ここでは、モデルサマリーの各行に対応するモデルの種類を知りたいと考えています。
これを行うために、カスタム関数を書く必要があります。
targets
を使用する際にはカスタム関数を頻繁に書く必要があるため、慣れておくと良いでしょう!
以下がその関数です。これを R/functions.R
に保存してください:
R
glance_with_mod_name <- function(model_in_list) {
model_name <- names(model_in_list)
model <- model_in_list[[1]]
glance(model) |>
mutate(model_name = model_name)
}
新しいパイプラインは以前とほぼ同じですが、今回は
glance() の代わりにカスタム関数を使用しています。
R
source("R/functions.R")
source("R/packages.R")
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build models
models = list(
combined_model = lm(
bill_depth_mm ~ bill_length_mm, data = penguins_data),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species, data = penguins_data),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species, data = penguins_data)
),
# Get model summaries
tar_target(
model_summaries,
glance_with_mod_name(models),
pattern = map(models)
)
)
出力
✔ skipped target penguins_data_raw_file
✔ skipped target penguins_data_raw
✔ skipped target penguins_data
✔ skipped target models
▶ dispatched branch model_summaries_812e3af782bee03f
● completed branch model_summaries_812e3af782bee03f [0.013 seconds]
▶ dispatched branch model_summaries_2b8108839427c135
● completed branch model_summaries_2b8108839427c135 [0.006 seconds]
▶ dispatched branch model_summaries_533cd9a636c3e05b
● completed branch model_summaries_533cd9a636c3e05b [0.003 seconds]
● completed pattern model_summaries
▶ ended pipeline [0.198 seconds]今回は、model_summaries
をロードすると、各行がどのモデルに対応しているかを知ることができます(右にスクロールする必要があるかもしれません)。
R
tar_read(model_summaries)
出力
# A tibble: 3 × 13
r.squared adj.r.squared sigma statistic p.value df logLik AIC BIC deviance df.residual nobs model_name
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int> <int> <chr>
1 0.0552 0.0525 1.92 19.9 1.12e- 5 1 -708. 1422. 1433. 1256. 340 342 combined_model
2 0.769 0.767 0.953 375. 3.65e-107 3 -467. 944. 963. 307. 338 342 species_model
3 0.770 0.766 0.955 225. 8.52e-105 5 -466. 947. 974. 306. 336 342 interaction_model次に、モデルに基づくくちばしの深さの予測を追加します。これはレポートでモデルをプロットする際に必要になります。
この予測は broom パッケージの augment()
関数を使用して取得できます。
R
tar_load(models)
augment(models[[1]])
出力
# A tibble: 342 × 8
bill_depth_mm bill_length_mm .fitted .resid .hat .sigma .cooksd .std.resid
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 18.7 39.1 17.6 1.14 0.00521 1.92 0.000924 0.594
2 17.4 39.5 17.5 -0.127 0.00485 1.93 0.0000107 -0.0663
3 18 40.3 17.5 0.541 0.00421 1.92 0.000168 0.282
4 19.3 36.7 17.8 1.53 0.00806 1.92 0.00261 0.802
5 20.6 39.3 17.5 3.06 0.00503 1.92 0.00641 1.59
6 17.8 38.9 17.6 0.222 0.00541 1.93 0.0000364 0.116
7 19.6 39.2 17.6 2.05 0.00512 1.92 0.00293 1.07
8 18.1 34.1 18.0 0.114 0.0124 1.93 0.0000223 0.0595
9 20.2 42 17.3 2.89 0.00329 1.92 0.00373 1.50
10 17.1 37.8 17.7 -0.572 0.00661 1.92 0.000296 -0.298
# ℹ 332 more rowsチャレンジ: ワークフローにモデル予測を追加する
augment() を使用してモデル予測を追加できますか?
glance() と同様にカスタム関数を定義する必要があります。
新しい関数を augment_with_mod_name()
として定義します。これは glance_with_mod_name()
と同じですが、glance() の代わりに augment()
を使用します:
R
augment_with_mod_name <- function(model_in_list) {
model_name <- names(model_in_list)
model <- model_in_list[[1]]
augment(model) |>
mutate(model_name = model_name)
}
ワークフローにステップを追加します:
R
source("R/functions.R")
source("R/packages.R")
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build models
models = list(
combined_model = lm(
bill_depth_mm ~ bill_length_mm, data = penguins_data),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species, data = penguins_data),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species, data = penguins_data)
),
# Get model summaries
tar_target(
model_summaries,
glance_with_mod_name(models),
pattern = map(models)
),
# Get model predictions
tar_target(
model_predictions,
augment_with_mod_name(models),
pattern = map(models)
)
)
ブランチングのベストプラクティス
動的ブランチングはデータフレーム(ティブル)と相性が良いように設計されています。
可能であれば、カスタム関数をデータフレームを入力として受け取り、データフレームを出力として返すように書き、必要なメタデータを列として常に含めるようにしてください。
チャレンジ: 他にどんな種類のパターンがありますか?
これまで、pattern 引数と組み合わせて map()
を使用し、入力の各要素に対して関数を順次適用する単一の関数のみを使用しました。
ブランチングパターンを適用する他の方法を考えてみてください。
ブランチングパターンを適用する他の方法には以下のようなものがあります:
- crossing:
要素の組み合わせごとに一つのブランチを作成する(
cross()関数) - slicing:
手動で選択した要素ごとに一つのブランチを作成する(
slice()関数) - sampling:
ランダムに選択した要素ごとに一つのブランチを作成する(
sample()関数)
ブランチングパターンの詳細については targets
マニュアル を参照してください。
まとめ
動的ブランチングは単一のコマンドで複数のターゲットを作成します
ブランチの出力に必要なメタデータを含めるために、通常カスタム関数を書く必要があります
Content from 並列処理
最終更新日:2025-04-15 | ページの編集
概要
質問
-
targetsのターゲットを並列でビルドするにはどうすればよいですか?
目的
- ターゲットを並列でビルドできるようにする
パイプラインに多くのターゲットが含まれ始めたら、並列処理を考えるかもしれません。 これはコンピュータの複数のプロセッサを活用して、同時に複数のターゲットをビルドします。
並列処理を使用するタイミング
並列処理は、ワークフローに独立したタスクがある場合にのみ使用すべきです—ワークフローがターゲットの線形シーケンスのみで構成されている場合、並列化するものはありません。 ブランチングを使用するほとんどのワークフローは並列処理の恩恵を受けることができます。
targets
は高性能コンピューティング、クラウドコンピューティング、およびさまざまな並列バックエンドをサポートしています。
ここでは、この分析をラップトップで実行していると仮定し、比較的シンプルなバックエンドを使用します。
高性能コンピューティングに興味がある場合は、targets
マニュアル を参照してください。
ワークフローのセットアップ
crew
を使用して並列処理を有効にするには、crew
パッケージをロードし、tar_option_set を使用して
targets にそれを使用するように指示するだけです。
具体的には、以下の行が crew
を有効にし、2つの並列ワーカーを使用するように指示します。
より強力なマシンでは、この数を増やすことができます:
R
library(crew)
tar_option_set(
controller = crew_controller_local(workers = 2)
)
ペンギンの分析にこれらの変更を加えましょう。 現在は次のようになっているはずです:
R
source("R/functions.R")
source("R/packages.R")
# Set up parallelization
library(crew)
tar_option_set(
controller = crew_controller_local(workers = 2)
)
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build models
models = list(
combined_model = lm(
bill_depth_mm ~ bill_length_mm, data = penguins_data),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species, data = penguins_data),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species, data = penguins_data)
),
# Get model summaries
tar_target(
model_summaries,
glance_with_mod_name(models),
pattern = map(models)
),
# Get model predictions
tar_target(
model_predictions,
augment_with_mod_name(models),
pattern = map(models)
)
)
このデモの目的のためにまだ1つだけ変更する必要があります:今、分析を並列で実行しても、関数が非常に高速であるため、計算時間の違いに気付かないでしょう。
そこで、Sys.sleep()
関数を使用して、glance_with_mod_name() と
augment_with_mod_name()
の「遅い」バージョンを作成しましょう。これはコンピュータに数秒待つよう指示します。
これにより、長時間実行される計算をシミュレートし、順次実行と並列実行の違いを確認できます。
これらの関数を functions.R
に追加します(元のものをコピー&ペーストしてから修正しても構いません):
R
glance_with_mod_name_slow <- function(model_in_list) {
Sys.sleep(4)
model_name <- names(model_in_list)
model <- model_in_list[[1]]
broom::glance(model) |>
mutate(model_name = model_name)
}
augment_with_mod_name_slow <- function(model_in_list) {
Sys.sleep(4)
model_name <- names(model_in_list)
model <- model_in_list[[1]]
broom::augment(model) |>
mutate(model_name = model_name)
}
次に、プランを「遅い」バージョンの関数を使用するように変更します:
R
source("R/functions.R")
source("R/packages.R")
# Set up parallelization
library(crew)
tar_option_set(
controller = crew_controller_local(workers = 2)
)
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build models
models = list(
combined_model = lm(
bill_depth_mm ~ bill_length_mm, data = penguins_data),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species, data = penguins_data),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species, data = penguins_data)
),
# Get model summaries
tar_target(
model_summaries,
glance_with_mod_name_slow(models),
pattern = map(models)
),
# Get model predictions
tar_target(
model_predictions,
augment_with_mod_name_slow(models),
pattern = map(models)
)
)
最後に、通常どおり tar_make()
を使用してパイプラインを実行します。
出力
✔ skip target penguins_data_raw_file
✔ skip target penguins_data_raw
✔ skip target penguins_data
✔ skip target models
• start branch model_predictions_5ad4cec5
• start branch model_predictions_c73912d5
• start branch model_predictions_91696941
• start branch model_summaries_5ad4cec5
• start branch model_summaries_c73912d5
• start branch model_summaries_91696941
• built branch model_predictions_5ad4cec5 [4.884 seconds]
• built branch model_predictions_c73912d5 [4.896 seconds]
• built branch model_predictions_91696941 [4.006 seconds]
• built pattern model_predictions
• built branch model_summaries_5ad4cec5 [4.011 seconds]
• built branch model_summaries_c73912d5 [4.011 seconds]
• built branch model_summaries_91696941 [4.011 seconds]
• built pattern model_summaries
• end pipeline [15.153 seconds]各個別ターゲットをビルドするのに約4秒かかるにもかかわらず、ワークフロー全体を実行するのにかかる総時間は、個々のターゲットの合計時間よりも短いことに注目してください! これはプロセスが並列で実行されており、時間を節約している ことの証明です。
targets
の独自で強力な点は、並列で実行するためにカスタム関数を変更する必要がなかった
ことです。ワークフローを調整しただけです。これは、ワークフローを順次にローカルで実行するか、高性能なコンテキストで並列に実行するようにリファクタリング(修正)するのが比較的簡単であることを意味します。
これがどのように機能するかを実演したので、分析プランを作成した関数の元のバージョンに戻すことができます。
まとめ
- 動的ブランチングは単一のコマンドで複数のターゲットを作成します
- ブランチの出力に必要なメタデータを含めるために、通常カスタム関数を書く必要があります
- 並列コンピューティングは関数ではなく、ワークフローのレベルで機能します
Content from Quartoを用いた再現可能なレポート
最終更新日:2025-04-15 | ページの編集
概要
質問
- 再現可能なレポートをどのように作成できますか?
目的
-
targetsを使用してレポートを生成できるようになる
コピー&ペースト vs. 動的ドキュメント
通常、データ分析の結果をより広いオーディエンスに伝えたいと考えるでしょう。
以前は、統計、プロット、その他の結果をテキストドキュメントやプレゼンテーションにコピー&ペーストすることでこれを行っていたかもしれません。 これは、分析を一度だけ行う場合には問題ありません。 しかし、それはほとんどの場合ではありません。分析の一部を調整したり、新しいデータを追加してパイプラインを再実行する可能性がはるかに高いです。 コピー&ペーストの方法では、どの結果が変更されたかを覚えておき、手動ですべてが最新であることを確認しなければなりません。 これは危険な作業です!
幸いにも、targets
はドキュメントをパイプラインの結果と同期させるための関数を提供しており、このような落とし穴を避けることができます。
ドキュメントを生成するために使用する主なツールは Quarto
です。 Quarto は targets
とは別に使用することもできます(これは独自に大きなトピックですが)、targets
と組み合わせて動的にレポートを生成する優れた方法でもあります。
Quarto を使用すると、Rコードの結果をドキュメントに直接挿入できるため、コピー&ペーストのミスの危険がありません。 さらに、PDF、HTML、Microsoft Word など、同じ基礎となるスクリプトから複数の形式で出力を生成することができます。
Quartoのインストール
v2022.07.1以降、RStudioにはQuartoが含まれていますので、別途インストールする必要はありません。RStudioからQuartoを実行できない場合は、最新バージョンのRStudioをインストールすることをお勧めします。
Quartoファイルについて
.qmd または .Qmd
はQuartoファイルの拡張子で、「Quarto markdown」の略です。
Quartoファイルは、コードとコメントの通常の書き方を逆転させています。典型的なRスクリプトでは、すべてのテキストはコメントでない限りRコードと見なされますが、Quartoではすべてのテキストが散文と見なされ、評価されるRコードの行を示すために特別な表記を使用します。
コードが評価されると、その結果が最終的なレンダリングされたドキュメントに挿入され、さまざまな形式の一つになる可能性があります。
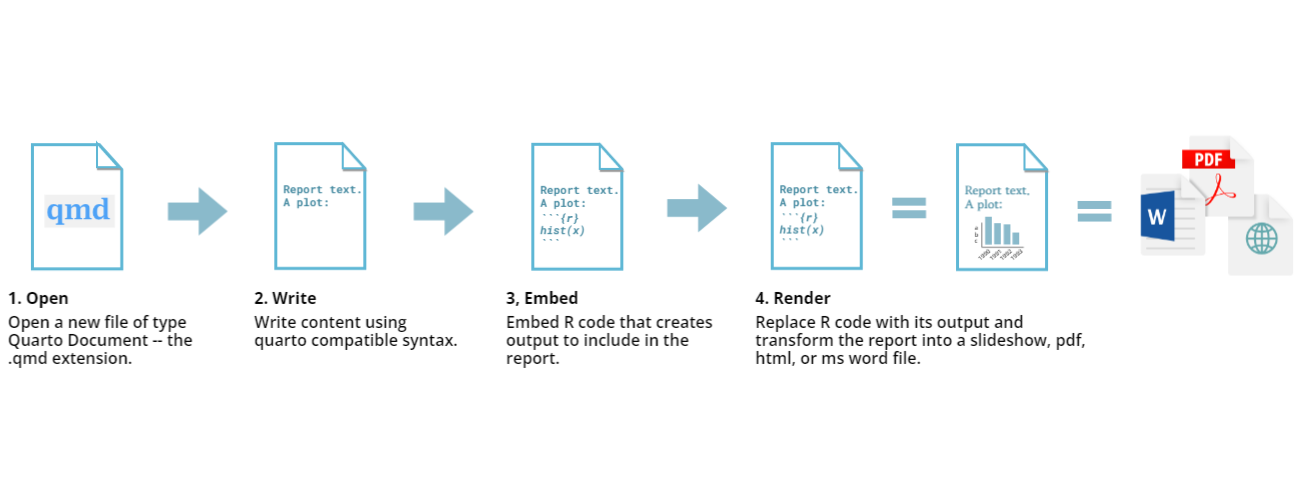
このレッスンではQuartoの詳細に踏み込む時間がありませんが、このトピックについては “RStudioによる再現可能な出版物のイントロダクション” インキュベーター(開発中)のレッスン をお勧めします。
推奨ワークフロー
Quartoのような動的ドキュメント(またはQuartoの前身であるRmarkdown)は、実際にはデータ分析パイプラインの管理に使用できます。
しかし、これはスケーラビリティが低く、targets
が提供する高度な依存関係追跡が欠けているため、推奨されません。
私たちの推奨アプローチは、データ分析の大部分(つまり「重い作業」)を
targets
パイプラインで実行し、Quartoドキュメントを使用して結果を
要約 し、 プロット することです。
ペンギンのくちばしの大きさに関するレポート
ペンギンのくちばしの大きさの分析を続けて、各モデルを評価するレポートを書きましょう。
時間を節約するために、レポートはすでに https://github.com/joelnitta/penguins-targets にあります。
こちらから生のコードをコピーして、新しいファイル
penguin_report.qmd
としてプロジェクトフォルダに保存してください(ブラウザで右クリックして「名前を付けて保存」を選択することもできます)。
次に、tar_quarto()
関数を使用してパイプラインにもう一つターゲットを追加します。以下のようにします:
R
source("R/functions.R")
source("R/packages.R")
tar_plan(
# Load raw data
tar_file_read(
penguins_data_raw,
path_to_file("penguins_raw.csv"),
read_csv(!!.x, show_col_types = FALSE)
),
# Clean data
penguins_data = clean_penguin_data(penguins_data_raw),
# Build models
models = list(
combined_model = lm(
bill_depth_mm ~ bill_length_mm, data = penguins_data),
species_model = lm(
bill_depth_mm ~ bill_length_mm + species, data = penguins_data),
interaction_model = lm(
bill_depth_mm ~ bill_length_mm * species, data = penguins_data)
),
# Get model summaries
tar_target(
model_summaries,
glance_with_mod_name(models),
pattern = map(models)
),
# Get model predictions
tar_target(
model_predictions,
augment_with_mod_name(models),
pattern = map(models)
),
# Generate report
tar_quarto(
penguin_report,
path = "penguin_report.qmd",
quiet = FALSE,
packages = c("targets", "tidyverse")
)
)
レポートを生成する関数は tar_quarto()
で、tarchetypes パッケージから提供されています。
ご覧のとおり、モデルの実行という「重い」分析はワークフロー内で行われ、その後
tar_quarto()
を使用してレポートをレンダリングする一つの呼び出しが最後にあります。
targets
はいつレポートをレンダリングするかをどのように知るのか?
このままでは、targets がワークフローの
最後
にレポートを生成することをどのように知っているのかがすぐには明らかではありません(ビルド順序はターゲットがワークフロー内で書かれた順序ではなく、それらの依存関係によって決まることを思い出してください)。
penguin_report
は他のターゲットに依存していないように見えます。なぜなら、それらは
tar_quarto() の呼び出しに表示されていないからです。
これはどのように機能するのでしょうか?
答えは penguin_report.qmd ファイルの
内部
にあります。ファイルの開始部分を見てみましょう:
MARKDOWN
---
title: "Simpson's Paradox in Palmer Penguins"
format:
html:
toc: true
execute:
echo: false
---
```{r}
#| label: load
#| message: false
targets::tar_load(penguin_models_augmented)
targets::tar_load(penguin_models_summary)
library(tidyverse)
```
これは南極のパルマー諸島におけるペンギンの分析の一例です。最初の --- と ---
の間の行は「YAMLヘッダー」と呼ばれ、ドキュメントをレンダリングする方法に関する指示が含まれています。
実行されるRコードは ```{r} と ```
の間の行で指定されます。これは「コードチャンク」と呼ばれ、散文テキストに挿入されたコードの一部です。
Rコードチャンクを詳しく見てみましょう。
targets::tar_load()
の2つの呼び出しに注目してください。この関数が何をするか覚えていますか?これはワークフロー中にビルドされたターゲットをロードします。
これで少し理解が深まったはずです。targets
はレポートがワークフロー中にビルドされたターゲット、penguin_models_augmented
および penguin_models_summary に依存していることを
レポート内で tar_load()
を使用してロードしているため 知っています。
動的コンテンツの生成
penguin_report.qmd の開始部分での
tar_load()
の呼び出しは、最新のレポートを生成するための鍵です。これらがワークフローからロードされると、データと同期していることがわかり、それらを使用して「洗練された」テキストやプロットを生成することができます。
チャレンジ: 動的コンテンツを見つけよう
penguin_report.qmd
を読み、ワークフロー中にビルドされたターゲット
(penguin_models_augmented および
penguin_models_summary)
がテキストやプロットを動的に生成するために使用されている箇所を見つけてみてください。
results-statsとラベル付けされたコードチャンクでは、P-値や調整済み R 二乗値などのモデルからの統計が抽出され、`r mod_stats$combined$r.squared`のようなインラインコードを使用してテキストに挿入されています。2つの図があります。一つは結合モデル用、もう一つは別々のモデル用です(それぞれ
fig-combined-plotとfig-separate-plotとラベル付けされたコードチャンク)。これらはpenguin_models_augmentedのモデルから予測されたポイントを使用して構築されています。
penguin_report.qmd
のコードを対話的に実行して、tar_load()
から何が起こっているのかをよりよく理解するべきです。実際、このレポートはそのように書かれました。コードが対話セッションで実行され、望ましい結果を得るために徐々に調整されながらレポートに保存されました。
このレポート生成アプローチを学ぶ最良の方法は、自分で試すこと です。
したがって、最終的なチャレンジは、自分のデータを使用して
targets
ワークフローを構築し、レポートを生成することです。頑張ってください!
まとめ
-
tarchetypes::tar_quarto()は Quarto ドキュメントをレンダリングするために使用されます - Quarto ドキュメント内で
tar_load()およびtar_read()を使用してターゲットをロードする必要があります - 重い計算は主な
targetsワークフローで行い、軽いフォーマットやプロットの生成は Quarto ドキュメントで行うことが推奨されます
